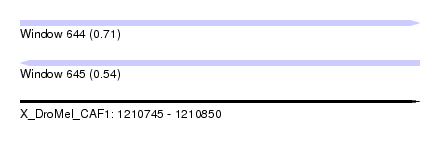
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 1,210,745 – 1,210,850 |
| Length | 105 |
| Max. P | 0.709735 |

| Location | 1,210,745 – 1,210,850 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 77.53 |
| Mean single sequence MFE | -32.72 |
| Consensus MFE | -15.29 |
| Energy contribution | -17.53 |
| Covariance contribution | 2.24 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.47 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.709735 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
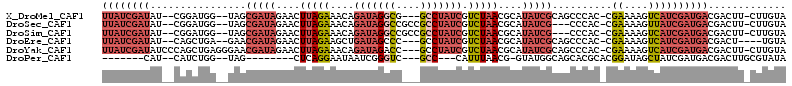
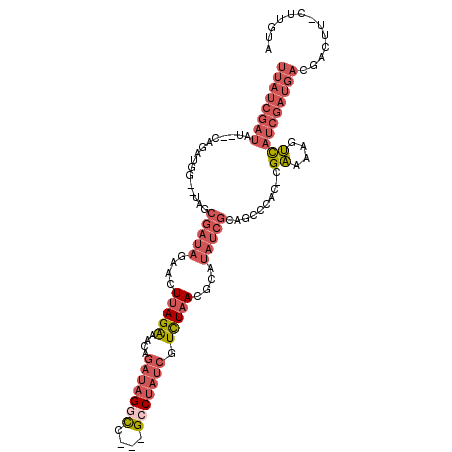
>X_DroMel_CAF1 1210745 105 + 22224390 UACAAG-AAGUCGUCAUCGAUGACUUUUCG-GUGGGCUGCGAUAUGCGUUAGACGAUAGGC---CGCCUAUCUGUUUCUAAGUUCUAUCGCUA--CCAUCCG--AUAUCGAUAA .....(-((((((((...)))))))))(((-((((...((((((.((.(((((.((((((.---..))))))....)))))))..))))))..--))).)))--)......... ( -35.40) >DroSec_CAF1 3110 105 + 1 UACAAG-AAGUCGUCAUCGAUAACUUUUCG-GUGGG---CGAUAUGCGUUAGACGAUAGGCGGCGGCCUAUCUGUUUCUAAGUUCUAUCGCUA--CCAUCCG--AUAUCGAUAA ......-........(((((((.....(((-(((((---(((((.((.(((((.(((((((....)))))))....)))))))..))))))..--))).)))--)))))))).. ( -35.80) >DroSim_CAF1 3558 105 + 1 UACAAG-AAGUCGUCAUCGAUGACUUUUCG-GUGGG---CGAUAUGCGUUAGACGAUAGGCGGCGGCCUAUCUGUUUCUAAGUUCUAUCGCUA--CCAUCCG--AUAUCGAUAA .....(-((((((((...)))))))))(((-(((((---(((((.((.(((((.(((((((....)))))))....)))))))..))))))..--))).)))--)......... ( -38.20) >DroEre_CAF1 3067 102 + 1 UACA----AGUCGUCAUCGAUGACUUUUCG-GUGGGCUGCGAUAUGCGUUAGACGAUAGGC---GGGCUAUCAGCUUCUAAGUUCUAUCGUUC--UCAGCUG--AUAUCGAUAA ...(----(((((((...)))))))).(((-((((((((.((...(((.((((.(((((..---...))))).(((....))))))).)))))--.))))).--.))))))... ( -29.30) >DroYak_CAF1 3100 109 + 1 UACAAG-AAGUCGUCAUCGAUGACUUUUCG-GUGGGCUGCGAUAUGCGUUAGACGAUAGGC---GGUCUAUCUGUUUCUAAGUUCUAUCGUUCCCUCAGCUGGGAUAUCGAUAA ....((-((((((((...)))))))))).(-(((((((((..(((.((.....))))).))---)))))))).............(((((.((((......))))...))))). ( -36.60) >DroPer_CAF1 4844 88 + 1 UAUACGCAAGUCGUCAUCGAUAGCUAUCCGUGCGUGCUGCCAUAC-CGUUAAAUG---GGC---GACCCGAUUAUUCCUGAG--------CUA--CCAGAUG--AUG------- ...((....))(((((((..(((((...(((.(((...((.....-.))...)))---.))---)....((....))...))--------)))--...))))--)))------- ( -21.00) >consensus UACAAG_AAGUCGUCAUCGAUGACUUUUCG_GUGGGCUGCGAUAUGCGUUAGACGAUAGGC___GGCCUAUCUGUUUCUAAGUUCUAUCGCUA__CCAGCCG__AUAUCGAUAA ........(((((((...))))))).......(((...((((((.((.(((((.(((((((....)))))))....)))))))..))))))....)))................ (-15.29 = -17.53 + 2.24)


| Location | 1,210,745 – 1,210,850 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 77.53 |
| Mean single sequence MFE | -29.60 |
| Consensus MFE | -13.55 |
| Energy contribution | -15.97 |
| Covariance contribution | 2.42 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.46 |
| SVM decision value | 0.01 |
| SVM RNA-class probability | 0.537972 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 1210745 105 - 22224390 UUAUCGAUAU--CGGAUGG--UAGCGAUAGAACUUAGAAACAGAUAGGCG---GCCUAUCGUCUAACGCAUAUCGCAGCCCAC-CGAAAAGUCAUCGAUGACGACUU-CUUGUA ((((((((.(--(((..((--(.((((((....(((((....((((((..---.)))))).)))))....)))))).)))..)-)))......))))))))......-...... ( -33.80) >DroSec_CAF1 3110 105 - 1 UUAUCGAUAU--CGGAUGG--UAGCGAUAGAACUUAGAAACAGAUAGGCCGCCGCCUAUCGUCUAACGCAUAUCG---CCCAC-CGAAAAGUUAUCGAUGACGACUU-CUUGUA ((((((((((--(((.(((--..((((((....(((((....(((((((....))))))).)))))....)))))---)))))-))).....)))))))))......-...... ( -38.90) >DroSim_CAF1 3558 105 - 1 UUAUCGAUAU--CGGAUGG--UAGCGAUAGAACUUAGAAACAGAUAGGCCGCCGCCUAUCGUCUAACGCAUAUCG---CCCAC-CGAAAAGUCAUCGAUGACGACUU-CUUGUA ((((((((.(--(((.(((--..((((((....(((((....(((((((....))))))).)))))....)))))---)))))-)))......))))))))......-...... ( -36.50) >DroEre_CAF1 3067 102 - 1 UUAUCGAUAU--CAGCUGA--GAACGAUAGAACUUAGAAGCUGAUAGCCC---GCCUAUCGUCUAACGCAUAUCGCAGCCCAC-CGAAAAGUCAUCGAUGACGACU----UGUA ((((((((..--..((((.--...(((((....(((((...((((((...---..)))))))))))....)))))))))....-.((....)))))))))).....----.... ( -22.20) >DroYak_CAF1 3100 109 - 1 UUAUCGAUAUCCCAGCUGAGGGAACGAUAGAACUUAGAAACAGAUAGACC---GCCUAUCGUCUAACGCAUAUCGCAGCCCAC-CGAAAAGUCAUCGAUGACGACUU-CUUGUA ((((((((.((((......)))).(((((....(((((....(((((...---..))))).)))))....)))))........-.........))))))))......-...... ( -26.50) >DroPer_CAF1 4844 88 - 1 -------CAU--CAUCUGG--UAG--------CUCAGGAAUAAUCGGGUC---GCC---CAUUUAACG-GUAUGGCAGCACGCACGGAUAGCUAUCGAUGACGACUUGCGUAUA -------..(--((((.((--(((--------((...........(((..---.))---)......((-(..((....))..).))...))))))))))))............. ( -19.70) >consensus UUAUCGAUAU__CAGAUGG__UAGCGAUAGAACUUAGAAACAGAUAGGCC___GCCUAUCGUCUAACGCAUAUCGCAGCCCAC_CGAAAAGUCAUCGAUGACGACUU_CUUGUA ((((((((................(((((....(((((....(((((((....))))))).)))))....)))))..........((....))))))))))............. (-13.55 = -15.97 + 2.42)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:44:09 2006