
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 10,983,847 – 10,983,991 |
| Length | 144 |
| Max. P | 0.821585 |

| Location | 10,983,847 – 10,983,951 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 76.39 |
| Mean single sequence MFE | -39.45 |
| Consensus MFE | -17.61 |
| Energy contribution | -18.94 |
| Covariance contribution | 1.34 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.22 |
| Structure conservation index | 0.45 |
| SVM decision value | 0.68 |
| SVM RNA-class probability | 0.821585 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
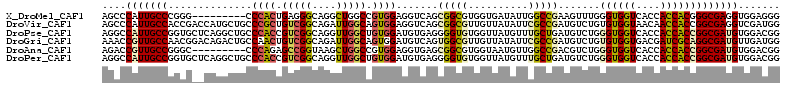
>X_DroMel_CAF1 10983847 104 + 22224390 CCCUCCACCUCGCCCGUGGUGGUGACCACCCAAACUUCGGCCAAUAUCACCACGCCGCUGACCUCCACGGCCAGCCUGCCCUCAGUGGG---------CCCGGGCAAUGGGCU ...........(((((((((((((....((........))....)))))))))(((((((.((.....)).))))..((((.....)))---------)...)))...)))). ( -42.40) >DroVir_CAF1 10514 113 + 1 CCAUCGACCUCGCCGGUGGUUGUUACCACACAGACAUCGGCGAAUAUAACAACGCCGCUGACCUCCACUGCCAAUCUGCCGACAGCGGGCAGCAUGGUCGGUGGCAAUGGGCU ((((.....(((((((((.((((......)))).)))))))))..........((((((((((....(((((...(((....)))..)))))...)))))))))).))))... ( -52.30) >DroPse_CAF1 7076 113 + 1 CCGUCCACAUCGCCGGUGGUGGUGACCACCCAGACAUCAGCAAACAUAACCACACCCCUCACAUCCACAGCCAACCUGCCGACGGUGGGCAGCCUGAGCACCGGCAAUGGCCU .......(((.((((((((((((.........................)))))..............(((.(..((..((...))..))..).)))..))))))).))).... ( -32.51) >DroGri_CAF1 8874 113 + 1 CCAUCAACAUCGCCUGCGAUCGUCACCACACAGACAUCGGCGAAUAUAACAACGCCACUGACAUCCACUGCCAAUCUGCCGACAGUUGGCAGUCUGUCCGUUGGCAACGGUUU ((.......(((((...(((.(((........)))))))))))..........(((((.((((...((((((((.(((....))))))))))).)))).).))))...))... ( -38.10) >DroAna_CAF1 11273 104 + 1 CCGUCCACAUCGCCGGUGGUGGUGACCACCCAGACGUCGGCCAACAUUACCACGCCGCUCACCUCCACGGCCAGCUUACCGGCUCUGGG---------GCCCGGCAACGGUCU ..((((.((..((((((((((((((....((.......))......)))))))((((..........)))).....)))))))..))))---------))(((....)))... ( -38.90) >DroPer_CAF1 7054 113 + 1 CCGUCCACAUCGCCGGUGGUGGUGACCACCCAGACAUCAGCAAACAUAACCACACCCCUCACAUCCACAGCCAACCUGCCGACGGUGGGCAGCCUGAGCACCGGCAAUGGCCU .......(((.((((((((((((.........................)))))..............(((.(..((..((...))..))..).)))..))))))).))).... ( -32.51) >consensus CCGUCCACAUCGCCGGUGGUGGUGACCACCCAGACAUCGGCAAACAUAACCACGCCGCUCACAUCCACAGCCAACCUGCCGACAGUGGGCAGCCUG__CACCGGCAAUGGCCU ...........((((((((((((.........................)))))......................(((((.......)))))......)))))))........ (-17.61 = -18.94 + 1.34)


| Location | 10,983,847 – 10,983,951 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 76.39 |
| Mean single sequence MFE | -48.36 |
| Consensus MFE | -27.38 |
| Energy contribution | -27.72 |
| Covariance contribution | 0.34 |
| Combinations/Pair | 1.41 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.621681 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
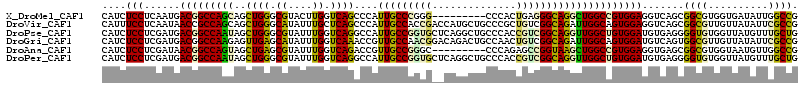
>X_DroMel_CAF1 10983847 104 - 22224390 AGCCCAUUGCCCGGG---------CCCACUGAGGGCAGGCUGGCCGUGGAGGUCAGCGGCGUGGUGAUAUUGGCCGAAGUUUGGGUGGUCACCACCACGGGCGAGGUGGAGGG ...((((((((((.(---------(((.....))))..(((((((.....))))))).(.((((((((....(((........))).))))))))).))))))..)))).... ( -50.90) >DroVir_CAF1 10514 113 - 1 AGCCCAUUGCCACCGACCAUGCUGCCCGCUGUCGGCAGAUUGGCAGUGGAGGUCAGCGGCGUUGUUAUAUUCGCCGAUGUCUGUGUGGUAACAACCACCGGCGAGGUCGAUGG ...((((((((...(((.(((((((((((((((((....))))))))))......))))))).)))....((((((.((..(((......)))..)).)))))))).)))))) ( -48.10) >DroPse_CAF1 7076 113 - 1 AGGCCAUUGCCGGUGCUCAGGCUGCCCACCGUCGGCAGGUUGGCUGUGGAUGUGAGGGGUGUGGUUAUGUUUGCUGAUGUCUGGGUGGUCACCACCACCGGCGAUGUGGACGG ...((((((((((((....(((..((((.((((((((..(((((..(.............)..)))).)..))))))))..))))..))).....))))))))))).)..... ( -48.22) >DroGri_CAF1 8874 113 - 1 AAACCGUUGCCAACGGACAGACUGCCAACUGUCGGCAGAUUGGCAGUGGAUGUCAGUGGCGUUGUUAUAUUCGCCGAUGUCUGUGUGGUGACGAUCGCAGGCGAUGUUGAUGG ....(((..((((((((((.(((((((((((....))).)))))))).........(((((..........))))).))))))).)))..)))((((....))))........ ( -46.00) >DroAna_CAF1 11273 104 - 1 AGACCGUUGCCGGGC---------CCCAGAGCCGGUAAGCUGGCCGUGGAGGUGAGCGGCGUGGUAAUGUUGGCCGACGUCUGGGUGGUCACCACCACCGGCGAUGUGGACGG ...((((((((((((---------((((..(((((....)))))..))).)))....((((((((.......))).)))))..(((((...))))).))))))))).)..... ( -48.70) >DroPer_CAF1 7054 113 - 1 AGGCCAUUGCCGGUGCUCAGGCUGCCCACCGUCGGCAGGUUGGCUGUGGAUGUGAGGGGUGUGGUUAUGUUUGCUGAUGUCUGGGUGGUCACCACCACCGGCGAUGUGGACGG ...((((((((((((....(((..((((.((((((((..(((((..(.............)..)))).)..))))))))..))))..))).....))))))))))).)..... ( -48.22) >consensus AGACCAUUGCCGGCG__CAGGCUGCCCACUGUCGGCAGGUUGGCAGUGGAGGUCAGCGGCGUGGUUAUAUUCGCCGAUGUCUGGGUGGUCACCACCACCGGCGAUGUGGACGG ....(((((((..............((((.(((((....))))).)))).......(((((..........))))).......((((((....)))))))))))))....... (-27.38 = -27.72 + 0.34)


| Location | 10,983,884 – 10,983,991 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 77.59 |
| Mean single sequence MFE | -46.27 |
| Consensus MFE | -28.47 |
| Energy contribution | -27.81 |
| Covariance contribution | -0.66 |
| Combinations/Pair | 1.55 |
| Mean z-score | -1.43 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.15 |
| SVM RNA-class probability | 0.608857 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10983884 107 - 22224390 CAUCUCCUCAAUGACGGCCAGCAGCUGGGCGUACUUGGUCAGCCCAUUGCCCGGG---------CCCACUGAGGGCAGGCUGGCCGUGGAGGUCAGCGGCGUGGUGAUAUUGGCCG .((((((......(((((((((..(((((((....(((.....))).)))))))(---------(((.....))))..)))))))))))))))...((((............)))) ( -50.70) >DroVir_CAF1 10551 116 - 1 CAUUUCCUCAAUAACCGCCAGCAGCUGGGCAUAUUUGCUCAGCCCAUUGCCACCGACCAUGCUGCCCGCUGUCGGCAGAUUGGCAGUGGAGGUCAGCGGCGUUGUUAUAUUCGCCG ..........(((((((((.((.((((((((....))))))))((((((((((((((...((.....)).))))).....)))))))))......))))))..)))))........ ( -47.50) >DroPse_CAF1 7113 116 - 1 CAUCUCCUCGAUGACGGCCAAUAGCUGGGCGUAUUUGGUCAGGCCAUUGCCGGUGCUCAGGCUGCCCACCGUCGGCAGGUUGGCUGUGGAUGUGAGGGGUGUGGUUAUGUUUGCUG ((((((((((.(.(((((((((..(((((((...((((.(((....)))))))))))))).(((((.......)))))))))))))).)...))))))).)))............. ( -45.60) >DroGri_CAF1 8911 116 - 1 CAUCUCCUCGAUGACGGCCAAGAGUUGAGCAUAUUUGGUCAAACCGUUGCCAACGGACAGACUGCCAACUGUCGGCAGAUUGGCAGUGGAUGUCAGUGGCGUUGUUAUAUUCGCCG ((((.....)))).((((...(..((((.((....)).))))..)...((((...((((.(((((((((((....))).))))))))...))))..))))............)))) ( -42.60) >DroAna_CAF1 11310 107 - 1 CAUCUCCUCGAUAACGGCCAGUAGCUGAGCGUAUUUGGUCAGACCGUUGCCGGGC---------CCCAGAGCCGGUAAGCUGGCCGUGGAGGUGAGCGGCGUGGUAAUGUUGGCCG (((((((......(((((((((..((((.((....)).))))....(((((((.(---------......))))))))))))))))))))))))..((((((....)))))).... ( -45.60) >DroPer_CAF1 7091 116 - 1 CAUCUCCUCGAUGACGGCCAAUAGCUGGGCGUAUUUGGUCAGGCCAUUGCCGGUGCUCAGGCUGCCCACCGUCGGCAGGUUGGCUGUGGAUGUGAGGGGUGUGGUUAUGUUUGCUG ((((((((((.(.(((((((((..(((((((...((((.(((....)))))))))))))).(((((.......)))))))))))))).)...))))))).)))............. ( -45.60) >consensus CAUCUCCUCGAUGACGGCCAAUAGCUGGGCGUAUUUGGUCAGACCAUUGCCGGCG__CAGGCUGCCCACUGUCGGCAGGUUGGCAGUGGAGGUCAGCGGCGUGGUUAUAUUCGCCG ....(((......(((((((((..((((.((....)).))))....(((((((((..............))))))))))))))))))))).......((((..........)))). (-28.47 = -27.81 + -0.66)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:18:25 2006