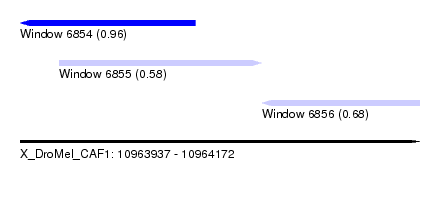
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 10,963,937 – 10,964,172 |
| Length | 235 |
| Max. P | 0.964211 |

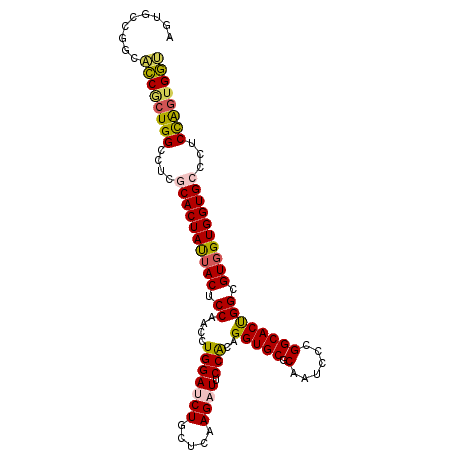
| Location | 10,963,937 – 10,964,040 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 103 |
| Reading direction | reverse |
| Mean pairwise identity | 87.06 |
| Mean single sequence MFE | -41.22 |
| Consensus MFE | -37.59 |
| Energy contribution | -37.57 |
| Covariance contribution | -0.02 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.06 |
| Structure conservation index | 0.91 |
| SVM decision value | 1.56 |
| SVM RNA-class probability | 0.964211 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10963937 103 - 22224390 AGUGCCGGCACCGCUGGCCUCGCACUAUUACUCCAAUCUGGAUCUGCUCAAGAUUCCACAGGUGCGCAAUCCCGGCACUGGCGUUGUGGUGCCCUCCAGUGGG ..........(((((((....(((((((.((.((....(((((((.....))).))))..(((((.(......)))))))).)).)))))))...))))))). ( -38.30) >DroSec_CAF1 8746 103 - 1 AGUGCCUACACCGCUGGCUUCGCACUAUUACUCCAACCUGGAUCUGCUCAAGAUUCCACAGGUGCGCAAUCCCGGCACUGGCGUGGUGGUGCCCUCCAGUGGU .........((((((((....((((((((((.((....(((((((.....))).))))..(((((.(......)))))))).))))))))))...)))))))) ( -41.70) >DroSim_CAF1 8652 103 - 1 AGUGCCUACACCGCUGGCUUCGCACUAUUACUCCAACCUGGAUCUGCUCAAGAUUCCACAGGUGCGCAAUCCCGGCACUGGCGUGGUGGUGCCCUCCAGUGGU .........((((((((....((((((((((.((....(((((((.....))).))))..(((((.(......)))))))).))))))))))...)))))))) ( -41.70) >DroEre_CAF1 8632 103 - 1 AGUGCCGGCACCACUGGCCUCGCACUAUUACUCCAACUUGGACCUGCUCAAGAUCCCACAGGUGCGCAAUCCUGGCACUGGUGUGGUGGUGACCUCCAGUGGU .........((((((((.....(((((((((.((..(((((......)))))........(((((.((....))))))))).)))))))))....)))))))) ( -38.60) >DroYak_CAF1 8626 103 - 1 AGUGCCGGCACCACUGGCCUCGCACUAUUACUCCAACUUGGAUCUGCUGAAGAUCCCACAGGUGCGCAAUCCCGGCACUGGCGUGGUGGUGCCCUCCAGUGGU .........((((((((....((((((((((.((.....((((((.....))))))....(((((.(......)))))))).))))))))))...)))))))) ( -44.50) >DroAna_CAF1 9911 95 - 1 ----C----CGCUCUGGCGGCGCACUACUACUCCAACCUGGACCUGCUGAAGAUACCGCAGGUGCGCAAUCCCGGCACCGGGGUGGUGGUGGCCUCUGUUGUG ----.----(((..(((.(((.((((((((((((.....(.((((((.(......).)))))).)((.......))...))))))))))))))).)))..))) ( -42.50) >consensus AGUGCCGGCACCGCUGGCCUCGCACUAUUACUCCAACCUGGAUCUGCUCAAGAUUCCACAGGUGCGCAAUCCCGGCACUGGCGUGGUGGUGCCCUCCAGUGGU .........((((((((....((((((((((.((....(((((((.....)))).)))..(((((.(......)))))))).))))))))))...)))))))) (-37.59 = -37.57 + -0.02)


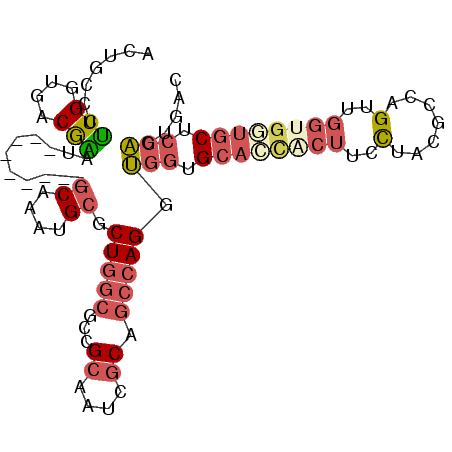
| Location | 10,963,960 – 10,964,079 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 82.01 |
| Mean single sequence MFE | -46.27 |
| Consensus MFE | -31.10 |
| Energy contribution | -31.20 |
| Covariance contribution | 0.10 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.08 |
| SVM RNA-class probability | 0.575649 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10963960 119 + 22224390 CAGUGCCGGGAUUGCGCACCUGUGGAAUCUUGAGCAGAUCCAGAUUGGAGUAAUAGUGCGAGGCCAGCGGUGCCGGCACUCCUGGCACUCCUGACACUCCCGACAUGGGCAGCUGGCAG .((((((((((.((((((((..(((...((((.((...(((.....)))......)).)))).)))..)))))..))).)))))))))).(((.(((((((.....))).)).)).))) ( -48.00) >DroSec_CAF1 8769 110 + 1 CAGUGCCGGGAUUGCGCACCUGUGGAAUCUUGAGCAGAUCCAGGUUGGAGUAAUAGUGCGAAGCCAGCGGUGUAGGCACUCCUGGCA---------CUCCCGGCAUGGGCAGCUGGCAG .((((((((((.((((((((..((((.(((.....))))))).(((((.(((....)))....))))))))))..))).))))))))---------)).(((((.......)))))... ( -48.10) >DroSim_CAF1 8675 110 + 1 CAGUGCCGGGAUUGCGCACCUGUGGAAUCUUGAGCAGAUCCAGGUUGGAGUAAUAGUGCGAAGCCAGCGGUGUAGGCACUCCUGGCA---------CUCCCGGCAUGGGCAGCUGGCAG .((((((((((.((((((((..((((.(((.....))))))).(((((.(((....)))....))))))))))..))).))))))))---------)).(((((.......)))))... ( -48.10) >DroEre_CAF1 8655 104 + 1 CAGUGCCAGGAUUGCGCACCUGUGGGAUCUUGAGCAGGUCCAAGUUGGAGUAAUAGUGCGAGGCCAGUGGUGCCGGCACUCC---------------GCCUGGCAUGGGCAGCUGGCAA (((((((((((((.(((....))).))))))).))..(((((.((..(.((...(((((..((((....).))).)))))..---------------)))..)).))))).)))).... ( -44.50) >DroYak_CAF1 8649 104 + 1 CAGUGCCGGGAUUGCGCACCUGUGGGAUCUUCAGCAGAUCCAAGUUGGAGUAAUAGUGCGAGGCCAGUGGUGCCGGCACUCC---------------GCCUGCUAUGGGCAGCUGGCAG .((((((((.((..(((((.(((.....(((((((........)))))))..)))))))(....).)..)).))))))))..---------------(((.(((......))).))).. ( -42.70) >DroAna_CAF1 9934 94 + 1 CGGUGCCGGGAUUGCGCACCUGCGGUAUCUUCAGCAGGUCCAGGUUGGAGUAGUAGUGCGCCGCCAGAGCG----G--------------------CUCCCGGCAUGGGCAUC-GGCAG (.(((((((((..((((.(((((..........)))))(((.....)))......))))(((((....)))----)--------------------)))))))))).)((...-.)).. ( -46.20) >consensus CAGUGCCGGGAUUGCGCACCUGUGGAAUCUUGAGCAGAUCCAGGUUGGAGUAAUAGUGCGAGGCCAGCGGUGCCGGCACUCC______________CUCCCGGCAUGGGCAGCUGGCAG ...((((((((((.(((....))).))))))).)))..(((.....)))(((....)))...((((((((((....))))..................(((.....)))..)))))).. (-31.10 = -31.20 + 0.10)



| Location | 10,964,079 – 10,964,172 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | reverse |
| Mean pairwise identity | 75.62 |
| Mean single sequence MFE | -37.35 |
| Consensus MFE | -21.24 |
| Energy contribution | -22.60 |
| Covariance contribution | 1.36 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.679041 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10964079 93 - 22224390 ACUGCCCUUGGUGACGAAU---------GCAAAUGCGCUGGCGCCGCAAUCGCAGCCAGGUGGUGCACCACUUCCUACGCCAGUUGGUGGUGCUCCAGUGAC ((((((((((((..(((.(---------((....(((....))).))).)))..)))))).)).((((((((..((.....))..))))))))..))))... ( -39.40) >DroSec_CAF1 8879 93 - 1 ACUGCCCUUGGUGACGAAU---------GCAAAUGCGCUGGCGCCGCAAUCGCAGCCAGGUGGUGCACCGCUUCCUACACCAGUUGGUGGUGCUCCAGUGAG ((((((((((((..(((.(---------((....(((....))).))).)))..)))))).)).((((((((..((.....))..))))))))..))))... ( -39.00) >DroSim_CAF1 8785 93 - 1 ACUGCCCUUGGUGACGAAU---------GCAAAUGCGCUGGCGCCGCAAUCGCAGCCAGGUGGUGCACCACUUCCUACACCAGUUGGUGGUGCUCCAGUGAG ((((((((((((..(((.(---------((....(((....))).))).)))..)))))).)).((((((((..((.....))..))))))))..))))... ( -39.40) >DroEre_CAF1 8759 93 - 1 ACUGCCCUUGGUGACGGGC---------GCAAAUGCGCUGGCGCCGCAAACGCAGCCAGGUGGUGCAGUACUUGCGACGCCAGGUGGAGGUGCUCCAGUGCC ..(((.((((....)))).---------)))...(((((((((((((....)).(((.((((.(((((...))))).)))).)))...)))))..)))))). ( -40.50) >DroYak_CAF1 8753 93 - 1 ACUGCCCUUGGUGACGGGU---------GCAAAUGCGCUGGCGCCGCAACCGCAGCCAGGUGGUGCAGCACUUACGACGCCAGGUGGUGUUGCUCCAGUGCC ..(((.((((....)))).---------)))...(((((((.((.(((.((((......))))))).)).....(((((((....)))))))..))))))). ( -42.00) >DroPer_CAF1 14818 94 - 1 GCUGCCCCUGGUAACAGCUCCUGCCGGACCAGCUGCUCUCUCACUGCAAGCUCUUCCAGGGCUCGUUCCAGCUCUCGUUCCAGUU------CCUCUUCU--C .......((((.(((.((....)).(((..((((((.........)).))))..)))((((((......)))))).)))))))..------........--. ( -23.80) >consensus ACUGCCCUUGGUGACGAAU_________GCAAAUGCGCUGGCGCCGCAAUCGCAGCCAGGUGGUGCACCACUUCCUACGCCAGUUGGUGGUGCUCCAGUGAC .......(((....)))...........((....)).(((((...((....)).))))).(((.((((((((..(.......)..)))))))).)))..... (-21.24 = -22.60 + 1.36)

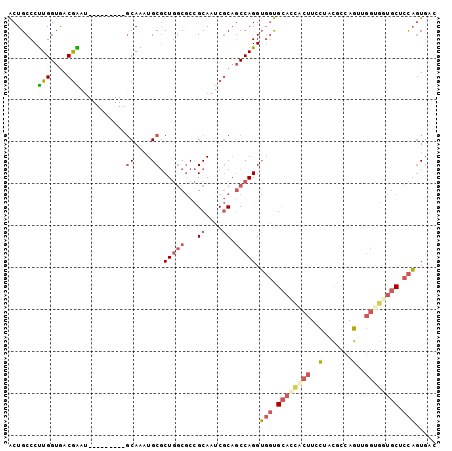
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:18:15 2006