
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 10,959,802 – 10,959,901 |
| Length | 99 |
| Max. P | 0.920241 |

| Location | 10,959,802 – 10,959,901 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 82.98 |
| Mean single sequence MFE | -49.63 |
| Consensus MFE | -38.00 |
| Energy contribution | -39.25 |
| Covariance contribution | 1.25 |
| Combinations/Pair | 1.18 |
| Mean z-score | -3.07 |
| Structure conservation index | 0.77 |
| SVM decision value | 1.13 |
| SVM RNA-class probability | 0.920241 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
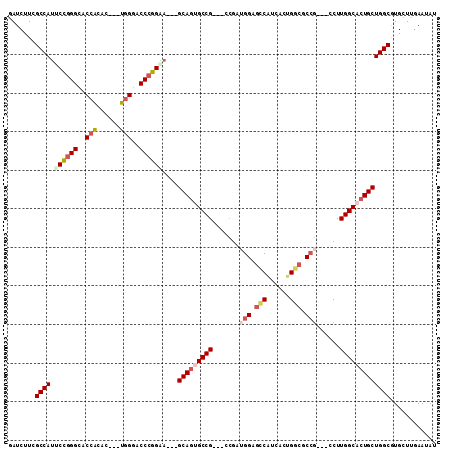
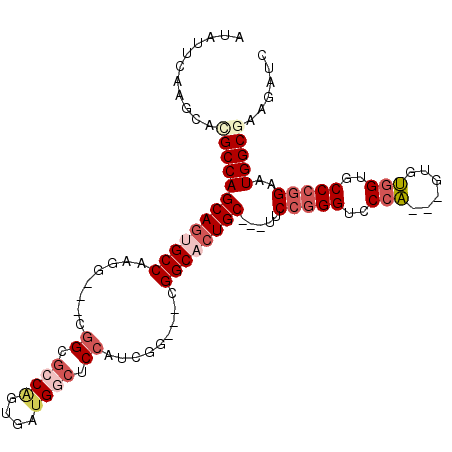
>X_DroMel_CAF1 10959802 99 + 22224390 GAUCUUCGCCAUUCCGGGCACCACAC---UGGGACCCGGAA---GCAGUGCCG---CAGAUGGAGCCAUCACUGGCGCCG---CCUUGGCACUGCUGGCGUGCUUAAAUAU ......((((((((((((..((....---.))..)))))))---(((((((((---.((.(((.((((....)))).)))---.))))))))))))))))........... ( -52.20) >DroSec_CAF1 4602 99 + 1 AAUCUUAGCCAUCCCGGGCACCACAC---UGGGACCCGGAA---GCAGUGCCG---CCGAUGGAGCCAUCACUGGCGCCG---CCUUGGCACUGCUGGCAUGCUUGAAUAU .......(((...(((((..((....---.))..))))).(---(((((((((---..(.(((.((((....)))).)))---)..)))))))))))))............ ( -47.40) >DroSim_CAF1 4733 99 + 1 GAUCUUCGCCAUUCCGGGCACCACAC---UGGGACCCGGAA---GCAGUGCCG---CCGAUGGAGCCAUCACUGGCGCCG---CCUUGGCACUGCUGGCGUGCUUGAAUAU ......((((((((((((..((....---.))..)))))))---(((((((((---..(.(((.((((....)))).)))---)..))))))))))))))........... ( -50.50) >DroEre_CAF1 4509 99 + 1 GAUCUUCGCCAUACCUGGCACCACAC---UGGGACCCGGAA---GCAGUGCCG---CCGAUGGAGGCGCCGCUGGCGCCG---CCUUGGCACUGCUGGCAUGCUUGAAUAU .......(((...((.((..((....---.))..)).)).(---(((((((((---.....((.((((((...)))))).---)).)))))))))))))............ ( -46.00) >DroYak_CAF1 4525 99 + 1 GAUCUUCGCCAUUCCGGGCACCACAC---UGGGACCCGGGA---GCAGGGCCG---CCGAGGGUGCCAUCGCUGGCGCGG---CCUUGGCACUGCUGGCGUGCUUGAAAAU ......((((((((((((..((....---.))..)))))))---((((....(---((((((((((((....))))))..---))))))).)))))))))........... ( -49.70) >DroAna_CAF1 5535 111 + 1 GAUCUUGGCCAGGCUGGGCACUGGAUUACCCGCUCCCGGUACCGGCAUGGCCGCAGCCGCAGGCGUCACCACCGGGGCCGAGUGCUUGGCACUGCUGGCGUGCUUGAAGAU .((((((((((.(((((..(((((...........))))).))))).)))))((((((((((..((((.(((((....)).)))..)))).)))).))).)))...))))) ( -52.00) >consensus GAUCUUCGCCAUUCCGGGCACCACAC___UGGGACCCGGAA___GCAGUGCCG___CCGAUGGAGCCAUCACUGGCGCCG___CCUUGGCACUGCUGGCGUGCUUGAAUAU .......((((.((((((..(((......)))..))))))....(((((((((.......(((.(((......))).)))......)))))))))))))............ (-38.00 = -39.25 + 1.25)


| Location | 10,959,802 – 10,959,901 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 82.98 |
| Mean single sequence MFE | -50.13 |
| Consensus MFE | -36.25 |
| Energy contribution | -37.95 |
| Covariance contribution | 1.70 |
| Combinations/Pair | 1.11 |
| Mean z-score | -3.08 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.97 |
| SVM RNA-class probability | 0.892494 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10959802 99 - 22224390 AUAUUUAAGCACGCCAGCAGUGCCAAGG---CGGCGCCAGUGAUGGCUCCAUCUG---CGGCACUGC---UUCCGGGUCCCA---GUGUGGUGCCCGGAAUGGCGAAGAUC ...........(((((((((((((.((.---.((.((((....)))).))..)).---.))))))))---(((((((..((.---....))..))))))))))))...... ( -56.40) >DroSec_CAF1 4602 99 - 1 AUAUUCAAGCAUGCCAGCAGUGCCAAGG---CGGCGCCAGUGAUGGCUCCAUCGG---CGGCACUGC---UUCCGGGUCCCA---GUGUGGUGCCCGGGAUGGCUAAGAUU ............((((((((((((..(.---.((.((((....)))).))..)..---.))))))))---(((((((..((.---....))..)))))))))))....... ( -51.20) >DroSim_CAF1 4733 99 - 1 AUAUUCAAGCACGCCAGCAGUGCCAAGG---CGGCGCCAGUGAUGGCUCCAUCGG---CGGCACUGC---UUCCGGGUCCCA---GUGUGGUGCCCGGAAUGGCGAAGAUC ...........(((((((((((((..(.---.((.((((....)))).))..)..---.))))))))---(((((((..((.---....))..))))))))))))...... ( -54.50) >DroEre_CAF1 4509 99 - 1 AUAUUCAAGCAUGCCAGCAGUGCCAAGG---CGGCGCCAGCGGCGCCUCCAUCGG---CGGCACUGC---UUCCGGGUCCCA---GUGUGGUGCCAGGUAUGGCGAAGAUC ...(((...(((((((((((((((..((---.((((((...)))))).)).....---.))))))))---)....((..((.---....))..)).))))))..))).... ( -45.00) >DroYak_CAF1 4525 99 - 1 AUUUUCAAGCACGCCAGCAGUGCCAAGG---CCGCGCCAGCGAUGGCACCCUCGG---CGGCCCUGC---UCCCGGGUCCCA---GUGUGGUGCCCGGAAUGGCGAAGAUC ...........(((((((((.(((...(---(((.((((....)))).....)))---)))).))))---..(((((..((.---....))..)))))..)))))...... ( -45.30) >DroAna_CAF1 5535 111 - 1 AUCUUCAAGCACGCCAGCAGUGCCAAGCACUCGGCCCCGGUGGUGACGCCUGCGGCUGCGGCCAUGCCGGUACCGGGAGCGGGUAAUCCAGUGCCCAGCCUGGCCAAGAUC (((((...(((.(((.(((((((...))))........((((....)))))))))))))(((((.((.(((((..(((........))).)))))..)).)))))))))). ( -48.40) >consensus AUAUUCAAGCACGCCAGCAGUGCCAAGG___CGGCGCCAGUGAUGGCUCCAUCGG___CGGCACUGC___UUCCGGGUCCCA___GUGUGGUGCCCGGAAUGGCGAAGAUC ...........(((((((((((((........((.((((....)))).)).........)))))))).....(((((..(((......)))..)))))..)))))...... (-36.25 = -37.95 + 1.70)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:18:11 2006