
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 10,904,490 – 10,904,604 |
| Length | 114 |
| Max. P | 0.745438 |

| Location | 10,904,490 – 10,904,604 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.97 |
| Mean single sequence MFE | -35.98 |
| Consensus MFE | -18.74 |
| Energy contribution | -18.13 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.39 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.52 |
| SVM decision value | 0.46 |
| SVM RNA-class probability | 0.745438 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10904490 114 + 22224390 UACAUACUUCCAUGGUUGUAAUCACAUAAACAGCAGAGGAUUAACAGCAGAACAAAUGCGAUUGUUGUG---GUGCUUUUGCGAGUGUGUAUUGCA---GUUAUGGUUGCCGCCGUAGAA .........((((..((((((((((((.....(((((((.....((.((.(((((......))))).))---.)))))))))..))))).))))))---)..)))).............. ( -32.40) >DroPse_CAF1 15791 117 + 1 UACGUACUUCCAUAGUUGUAAUCGCACAAGCAGCAGAGGACCAGCAGCAGCACAAAUGUGACUGUUGUG---GUGCCCCUGCGUGUAUGUAUGGUUGUGGCAGUGGCAGCGGCUGUGGCG ..(((....((((.((..((((((.(((.(((((((.((((((.(((((((((....))).))))))))---)).)).)))).))).))).))))))..)).))))..)))((....)). ( -49.70) >DroEre_CAF1 1471 114 + 1 UACAUACUUCCAUAGUUGUAAUCACAUAAACAGCAGAGGAUUAACAGCAGAACAAAUGCGAUUGUUGUG---GUGCUUUUGCGAGUGUGUAUUGCA---GUUAUGGUAGCCGCCGUAGAA ......((.(((((..(((((((((((.....(((((((.....((.((.(((((......))))).))---.)))))))))..))))).))))))---..))))).))........... ( -33.20) >DroYak_CAF1 3054 114 + 1 UACAUACUUCCAUAGUUGUAAUCACAUAAACAGCAGAGGAUUAACAGCAGAACAAAUGCGAUUGUUGUG---GUGCUUUUGCGAGUGUGUAUUGCA---GUUAUGGUUGCUGCCGUAGAA .........(((((..(((((((((((.....(((((((.....((.((.(((((......))))).))---.)))))))))..))))).))))))---..)))))((((....)))).. ( -32.40) >DroAna_CAF1 2230 110 + 1 GACAUACUUCCAUAAUUGUAAUCACAUAAACAACAAAGAAUUAAAAGCAAAAUAAAUGCCACUGUUGUUGCUGUUGUUUUGCGUGUAUAUAUCGGA---GUGAUC------ACUGUAGC- ....((((((.(((..((((..(((.....(......)........(((((((((..((.((....)).))..))))))))))))))))))).)))---)))...------........- ( -18.50) >DroPer_CAF1 9813 117 + 1 UACGUACUUCCAUAGUUGUAAUCGCACAAGCAGCAGAGGACCAGCAGCAGCACAAAUGUGACUGUUGUG---GUGCCCCUGCGUGUAUGUAUGGUUGUGGCAGUGGCAGCGGCUGUGGCG ..(((....((((.((..((((((.(((.(((((((.((((((.(((((((((....))).))))))))---)).)).)))).))).))).))))))..)).))))..)))((....)). ( -49.70) >consensus UACAUACUUCCAUAGUUGUAAUCACAUAAACAGCAGAGGAUUAACAGCAGAACAAAUGCGACUGUUGUG___GUGCUUUUGCGAGUAUGUAUUGCA___GUUAUGGUAGCCGCCGUAGAA (((((((.........(((....)))......(((((((((..(((((((...........)))))))....)).)))))))..)))))))...........(((((....))))).... (-18.74 = -18.13 + -0.61)

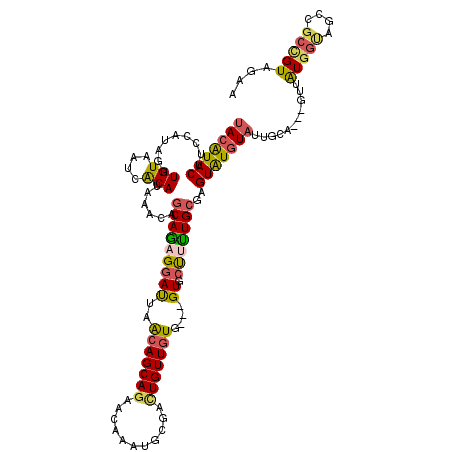
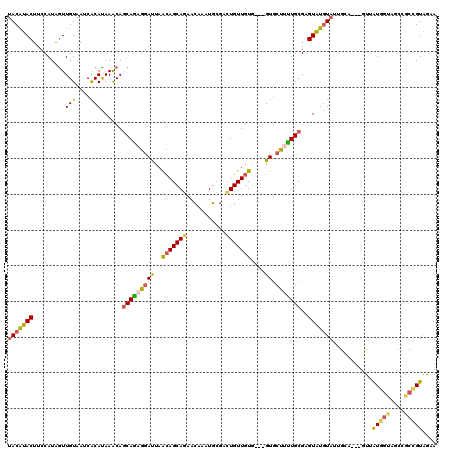
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:17:47 2006