
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 10,683,090 – 10,683,229 |
| Length | 139 |
| Max. P | 0.999815 |

| Location | 10,683,090 – 10,683,196 |
|---|---|
| Length | 106 |
| Sequences | 5 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 73.88 |
| Mean single sequence MFE | -30.70 |
| Consensus MFE | -9.73 |
| Energy contribution | -10.98 |
| Covariance contribution | 1.25 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.49 |
| Structure conservation index | 0.32 |
| SVM decision value | 2.48 |
| SVM RNA-class probability | 0.994502 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10683090 106 + 22224390 GGUCGAUGGCUAUCUAAUCGGACUGACUGCAUCGCAUCCAGACGUACAUACAUAUAUAUGUACGUACACAUGUGCGCCAUAUAU--AUGACCAUG---GUGCUAUGUGCUU ((.(((((.(..((..........))..))))))...))..(((((((((......))))))))).((((((.(((((((....--......)))---))))))))))... ( -33.00) >DroSec_CAF1 8507 96 + 1 GGUCGAUGGCUAUCUAAUCGCACUGACUGCAUCGCAUCCAGACGUACA--------UAUGUACGUACGCAUUUGCGCCAUAUAU----GGCAAUG---GUGCUAUGUGCUU ((.(((((.(..((..........))..))))))...))..((((((.--------...))))))..((((..(((((((....----....)))---))))...)))).. ( -30.20) >DroSim_CAF1 4023 100 + 1 GCUCGAUGGCUAUCUAAUCGCACUGACUGCAUCGCAUCCAGACGUACA--------UAUGUACGUGCACAUGUGCGCCAUAUAUGGAUGGCAAUG---GUGCUAUGUGCUU ...((((.........))))..(((..(((...)))..)))((((((.--------...))))))(((((((.(((((((............)))---))))))))))).. ( -31.60) >DroEre_CAF1 9071 86 + 1 GCUCGAUGGCUAUCUAAUCGGACGGACUGCA-----UCCACAUGUACA--------UAUGUACGAACACAUAUGCGCCGUACAU----G-------GUGUACUAUGUGU-A (..((((.........))))..)(((.....-----)))...(((((.--------...))))).((((((((((((((....)----)-------))))).)))))))-. ( -25.80) >DroYak_CAF1 4595 85 + 1 UGUCGAUGGCUAUCUAAUCGGACUGAGUGCA-----UCCA--GGCACA--------UAU------ACACAUAUGAGCCAAUGAU----GGCUAUGUGUGUACUGUGUGC-A ....(((((((.(((....)))...))).))-----))..--.(((((--------(((------((((((((.(((((....)----))))))))))))).)))))))-. ( -32.90) >consensus GGUCGAUGGCUAUCUAAUCGGACUGACUGCAUCGCAUCCAGACGUACA________UAUGUACGUACACAUAUGCGCCAUAUAU____GGCAAUG___GUGCUAUGUGCUU ...((((.........)))).....................(((((((..........))))))).((((((.((((.....................))))))))))... ( -9.73 = -10.98 + 1.25)



| Location | 10,683,090 – 10,683,196 |
|---|---|
| Length | 106 |
| Sequences | 5 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 73.88 |
| Mean single sequence MFE | -28.68 |
| Consensus MFE | -8.47 |
| Energy contribution | -10.16 |
| Covariance contribution | 1.69 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.30 |
| SVM decision value | 1.80 |
| SVM RNA-class probability | 0.978020 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10683090 106 - 22224390 AAGCACAUAGCAC---CAUGGUCAU--AUAUAUGGCGCACAUGUGUACGUACAUAUAUAUGUAUGUACGUCUGGAUGCGAUGCAGUCAGUCCGAUUAGAUAGCCAUCGACC ..((((((.((.(---((((.....--...))))).))..))))))((((((((((....))))))))))..((...((((((.(((..........))).).))))).)) ( -32.00) >DroSec_CAF1 8507 96 - 1 AAGCACAUAGCAC---CAUUGCC----AUAUAUGGCGCAAAUGCGUACGUACAUA--------UGUACGUCUGGAUGCGAUGCAGUCAGUGCGAUUAGAUAGCCAUCGACC ..((((...((((---((..(((----(....))))......((((((((....)--------))))))).))).)))(((...))).)))).....(((....))).... ( -26.70) >DroSim_CAF1 4023 100 - 1 AAGCACAUAGCAC---CAUUGCCAUCCAUAUAUGGCGCACAUGUGCACGUACAUA--------UGUACGUCUGGAUGCGAUGCAGUCAGUGCGAUUAGAUAGCCAUCGAGC ..((((...((..---((((((.(((((......((((....))))((((((...--------.)))))).)))))))))))..))..)))).....(((....))).... ( -31.20) >DroEre_CAF1 9071 86 - 1 U-ACACAUAGUACAC-------C----AUGUACGGCGCAUAUGUGUUCGUACAUA--------UGUACAUGUGGA-----UGCAGUCCGUCCGAUUAGAUAGCCAUCGAGC .-.(((((.(((((.-------.----(((((((((((....))).)))))))).--------))))))))))((-----(((.(((.(......).))).).)))).... ( -28.00) >DroYak_CAF1 4595 85 - 1 U-GCACACAGUACACACAUAGCC----AUCAUUGGCUCAUAUGUGU------AUA--------UGUGCC--UGGA-----UGCACUCAGUCCGAUUAGAUAGCCAUCGACA .-(((((..(((((((.((((((----(....))))).)).)))))------)).--------))))).--((((-----(.......)))))....(((....))).... ( -25.50) >consensus AAGCACAUAGCAC___CAUUGCC____AUAUAUGGCGCACAUGUGUACGUACAUA________UGUACGUCUGGAUGCGAUGCAGUCAGUCCGAUUAGAUAGCCAUCGACC ..((.....))......................((((((.((.((.(((((((..........))))))).)).))....)))((((.....)))).....)))....... ( -8.47 = -10.16 + 1.69)



| Location | 10,683,130 – 10,683,229 |
|---|---|
| Length | 99 |
| Sequences | 5 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 75.44 |
| Mean single sequence MFE | -30.54 |
| Consensus MFE | -12.57 |
| Energy contribution | -14.02 |
| Covariance contribution | 1.45 |
| Combinations/Pair | 1.17 |
| Mean z-score | -3.41 |
| Structure conservation index | 0.41 |
| SVM decision value | 4.15 |
| SVM RNA-class probability | 0.999815 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10683130 99 + 22224390 GACGUACAUACAUAUAUAUGUACGUACACAUGUGCGCCAUAUAU--AUGACCAUG---GUGCUAUGUGCUUCAGAGAUUCACUGGAAU----AAGUGGUAGGUAAUUC .(((((((((......))))))))).((((((.(((((((....--......)))---))))))))))..........(((((.....----.))))).......... ( -29.60) >DroSec_CAF1 8547 89 + 1 GACGUACA--------UAUGUACGUACGCAUUUGCGCCAUAUAU----GGCAAUG---GUGCUAUGUGCUUCAGAGAUUCACUGGAAU----CAGUGGUAGGUAAUUC .((((((.--------...))))))..((((..(((((((....----....)))---))))...)))).........((((((....----)))))).......... ( -28.50) >DroSim_CAF1 4063 93 + 1 GACGUACA--------UAUGUACGUGCACAUGUGCGCCAUAUAUGGAUGGCAAUG---GUGCUAUGUGCUUCAGAGAUUCACUGGAAU----CAGUGGUAGGUAAUUC .((((((.--------...))))))(((((((.(((((((............)))---))))))))))).........((((((....----)))))).......... ( -32.00) >DroEre_CAF1 9106 84 + 1 CAUGUACA--------UAUGUACGAACACAUAUGCGCCGUACAU----G-------GUGUACUAUGUGU-ACAGAGAAUCACUGGAAU----CAGUGGUAGGUAAUUC ...((((.--------...))))(.((((((((((((((....)----)-------))))).)))))))-.).....(((((((....----)))))))......... ( -27.10) >DroYak_CAF1 4630 87 + 1 --GGCACA--------UAU------ACACAUAUGAGCCAAUGAU----GGCUAUGUGUGUACUGUGUGC-ACAGAGAAUCACUGGAAUCCAUCAGUGGUAGGUAAUUC --.(((((--------(((------((((((((.(((((....)----))))))))))))).)))))))-.......((((((((......))))))))......... ( -35.50) >consensus GACGUACA________UAUGUACGUACACAUAUGCGCCAUAUAU____GGCAAUG___GUGCUAUGUGCUUCAGAGAUUCACUGGAAU____CAGUGGUAGGUAAUUC .(((((((..........))))))).((((((.((((.....................))))))))))..........((((((........)))))).......... (-12.57 = -14.02 + 1.45)

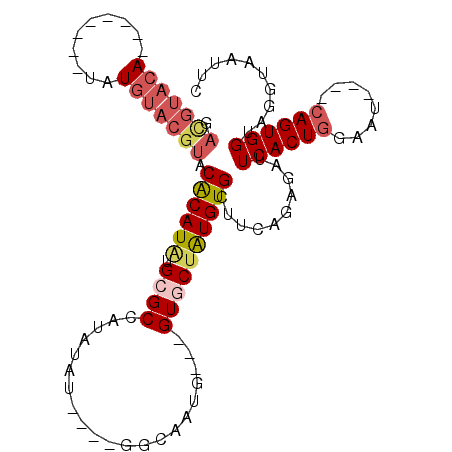
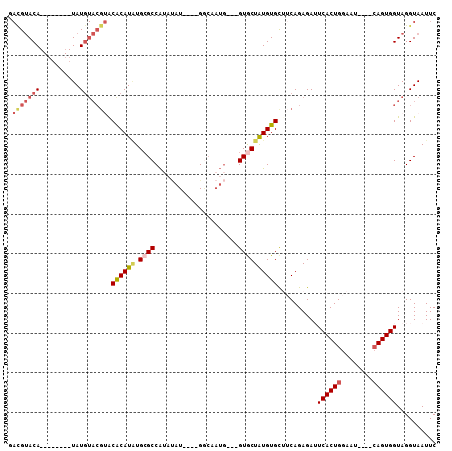
| Location | 10,683,130 – 10,683,229 |
|---|---|
| Length | 99 |
| Sequences | 5 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 75.44 |
| Mean single sequence MFE | -25.44 |
| Consensus MFE | -8.31 |
| Energy contribution | -9.48 |
| Covariance contribution | 1.17 |
| Combinations/Pair | 1.22 |
| Mean z-score | -3.35 |
| Structure conservation index | 0.33 |
| SVM decision value | 3.95 |
| SVM RNA-class probability | 0.999724 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10683130 99 - 22224390 GAAUUACCUACCACUU----AUUCCAGUGAAUCUCUGAAGCACAUAGCAC---CAUGGUCAU--AUAUAUGGCGCACAUGUGUACGUACAUAUAUAUGUAUGUACGUC ...........((((.----.....))))..........((((((.((.(---((((.....--...))))).))..))))))((((((((((....)))))))))). ( -27.50) >DroSec_CAF1 8547 89 - 1 GAAUUACCUACCACUG----AUUCCAGUGAAUCUCUGAAGCACAUAGCAC---CAUUGCC----AUAUAUGGCGCAAAUGCGUACGUACAUA--------UGUACGUC ...........(((((----....)))))..........(((....((.(---(((....----....)))).))...)))..((((((...--------.)))))). ( -21.60) >DroSim_CAF1 4063 93 - 1 GAAUUACCUACCACUG----AUUCCAGUGAAUCUCUGAAGCACAUAGCAC---CAUUGCCAUCCAUAUAUGGCGCACAUGUGCACGUACAUA--------UGUACGUC ...........(((((----....)))))..........((((((.((.(---(((............)))).))..))))))((((((...--------.)))))). ( -26.40) >DroEre_CAF1 9106 84 - 1 GAAUUACCUACCACUG----AUUCCAGUGAUUCUCUGU-ACACAUAGUACAC-------C----AUGUACGGCGCAUAUGUGUUCGUACAUA--------UGUACAUG ...........(((((----....))))).........-...(((.(((((.-------.----(((((((((((....))).)))))))).--------)))))))) ( -24.00) >DroYak_CAF1 4630 87 - 1 GAAUUACCUACCACUGAUGGAUUCCAGUGAUUCUCUGU-GCACACAGUACACACAUAGCC----AUCAUUGGCUCAUAUGUGU------AUA--------UGUGCC-- (((((((...(((....)))......))))))).....-(((((..(((((((.((((((----(....))))).)).)))))------)).--------))))).-- ( -27.70) >consensus GAAUUACCUACCACUG____AUUCCAGUGAAUCUCUGAAGCACAUAGCAC___CAUUGCC____AUAUAUGGCGCACAUGUGUACGUACAUA________UGUACGUC ...........(((((........)))))..........((.....)).......................((((....))))(((((((..........))))))). ( -8.31 = -9.48 + 1.17)

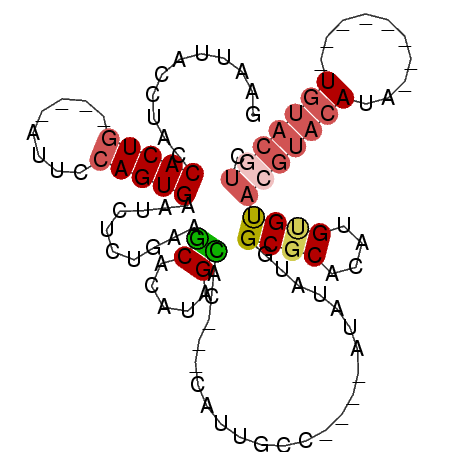
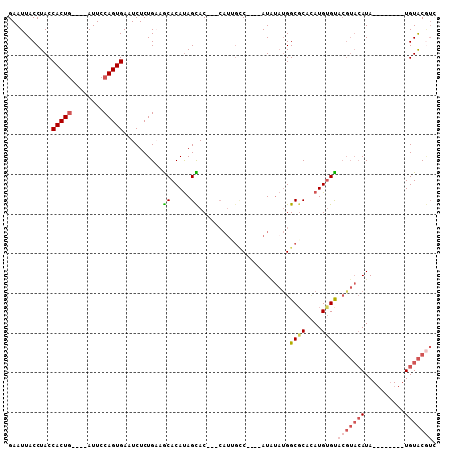
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:14:55 2006