
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 10,597,154 – 10,597,336 |
| Length | 182 |
| Max. P | 0.975688 |

| Location | 10,597,154 – 10,597,256 |
|---|---|
| Length | 102 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 89.19 |
| Mean single sequence MFE | -34.54 |
| Consensus MFE | -25.55 |
| Energy contribution | -26.03 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.02 |
| Structure conservation index | 0.74 |
| SVM decision value | 1.75 |
| SVM RNA-class probability | 0.975688 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10597154 102 + 22224390 UGUUUCGUGCGAUAAUAGCGAUAGUCGUACGGUGAUUUGAUUGCCGAUUA----------UGACUGCGCUGUAUCG------GUAUCGAUAGCAAUCGAAAUGCACCUAAAAC--UGAUC .((((.((((((((.(((((.(((((((((((..((...))..)))..))----------))))))))))))))).------...(((((....)))))...))))...))))--..... ( -35.40) >DroSec_CAF1 6029 102 + 1 UGUUUUGUGCGAUAAUAGCGAAAGUCGUACGGUGAUUUGAUUGCCGAUUA----------UGACUGCGCUGUAUCG------GUAUCGAUAGCAAUCGAAAUGCACCUAAAAC--UGAUC .(((((((((((((.(((((..((((((((((..((...))..)))..))----------))))).))))))))).------...(((((....)))))...))))..)))))--..... ( -34.20) >DroSim_CAF1 6525 102 + 1 UGUUUUGUGCGAUAAUAGCGAUAGUCGUACGGUGAUUUGAUUGCCGAUUA----------UGACUGCGCUGUAUCG------GUAUCGAUAGCAAUCGAAAUGCACCUAAAAC--UGAUC .(((((((((((((.(((((.(((((((((((..((...))..)))..))----------))))))))))))))).------...(((((....)))))...))))..)))))--..... ( -36.40) >DroEre_CAF1 6055 102 + 1 UGUUUCGUGCAAUAAUAGCGAUAGUCGUACGGUGAUUUGAUAGCCGAUUA----------UGACUGCGCUGUAUCG------GUAUCGAUAACAAUCGAAAUGCACCUAAAAC--UGAUC .((((.(((((...((((((.((((((((((((.........))))..))----------))))))))))))....------...(((((....)))))..)))))...))))--..... ( -32.00) >DroYak_CAF1 6177 120 + 1 UGUUUCGUGCGAUAAAAGCGAUAGUCGUACGGUGACUUGGUUGCCUGUUAUGACUUGUUAUGACUGCGCUGUAUCGGUAUCGGUAUCGAUAGCAAUCGAAAUGCACCUAAAAUCAUGAUC ......(((((((..........)))))))((((..((((((((..(((((((....))))))).......((((((((....))))))))))))))))....))))............. ( -34.70) >consensus UGUUUCGUGCGAUAAUAGCGAUAGUCGUACGGUGAUUUGAUUGCCGAUUA__________UGACUGCGCUGUAUCG______GUAUCGAUAGCAAUCGAAAUGCACCUAAAAC__UGAUC ......((((((((.(((((.((((((..(((..((...))..)))..............)))))))))))))))..........(((((....)))))...)))).............. (-25.55 = -26.03 + 0.48)

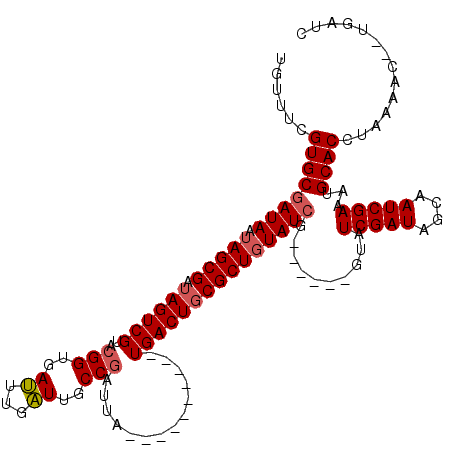
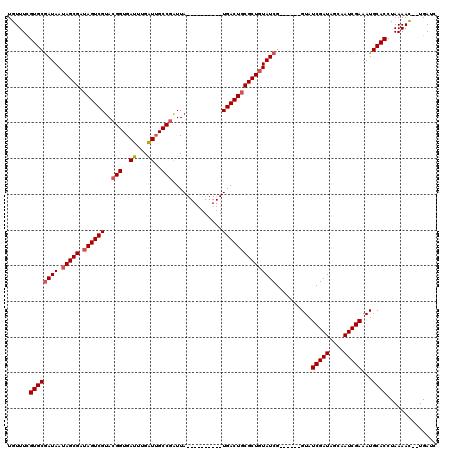
| Location | 10,597,154 – 10,597,256 |
|---|---|
| Length | 102 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.19 |
| Mean single sequence MFE | -24.20 |
| Consensus MFE | -19.65 |
| Energy contribution | -19.65 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.47 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.22 |
| SVM RNA-class probability | 0.637969 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10597154 102 - 22224390 GAUCA--GUUUUAGGUGCAUUUCGAUUGCUAUCGAUAC------CGAUACAGCGCAGUCA----------UAAUCGGCAAUCAAAUCACCGUACGACUAUCGCUAUUAUCGCACGAAACA .....--(((((..((((...(((((....)))))...------.((((.((((.((((.----------....(((...........)))...))))..))))..))))))))))))). ( -25.10) >DroSec_CAF1 6029 102 - 1 GAUCA--GUUUUAGGUGCAUUUCGAUUGCUAUCGAUAC------CGAUACAGCGCAGUCA----------UAAUCGGCAAUCAAAUCACCGUACGACUUUCGCUAUUAUCGCACAAAACA .....--((((...((((...(((((....)))))...------.((((.((((.((((.----------....(((...........)))...))))..))))..)))))))).)))). ( -24.80) >DroSim_CAF1 6525 102 - 1 GAUCA--GUUUUAGGUGCAUUUCGAUUGCUAUCGAUAC------CGAUACAGCGCAGUCA----------UAAUCGGCAAUCAAAUCACCGUACGACUAUCGCUAUUAUCGCACAAAACA .....--((((...((((...(((((....)))))...------.((((.((((.((((.----------....(((...........)))...))))..))))..)))))))).)))). ( -24.80) >DroEre_CAF1 6055 102 - 1 GAUCA--GUUUUAGGUGCAUUUCGAUUGUUAUCGAUAC------CGAUACAGCGCAGUCA----------UAAUCGGCUAUCAAAUCACCGUACGACUAUCGCUAUUAUUGCACGAAACA .....--(((((..(((((..(((((....)))))...------......((((.((((.----------....(((...........)))...))))..)))).....)))))))))). ( -22.90) >DroYak_CAF1 6177 120 - 1 GAUCAUGAUUUUAGGUGCAUUUCGAUUGCUAUCGAUACCGAUACCGAUACAGCGCAGUCAUAACAAGUCAUAACAGGCAACCAAGUCACCGUACGACUAUCGCUUUUAUCGCACGAAACA ..............((((...(((((....)))))..........((((.((((.((((.......((....)).(((......))).......))))..))))..))))))))...... ( -23.40) >consensus GAUCA__GUUUUAGGUGCAUUUCGAUUGCUAUCGAUAC______CGAUACAGCGCAGUCA__________UAAUCGGCAAUCAAAUCACCGUACGACUAUCGCUAUUAUCGCACGAAACA .......(((((..((((...(((((....)))))..........((((.((((.((((...............(((...........)))...))))..))))..))))))))))))). (-19.65 = -19.65 + 0.00)



| Location | 10,597,220 – 10,597,336 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.73 |
| Mean single sequence MFE | -31.50 |
| Consensus MFE | -26.70 |
| Energy contribution | -27.02 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.27 |
| Structure conservation index | 0.85 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.524984 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10597220 116 - 22224390 AUCGCUUAUCGAAAAGAUCGCAGUCUGUCAAUCGGAAUCUGAUAGCCACAGUCUGUCAGGCGGUUUCCCUAAUGUUUGAUGAUCA--GUUUUAGGUGCAUUUCGAUUGCUAUCGAUAC-- ...((..((((((((((((((..((((.....))))..((((((((....).)))))))))))))).(((((...(((.....))--)..)))))....))))))).)).........-- ( -31.80) >DroSec_CAF1 6095 116 - 1 AUCGCUUAUCGAAAAGAUCGCAGUCUGUCAAUCGGAAUCUGAUAGCCACAGUCUGUCAGGCGGUUUCCCUAAUGUUUGAUGAUCA--GUUUUAGGUGCAUUUCGAUUGCUAUCGAUAC-- ...((..((((((((((((((..((((.....))))..((((((((....).)))))))))))))).(((((...(((.....))--)..)))))....))))))).)).........-- ( -31.80) >DroSim_CAF1 6591 116 - 1 AUCGCUUAUCGAAAAGAUCGCAGUCUGUCAAUCGGAAUCUGAUAGCCACAGUCUGUCAGGCGGUUUCCCUAAUGUUUGAUGAUCA--GUUUUAGGUGCAUUUCGAUUGCUAUCGAUAC-- ...((..((((((((((((((..((((.....))))..((((((((....).)))))))))))))).(((((...(((.....))--)..)))))....))))))).)).........-- ( -31.80) >DroEre_CAF1 6121 116 - 1 AUCGCUUAUCGAAAAGAUCGCAGUCUGUGAAUCGGAAUCUGAUAGCCACAGUCUUUCAGGCGGUUUACCUAAUGUUUGAUGAUCA--GUUUUAGGUGCAUUUCGAUUGUUAUCGAUAC-- ((((...((((((((((((((((.(((((.(((((...)))))...))))).)).....)))))))((((((...(((.....))--)..))))))...)))))))......))))..-- ( -31.20) >DroYak_CAF1 6257 120 - 1 AUCGCUUAUCGAAAAGAAUGCAGUCUGUGCAUCGGAAUCUGAUAGCCACAGUCCGUCAGGUGGUUUCCCUAAUGUUUAAUGAUCAUGAUUUUAGGUGCAUUUCGAUUGCUAUCGAUACCG ......((((((.......((((((.(((((((((((((((((((((((..........))))))................)))).)))))).)))))))...))))))..))))))... ( -30.89) >consensus AUCGCUUAUCGAAAAGAUCGCAGUCUGUCAAUCGGAAUCUGAUAGCCACAGUCUGUCAGGCGGUUUCCCUAAUGUUUGAUGAUCA__GUUUUAGGUGCAUUUCGAUUGCUAUCGAUAC__ ...((..((((((((((((((..((((.....))))..((((((((....).)))))))))))))).(((((....((.....)).....)))))....))))))).))........... (-26.70 = -27.02 + 0.32)

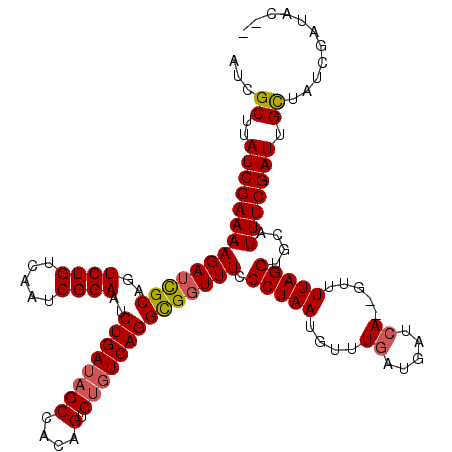
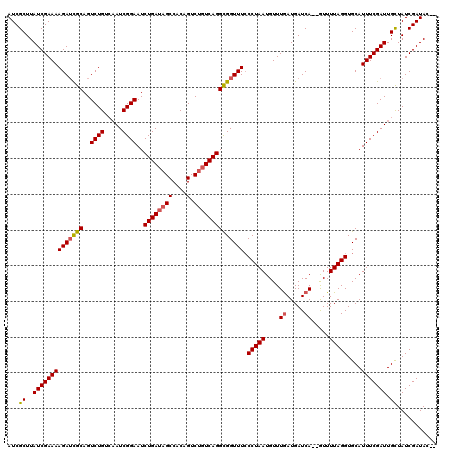
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:14:03 2006