
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 10,590,263 – 10,590,375 |
| Length | 112 |
| Max. P | 0.973469 |

| Location | 10,590,263 – 10,590,375 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.17 |
| Mean single sequence MFE | -33.20 |
| Consensus MFE | -22.00 |
| Energy contribution | -24.28 |
| Covariance contribution | 2.28 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.55 |
| Structure conservation index | 0.66 |
| SVM decision value | 1.71 |
| SVM RNA-class probability | 0.973469 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
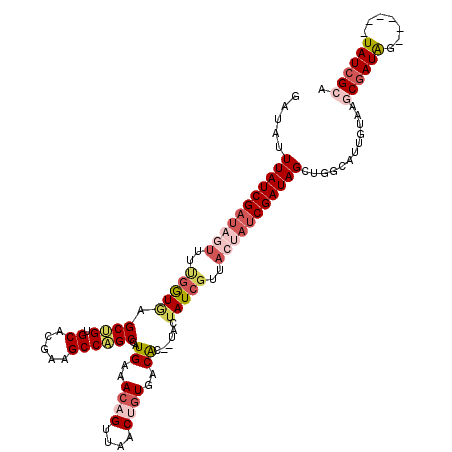
>X_DroMel_CAF1 10590263 112 + 22224390 GAUAUUUAUCGAUAGUCUCGGUGAGCUGUGCACGAAGCCAGCCAUGAAACAGUUAACUGUGACAC--UACUAUCGUUACUAUCGAUAGCUGGCAUUGUAAGCAAUAG------UAUCGCA ((((((((((((((((..(((((.((((.((.....))))))..((..((((....))))..)).--...)))))..))))))))))(((.((...)).)))...))------))))... ( -35.70) >DroSec_CAF1 29934 112 + 1 GAUAUUUAUCGAUAGUUUUGAUGAGCUGUGCACGAAGCCAGCCAUGAAACAGUUAACUGUGACAC--UACUAUCGUUACUAUCGAUAGCUAGAGUAGUAAGCGAUAG------UAUCGCA .....(((((((((((..(((((.((((.((.....))))))..((..((((....))))..)).--...)))))..)))))))))))............(((((..------.))))). ( -34.40) >DroSim_CAF1 31805 112 + 1 GAUAUUUAUCGAUAGUUUUGAUUAGCUGUGCACGAAGCCAGCCAUGAAACAGUUAACUGUGACAC--UACUAUCGUUACUAUCGAUAGCUUGAAUAGUAAGCGAUAG------UAUCGCA ((((((((((((((((..((((..((((.((.....))))))..((..((((....))))..)).--....))))..))))))))))(((((.....)))))...))------))))... ( -35.20) >DroEre_CAF1 29885 103 + 1 CAUAUUUAUCGAACAUUUUGGUGAGCGGUGCACGAAGCCUGCUAUGAAACAGCUACCAGUCACGCGUUACUAU---------CGAUAGCUGACAUUGUAAGCGAUGG------UAUCG-- ((((.(((((((.....)))))))((((.((.....)))))))))).....(.((((.((((.((........---------.....))))))((((....))))))------)).).-- ( -22.72) >DroYak_CAF1 32207 120 + 1 GAUAUUUAUCGAUACUUUUGGUAUGCGGUGCACGAAGCCAGCUAUGAAACAGUUAACACUGACGCGGUACUAUCGUAACAAUCGAUAGCUGACAUUGUAAGCGAUAGUAGAAGUAUCGAU .......(((((((((((((.(((.((.(..((((.(.(((((((((..((((....))))..(((((...))))).....)).))))))).).)))).).))))).))))))))))))) ( -38.00) >consensus GAUAUUUAUCGAUAGUUUUGGUGAGCUGUGCACGAAGCCAGCCAUGAAACAGUUAACUGUGACAC__UACUAUCGUUACUAUCGAUAGCUGGCAUUGUAAGCGAUAG______UAUCGCA .....(((((((((((..(((((.((((.((.....))))))..((..((((....))))..))......)))))..)))))))))))............((((((.......)))))). (-22.00 = -24.28 + 2.28)

| Location | 10,590,263 – 10,590,375 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.17 |
| Mean single sequence MFE | -29.89 |
| Consensus MFE | -18.16 |
| Energy contribution | -20.52 |
| Covariance contribution | 2.36 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.34 |
| Structure conservation index | 0.61 |
| SVM decision value | 1.11 |
| SVM RNA-class probability | 0.916863 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10590263 112 - 22224390 UGCGAUA------CUAUUGCUUACAAUGCCAGCUAUCGAUAGUAACGAUAGUA--GUGUCACAGUUAACUGUUUCAUGGCUGGCUUCGUGCACAGCUCACCGAGACUAUCGAUAAAUAUC .((....------.(((((....)))))...))((((((((((..((......--(((..((((....))))..)))(((((((.....)).)))))...))..))))))))))...... ( -31.50) >DroSec_CAF1 29934 112 - 1 UGCGAUA------CUAUCGCUUACUACUCUAGCUAUCGAUAGUAACGAUAGUA--GUGUCACAGUUAACUGUUUCAUGGCUGGCUUCGUGCACAGCUCAUCAAAACUAUCGAUAAAUAUC .(((((.------..))))).............((((((((((...(((....--(((..((((....))))..)))(((((((.....)).))))).)))...))))))))))...... ( -32.10) >DroSim_CAF1 31805 112 - 1 UGCGAUA------CUAUCGCUUACUAUUCAAGCUAUCGAUAGUAACGAUAGUA--GUGUCACAGUUAACUGUUUCAUGGCUGGCUUCGUGCACAGCUAAUCAAAACUAUCGAUAAAUAUC .(((((.------..))))).............((((((((((...(((....--(((..((((....))))..)))(((((((.....)).))))).)))...))))))))))...... ( -32.70) >DroEre_CAF1 29885 103 - 1 --CGAUA------CCAUCGCUUACAAUGUCAGCUAUCG---------AUAGUAACGCGUGACUGGUAGCUGUUUCAUAGCAGGCUUCGUGCACCGCUCACCAAAAUGUUCGAUAAAUAUG --.....------.....(((.((...)).)))(((((---------(.......(.((((.((((..(((((....)))))((.....)))))).))))).......))))))...... ( -21.74) >DroYak_CAF1 32207 120 - 1 AUCGAUACUUCUACUAUCGCUUACAAUGUCAGCUAUCGAUUGUUACGAUAGUACCGCGUCAGUGUUAACUGUUUCAUAGCUGGCUUCGUGCACCGCAUACCAAAAGUAUCGAUAAAUAUC ((((((((((....(((((..(((...(((((((((.((.......((((((..(((....)))...)))))))))))))))))...)))...)).)))....))))))))))....... ( -31.41) >consensus UGCGAUA______CUAUCGCUUACAAUGCCAGCUAUCGAUAGUAACGAUAGUA__GUGUCACAGUUAACUGUUUCAUGGCUGGCUUCGUGCACAGCUCACCAAAACUAUCGAUAAAUAUC .((((((.......)))))).............((((((((((...((((((...............))))))....(((((((.....)).))))).......))))))))))...... (-18.16 = -20.52 + 2.36)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:13:53 2006