
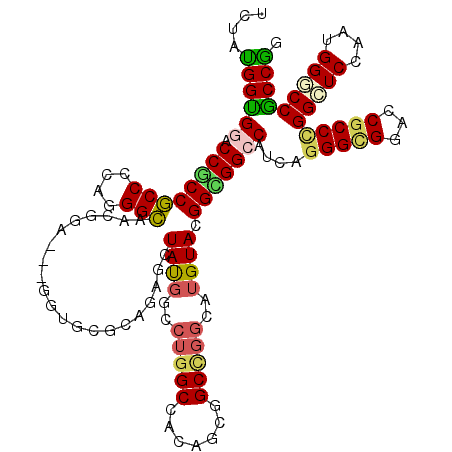
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 10,310,960 – 10,311,157 |
| Length | 197 |
| Max. P | 0.742037 |

| Location | 10,310,960 – 10,311,077 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.72 |
| Mean single sequence MFE | -60.62 |
| Consensus MFE | -36.20 |
| Energy contribution | -36.08 |
| Covariance contribution | -0.12 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.663198 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10310960 117 - 22224390 UCUACGGUGGUCCGCCGCCGCAGGGCAAUGGU---GGAGCGCAGAGCUAUGGACUGGCCACCGCGGCCGGCAUGUAUGGCGGCCAUCAGGGCGGACCGCCCGGCUCCAAUGGGCCGCCGG .....((((((((((((((....)))..((((---((..(((...((.(((..((((((.....)))))))))))...))).)))))).)))))))))))((((((....)))))).... ( -64.00) >DroVir_CAF1 54362 120 - 1 ACUAUGGCGGACCGCCACCCCAGGGUGCCGGCGGUGGUGGCCAAAACUAUGGCCUGGCCACCGCGGCUGGCAUGUACGGCGGCCAUCAAGGUGGCCCGCCGGGUUCGAAUGGACCGCCGG ....((((((.(((..((((..(.((((((((.((((((((((...........)))))))))).)))))))).)..(((((((((....)))).))))))))).....))).)))))). ( -72.30) >DroPse_CAF1 51825 117 - 1 UCUAUGGUGGACCCCCGCCGCAGGGCAACGGA---GGUGCGCAGAGCUACGGCCUGGCCACAGCCGCCGGCAUGUACGGGGGCCAUCAGGGCGGACCGCCCGGCUCCAAUGGGCCGCCAG ....((((((.((((((..(((..((..(((.---.((((.(((.((....)))))).)))..)))...)).))).))))))))))))(((((...)))))(((((....)))))..... ( -61.00) >DroGri_CAF1 49285 120 - 1 UCUAUGGUGGCCCGCCGCCCCAAGGCGGCGGUGGCGGUGCCCAGAAUUAUGGCCUCGCCUCGGCGGCCGGCAUGUAUGGUGGGCAUCAGGGAGGUCCGCCUGGCUCGAAUGGACCACCGG ....(((((((((((((((....)))))))..(((((((((((..((....(((((((....))))..)))....))..))))))))..((....)))))..........)).)))))). ( -60.00) >DroAna_CAF1 49858 99 - 1 UCUAUGGCGGACCACCGCCCCAAGGCAAUGGC---GGAGCGCAGAGCUAUGGGCU------------------GUAUGGCGGGCAUCAGGGCGGACCGCCCGGAUCCAAUGGGCCGCCGG ....((((((.((((((((....(((.(((((---..........)))))..)))------------------....)))))(.(((.(((((...))))).))).)..))).)))))). ( -45.40) >DroPer_CAF1 53504 117 - 1 UCUAUGGUGGACCCCCGCCGCAGGGCAACGGA---GGUGCGCAGAGCUACGGCCUGGCCACAGCCGCCGGCAUGUACGGGGGCCAUCAGGGCGGACCGCCCGGCUCCAAUGGGCCGCCAG ....((((((.((((((..(((..((..(((.---.((((.(((.((....)))))).)))..)))...)).))).))))))))))))(((((...)))))(((((....)))))..... ( -61.00) >consensus UCUAUGGUGGACCGCCGCCCCAGGGCAACGGA___GGUGCGCAGAGCUAUGGCCUGGCCACAGCGGCCGGCAUGUACGGCGGCCAUCAGGGCGGACCGCCCGGCUCCAAUGGGCCGCCGG ....((((((.((((((((....))).....................((((..(((((.......)))))..)))).)))))))....(((((...)))))(((((....))))))))). (-36.20 = -36.08 + -0.12)


| Location | 10,311,040 – 10,311,157 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.90 |
| Mean single sequence MFE | -58.45 |
| Consensus MFE | -36.25 |
| Energy contribution | -37.87 |
| Covariance contribution | 1.61 |
| Combinations/Pair | 1.27 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.14 |
| SVM RNA-class probability | 0.604353 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10311040 117 + 22224390 GCUCC---ACCAUUGCCCUGCGGCGGCGGACCACCGUAGAACUGUGGUCCGCUCGGACCGCCUGGCGCCGAAUUCGAGUAGAAACUGUUCGACUGCAGCUGAUGGGCACCCGGCGGUGGU ...((---(((...(((..(((((((((((((((.(.....).))))))))).))..))))..)))((((...((((((((...)))))))).....(((....)))...))))))))). ( -55.50) >DroVir_CAF1 54442 120 + 1 CCACCACCGCCGGCACCCUGGGGUGGCGGUCCGCCAUAGUACUGGGGACCGCUGGGGCCGGCGGGCGCUGAAUUCGAGUAGUAGCUGUUCGACUGCAGCUGAUGGGCGCCUGGUGGAUGU ((((((.(((((((.(((......((((((((.(((......)))))))))))))))))))))(((((((....)......(((((((......)))))))...)))))))))))).... ( -68.60) >DroGri_CAF1 49365 120 + 1 GCACCGCCACCGCCGCCUUGGGGCGGCGGGCCACCAUAGAACUGGGGACCGCUGGGUCCGCCGGGUGCCGAAUUCGAGUAGUAACUGUUCGACUGCAACUGAUGUGCGCCGGGCGGUGGU .(((((((..(((((((....)))))))((((((..(((..(((((((((....))))).)))).(((.(...((((((((...))))))))).))).)))..))).))).))))))).. ( -62.00) >DroMoj_CAF1 66565 117 + 1 GCCGCGCCCCCAGCGCCCUGAGGUGGC---CCGCCAUAGUACUGUGGACCGCUCGGACCUCCGGGUGCGGAAUUCGAGUAGUAGCUGUUCGACUGCAGCUGAUGGGCACCAGGCGGCGGU (((((((((((.((((((.((((((((---...(((((....)))))...)))...))))).))))))))...........(((((((......)))))))..)))).....)))))... ( -56.10) >DroAna_CAF1 49920 117 + 1 GCUCC---GCCAUUGCCUUGGGGCGGUGGUCCGCCAUAGAACUGGGGUCCACUGGGUCCGCCCGGCGCCGAAUUCGAGUAAUAGCUGUUCGAUUGCAGCUGAUGGGCGCCCGGCGGCGGU .....---(((((((((....)))))))))(((((.........((..((...))..))(((.(((((((....)......(((((((......)))))))...)))))).)))))))). ( -60.10) >DroPer_CAF1 53584 117 + 1 GCACC---UCCGUUGCCCUGCGGCGGGGGUCCACCAUAGAACUGAGGGCCACUGGGACCGCCUGGCGCCGAAUUCGAGUAGUAGCUGUUGGACUGGAGCUGAUGGGCGACCGGCGGGGGG ..(((---((((((((...)))))))))))((.((......((.((.....)).)).(((((.(((((((....)......(((((.(......).)))))...)))).))))))))))) ( -48.40) >consensus GCACC___ACCAUCGCCCUGGGGCGGCGGUCCACCAUAGAACUGGGGACCGCUGGGACCGCCGGGCGCCGAAUUCGAGUAGUAGCUGUUCGACUGCAGCUGAUGGGCGCCCGGCGGCGGU .........((.(((((....))))).)).(((((..........((.((....)).))(((.(((((((....)......(((((((......)))))))...)))))).)))))))). (-36.25 = -37.87 + 1.61)

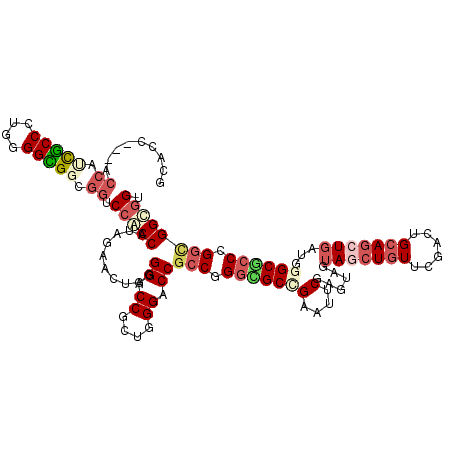

| Location | 10,311,040 – 10,311,157 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.90 |
| Mean single sequence MFE | -54.38 |
| Consensus MFE | -30.33 |
| Energy contribution | -31.67 |
| Covariance contribution | 1.34 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.10 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.742037 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10311040 117 - 22224390 ACCACCGCCGGGUGCCCAUCAGCUGCAGUCGAACAGUUUCUACUCGAAUUCGGCGCCAGGCGGUCCGAGCGGACCACAGUUCUACGGUGGUCCGCCGCCGCAGGGCAAUGGU---GGAGC .((((((((((((.......(((((........))))).........)))))))(((..((((..((.(((((((((.(.....).)))))))))))))))..)))...)))---))... ( -57.39) >DroVir_CAF1 54442 120 - 1 ACAUCCACCAGGCGCCCAUCAGCUGCAGUCGAACAGCUACUACUCGAAUUCAGCGCCCGCCGGCCCCAGCGGUCCCCAGUACUAUGGCGGACCGCCACCCCAGGGUGCCGGCGGUGGUGG ....(((((((((((.....(((((........))))).......(....).)))))((((((((((.((((((((((......))).))))))).......))).))))))).)))))) ( -61.30) >DroGri_CAF1 49365 120 - 1 ACCACCGCCCGGCGCACAUCAGUUGCAGUCGAACAGUUACUACUCGAAUUCGGCACCCGGCGGACCCAGCGGUCCCCAGUUCUAUGGUGGCCCGCCGCCCCAAGGCGGCGGUGGCGGUGC ..(((((((....((.(((((...((.((((((.(((....)))....))))))....((.(((((....))))))).))....)))))))((((((((....)))))))).))))))). ( -57.70) >DroMoj_CAF1 66565 117 - 1 ACCGCCGCCUGGUGCCCAUCAGCUGCAGUCGAACAGCUACUACUCGAAUUCCGCACCCGGAGGUCCGAGCGGUCCACAGUACUAUGGCGG---GCCACCUCAGGGCGCUGGGGGCGCGGC .(((((.((..((((((...(((((........)))))....((((..(((((....)))))...)))).(((((.((......))..))---)))......))))))..)).).)))). ( -54.70) >DroAna_CAF1 49920 117 - 1 ACCGCCGCCGGGCGCCCAUCAGCUGCAAUCGAACAGCUAUUACUCGAAUUCGGCGCCGGGCGGACCCAGUGGACCCCAGUUCUAUGGCGGACCACCGCCCCAAGGCAAUGGC---GGAGC .((((((((.((((((....(((((........))))).......(....)))))))((((((......(((.(((((......))).)).)))))))))...)))...)))---))... ( -53.40) >DroPer_CAF1 53584 117 - 1 CCCCCCGCCGGUCGCCCAUCAGCUCCAGUCCAACAGCUACUACUCGAAUUCGGCGCCAGGCGGUCCCAGUGGCCCUCAGUUCUAUGGUGGACCCCCGCCGCAGGGCAACGGA---GGUGC ....(((((((.((((....((((..........)))).......(....))))))).)))))........((((((.(((((.((((((....)))))).)))))....))---)).)) ( -41.80) >consensus ACCACCGCCGGGCGCCCAUCAGCUGCAGUCGAACAGCUACUACUCGAAUUCGGCGCCCGGCGGUCCCAGCGGUCCCCAGUUCUAUGGCGGACCGCCGCCCCAGGGCAACGGC___GGUGC ....(((((.(((((.....(((((........)))))......((....))))))).))))).((..((((.(((((......))).)).)))).(((....)))...))......... (-30.33 = -31.67 + 1.34)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:10:40 2006