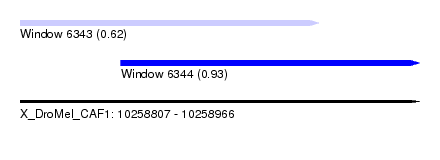
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 10,258,807 – 10,258,966 |
| Length | 159 |
| Max. P | 0.930093 |

| Location | 10,258,807 – 10,258,926 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 83.81 |
| Mean single sequence MFE | -53.28 |
| Consensus MFE | -38.12 |
| Energy contribution | -38.40 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.06 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.621571 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10258807 119 + 22224390 AUGGCCGAAGCAGCAAUGGCGCCGGUGAUCUCCUCCGGCUCGGUGCUCCGCUGCAAAGUGUCCUCCAGGGCGAUCCAGUUCUUGGUGGGACCGCGUUGCGGACGGAGCGUCACCGUGGU ((((((((....((....))(((((.(.....).))))))))).((((((((((((.((((((((((((((......).)))))).)))).))).)))))).))))))....))))... ( -55.10) >DroVir_CAF1 44762 119 + 1 AUGGCCGAAGCGGCAAUAGCUCCAGUUAUCUCAUCCGGCUCAGUGCUGCGCUGCAGUGUAUCCUCCAAUGCUAUCCAGUUCUUGGUGGGUCCGCGCUGCGGGCGCAGCGCCACGGUGGU ((.((((.(((((..(((((....))))).....)).)))..(((((((((((((((((((((.((((.(((....)))..)))).))))..))))))).))))))))))..)))).)) ( -51.50) >DroGri_CAF1 36810 119 + 1 AUAGCCGAUGCGGCGAUGACGCCAGUUAUCUCAUCCGGCUCGGUGCUGCGCUGCAGUGUAUCCUCCAGAGCUAUCCAGUUCUUGGUAGGUCCACGCUGCGGACGCAGCGCCACAGUCGU ...((((...))))((((((....)))))).....(((((.((((((((((((((((((..(((((((((((....))))).))).)))...))))))))).)))))))))..))))). ( -62.90) >DroWil_CAF1 30268 119 + 1 AAUGCCGAAGCAGCUAUCGCUCCAGUUAUCUCAUCCGGCUCGGUGCUGCGCUGCAAUGUAUCCUCCAAUGCAAUCCAGUUUUUGGUCGGCCCACGUUGCGGACGCAGCACCACCAUGGU ...((((..(.(((....))).).(......)...))))..((((((((((((((((((..((.((((.((......))..))))..))...))))))))).)))))))))........ ( -45.50) >DroMoj_CAF1 39711 119 + 1 AUGGCCGAGGCGGCAAUGGCGCCAGUUAUCUCAUCCGGCUCGGUGCUGCGCUGCAGCGUAUCCUCCAGUGCGAUCCAGUUCUUGGUGGGGCCGCGCUGAGGUCGCAGCGCUACGGUGGU ((.((((((.(((..(((((....))))).....))).)))(((((((((((.((((((.(((.((((.((......))..)))).)))...)))))).)).)))))))))..))).)) ( -53.30) >DroAna_CAF1 31820 119 + 1 AUGGCCGAGGCGGCAAUGGCGCCGGUGAUCUCUUCCGGUUCGGUGCUACGCUGCAAGGUGUCCUCCAAAGCGAUCCAGUUCUUGGUUGGGCCCCUCUGGGGACGGAGAGCCACGGUGGU ((.((((..(((((..((((((((..((((......)))))))))))).)))))..(((((((.(((((((......))).))))..))))..(((((....))))).))).)))).)) ( -51.40) >consensus AUGGCCGAAGCGGCAAUGGCGCCAGUUAUCUCAUCCGGCUCGGUGCUGCGCUGCAAUGUAUCCUCCAAUGCGAUCCAGUUCUUGGUGGGGCCGCGCUGCGGACGCAGCGCCACGGUGGU ..(((((....(((......)))((....))....))))).((((((((((((((((((..(((((((.((......))..)))).)))...))))))))).)))))))))........ (-38.12 = -38.40 + 0.28)
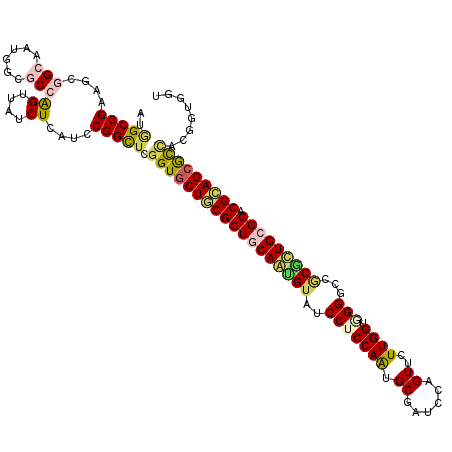

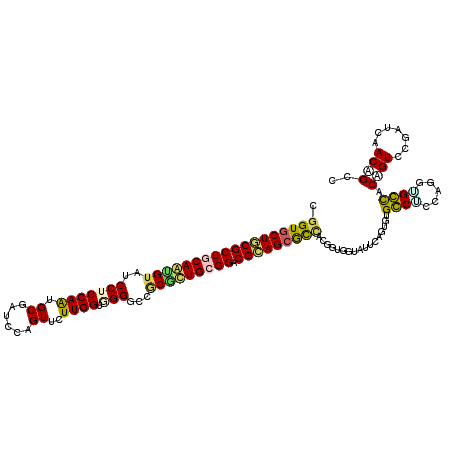
| Location | 10,258,847 – 10,258,966 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 78.04 |
| Mean single sequence MFE | -54.50 |
| Consensus MFE | -37.33 |
| Energy contribution | -37.00 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.49 |
| Mean z-score | -1.89 |
| Structure conservation index | 0.68 |
| SVM decision value | 1.20 |
| SVM RNA-class probability | 0.930093 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10258847 119 + 22224390 CGGUGCUCCGCUGCAAAGUGUCCUCCAGGGCGAUCCAGUUCUUGGUGGGACCGCGUUGCGGACGGAGCGUCACCGUGGUGUUCAACGAGGCUCCAGCGGCCACUGUGCGAUCCACCGGC .(..((((((((((((.((((((((((((((......).)))))).)))).))).)))))).))))))..).((((((...((.(((.((((.....))))..)))..)).))).))). ( -52.60) >DroVir_CAF1 44802 119 + 1 CAGUGCUGCGCUGCAGUGUAUCCUCCAAUGCUAUCCAGUUCUUGGUGGGUCCGCGCUGCGGGCGCAGCGCCACGGUGGUAUUCAGAGUGCCGCCAGAUGGCACUGUCCGGUCCACAGCC (((((((((((((((((((((((.((((.(((....)))..)))).))))..))))))).))))))))(((((((((((((.....)))))))).).))))))))...(((.....))) ( -59.10) >DroGri_CAF1 36850 119 + 1 CGGUGCUGCGCUGCAGUGUAUCCUCCAGAGCUAUCCAGUUCUUGGUAGGUCCACGCUGCGGACGCAGCGCCACAGUCGUAUUGAGUUGGCCACCAGGUGGCACGGUCCGAUCAACCGGC .((((((((((((((((((..(((((((((((....))))).))).)))...))))))))).)))))))))...((((.((((..((((((((...))))).)))..))))....)))) ( -60.80) >DroWil_CAF1 30308 119 + 1 CGGUGCUGCGCUGCAAUGUAUCCUCCAAUGCAAUCCAGUUUUUGGUCGGCCCACGUUGCGGACGCAGCACCACCAUGGUAUUAAGUGUGGCUCCCGGUGCCACAGUGCGAUCAACUGCU .((((((((((((((((((..((.((((.((......))..))))..))...))))))))).)))))))))....((((....(.((((((.......)))))).)...))))...... ( -51.60) >DroMoj_CAF1 39751 119 + 1 CGGUGCUGCGCUGCAGCGUAUCCUCCAGUGCGAUCCAGUUCUUGGUGGGGCCGCGCUGAGGUCGCAGCGCUACGGUGGUGUUCAGUGUGCCACCCGGCGGUACGGUGCGGUCCACGGCG .((.(((((((((..(((...((((.((((((.((((.(....).))))..)))))))))).)))..((((..(((((..(.....)..))))).))))...)))))))))))...... ( -57.50) >DroAna_CAF1 31860 119 + 1 CGGUGCUACGCUGCAAGGUGUCCUCCAAAGCGAUCCAGUUCUUGGUUGGGCCCCUCUGGGGACGGAGAGCCACGGUGGUGUUCAAGGAGGCUCCGGCAGCCACAGUCCUGUCAACAGGC ..(((....(((((..((.(.(((((...((..(((.(((((..(..(....)..)..))))))))..))(((....))).....)))))).)).))))))))...((((....)))). ( -45.40) >consensus CGGUGCUGCGCUGCAAUGUAUCCUCCAAUGCGAUCCAGUUCUUGGUGGGGCCGCGCUGCGGACGCAGCGCCACGGUGGUAUUCAGUGUGCCUCCAGGUGCCACAGUCCGAUCAACAGCC .((((((((((((((((((..(((((((.((......))..)))).)))...))))))))).))))))))).................((((.....)))).((((.......)))).. (-37.33 = -37.00 + -0.33)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:10:23 2006