
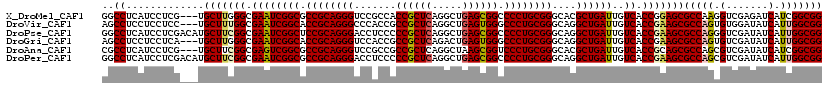
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 10,084,580 – 10,084,697 |
| Length | 117 |
| Max. P | 0.778732 |

| Location | 10,084,580 – 10,084,697 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 89.11 |
| Mean single sequence MFE | -61.32 |
| Consensus MFE | -48.92 |
| Energy contribution | -49.23 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.63 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.778732 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10084580 117 + 22224390 GGCCUCAUCCUCG---UGCUUGGGCGAAUCGGCGCCGCAGGGUCCGCCACCGCUCAGGCUGAGCGGCCCCUGCGGGCACGCUGAUUGUCACCGGAGCGCCAAGGUCGAGAUCAUCGGCGG (..(((..(((.(---(((((.((.(((((((((((((((((.((((((((.....)).)).)))).))))))))...)))))))..)).)).))))))..)))..)))..)........ ( -62.30) >DroVir_CAF1 12727 117 + 1 AGCCUCCUCCUCC---UGCUUUGGCGAAUCGGCACCGCAGGGCCCACCGCCGCUCAGGCUGAGUGGGCCCUGCGGGCAGGCUGAUUGUCACCGAAGCGCCAGUGUGGAUAUCAUUGGCGG ..((.........---.(((((((.((((((((.(((((((((((((((((.....))).).)))))))))))))....))))))..)).)))))))(((((((.......))))))))) ( -68.90) >DroPse_CAF1 956 120 + 1 GGCCUCAUCCUCGACAUGCUUCGGCGAAUCGGCUCCGCAGGGACCUCCCCCGCUCAGGCUGAGCGGCCCCUGCGGGCAGGCUGAUUGUCACCGAAGCGCCAGGGUCGAUAUCAUUGGCGG .(((......(((((.((((((((.(((((((((((((((((.......((((((.....)))))).))))))))...)))))))..)).)))))))).....))))).......))).. ( -59.02) >DroGri_CAF1 1045 117 + 1 AGCCUCCUCCUCA---UGCUUGGGCGAAUCGGCACCGCAGGGUCCACCGCCGCUCAGACUGAGUGGGCCCUGCGGGCAGGCUGAUUGUCACCGAAGCGCCAGUGUCGAUAUCAUUGGCGG ..((.........---.((((.((.((((((((.(((((((((((((((..........)).)))))))))))))....))))))..)).)).))))(((((((.......))))))))) ( -57.70) >DroAna_CAF1 871 117 + 1 CGCCUCAUCCUCG---UGCUUCGGCGAGUCGGCGCCGCAGGGUCCGCCGCCGCUCAGGCUAAGCGGUCCCUGCGGGCACGCUGAUUGUCACCGCAGCGCCAGCGUCGAUAUCAUCGGCGG .........((.(---((((.(((.(((((((((((((((((.((((.(((.....)))...)))).))))))))...)))))))..)).))).))))).))(((((((...))))))). ( -62.50) >DroPer_CAF1 968 120 + 1 GGCCUCAUCCUCGACAUGCUUCGGCGAAUCGGCGCCGCAGGGACCUCCCCCGCUCAGGCUGAGCGGCCCCUGCGGGCAGGCUGAUUGUCACCGAAGCGCCAGCGUCGAUAUCAUUGGCGG .(((......(((((..(((((((.(((((((((((((((((.......((((((.....)))))).)))))).)))..))))))..)).)))))))(....)))))).......))).. ( -57.52) >consensus GGCCUCAUCCUCG___UGCUUCGGCGAAUCGGCGCCGCAGGGUCCACCGCCGCUCAGGCUGAGCGGCCCCUGCGGGCAGGCUGAUUGUCACCGAAGCGCCAGCGUCGAUAUCAUUGGCGG ..((.............(((((((.((((((((.((((((((.......((((((.....)))))).))))))))....))))))..)).)))))))(((((.(.......).))))))) (-48.92 = -49.23 + 0.31)

| Location | 10,084,580 – 10,084,697 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.11 |
| Mean single sequence MFE | -58.20 |
| Consensus MFE | -47.44 |
| Energy contribution | -48.17 |
| Covariance contribution | 0.73 |
| Combinations/Pair | 1.13 |
| Mean z-score | -1.83 |
| Structure conservation index | 0.82 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.514850 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 10084580 117 - 22224390 CCGCCGAUGAUCUCGACCUUGGCGCUCCGGUGACAAUCAGCGUGCCCGCAGGGGCCGCUCAGCCUGAGCGGUGGCGGACCCUGCGGCGCCGAUUCGCCCAAGCA---CGAGGAUGAGGCC ..........(((((.(((((..(((..(((((..(((.(((...((((((((.(((((..(((.....)))))))).))))))))))).))))))))..))).---))))).))))).. ( -57.80) >DroVir_CAF1 12727 117 - 1 CCGCCAAUGAUAUCCACACUGGCGCUUCGGUGACAAUCAGCCUGCCCGCAGGGCCCACUCAGCCUGAGCGGCGGUGGGCCCUGCGGUGCCGAUUCGCCAAAGCA---GGAGGAGGAGGCU ..((((.((.......)).))))((((((....)..((..(((((..((((((((((((..(((.....)))))))))))))))((((......))))...)))---))..)).))))). ( -62.40) >DroPse_CAF1 956 120 - 1 CCGCCAAUGAUAUCGACCCUGGCGCUUCGGUGACAAUCAGCCUGCCCGCAGGGGCCGCUCAGCCUGAGCGGGGGAGGUCCCUGCGGAGCCGAUUCGCCGAAGCAUGUCGAGGAUGAGGCC ..(((.......(((.((.((((((((((((((..(((.((....(((((((((((.(((.((....))..))).))))))))))).)).)))))))))))))..)))).)).)))))). ( -59.71) >DroGri_CAF1 1045 117 - 1 CCGCCAAUGAUAUCGACACUGGCGCUUCGGUGACAAUCAGCCUGCCCGCAGGGCCCACUCAGUCUGAGCGGCGGUGGACCCUGCGGUGCCGAUUCGCCCAAGCA---UGAGGAGGAGGCU ((((((.((.......)).))))((((.(((((..(((.((....((((((((.(((((..(((.....)))))))).)))))))).)).)))))))).)))).---...))........ ( -50.10) >DroAna_CAF1 871 117 - 1 CCGCCGAUGAUAUCGACGCUGGCGCUGCGGUGACAAUCAGCGUGCCCGCAGGGACCGCUUAGCCUGAGCGGCGGCGGACCCUGCGGCGCCGACUCGCCGAAGCA---CGAGGAUGAGGCG .(((((((...)))....((.(.(((.((((((...((.(((...((((((((.(((((..(((.....)))))))).))))))))))).)).)))))).))).---).)).....)))) ( -57.40) >DroPer_CAF1 968 120 - 1 CCGCCAAUGAUAUCGACGCUGGCGCUUCGGUGACAAUCAGCCUGCCCGCAGGGGCCGCUCAGCCUGAGCGGGGGAGGUCCCUGCGGCGCCGAUUCGCCGAAGCAUGUCGAGGAUGAGGCC ..(((.......((((((....)((((((((((..(((.((..(((.(((((((((.(((.((....))..))).)))))))))))))).)))))))))))))..)))))......))). ( -61.82) >consensus CCGCCAAUGAUAUCGACACUGGCGCUUCGGUGACAAUCAGCCUGCCCGCAGGGGCCGCUCAGCCUGAGCGGCGGAGGACCCUGCGGCGCCGAUUCGCCGAAGCA___CGAGGAUGAGGCC ((((((.((.......)).))))((((.(((((..(((.((....((((((((.(((((..(((.....)))))))).)))))))).)).)))))))).)))).......))........ (-47.44 = -48.17 + 0.73)

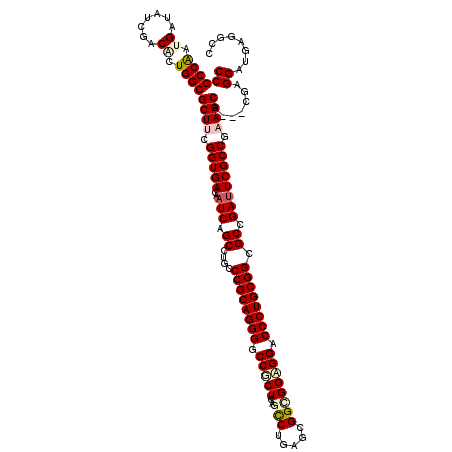

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:08:54 2006