
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 9,453,533 – 9,453,666 |
| Length | 133 |
| Max. P | 0.998373 |

| Location | 9,453,533 – 9,453,626 |
|---|---|
| Length | 93 |
| Sequences | 5 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 77.07 |
| Mean single sequence MFE | -30.08 |
| Consensus MFE | -11.67 |
| Energy contribution | -13.56 |
| Covariance contribution | 1.89 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.23 |
| Structure conservation index | 0.39 |
| SVM decision value | 1.16 |
| SVM RNA-class probability | 0.924379 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 9453533 93 + 22224390 UUAGGGUAUUAGCAGACAAUGGAGAUGUUGCCACAAGGCUGUUAUAACCACGCAU---GGAUAUAUAUAUAUAUAUAUCC-------------AGCCUGGCCAACGGUG ...((....((((((....(((........))).....))))))...(((.((.(---(((((((((....)))))))))-------------))).)))))....... ( -26.20) >DroSec_CAF1 2143 90 + 1 UU-GGGUAUUAGCCGACAAUGGAGAUGUUGCCACAAGGCUGUUCUAACCACGCAU---GGAUAUAUAU--GCAUAUAUCC-------------AGCCUGGCCAACGGUG ..-........((((....((((((....(((....)))...)))..(((.((.(---((((((((..--..))))))))-------------))).)))))).)))). ( -27.80) >DroSim_CAF1 1835 90 + 1 UU-GGGUAUUAGCCGACAAUGGAGAUGUUGCCACAAGGCUGUUCUAACCACGCAU---GGAUAUAUAU--GCAUAUAUCC-------------AGCCUGGCCAACGGUG ..-........((((....((((((....(((....)))...)))..(((.((.(---((((((((..--..))))))))-------------))).)))))).)))). ( -27.80) >DroEre_CAF1 1856 107 + 1 UU-GGGAAUUAGCAGGCAAUGGAGGUUCUACCACAAGGCUGUUAUAACCACGGAUUAUGUAUGUAUA-AUACAUGUAUCCAUGCAGUUGUGACAGCCUGGCCAACGGUG ..-...........(((......((.....))...(((((((((((((((.(((((((((((.....-))))))).)))).))..))))))))))))).)))....... ( -37.80) >DroYak_CAF1 1865 98 + 1 UU-GGGUAAUAGCAGGCAAUGGAGGUAUUACCACAAGGCUGUUAUAACCACGGAUUAUAUACGAA----------UAUCCAGGCAGUUGUGACAGCCUGGCCAACGGUG .(-((((((((.(..........).))))))).))(((((((((((((..(((((..........----------.)))).)...))))))))))))).(((...))). ( -30.80) >consensus UU_GGGUAUUAGCAGACAAUGGAGAUGUUGCCACAAGGCUGUUAUAACCACGCAU___GGAUAUAUAU__ACAUAUAUCC_____________AGCCUGGCCAACGGUG ....(((..((((((.(..(((........)))...).))))))..))).........((((((((......))))))))..............(((........))). (-11.67 = -13.56 + 1.89)


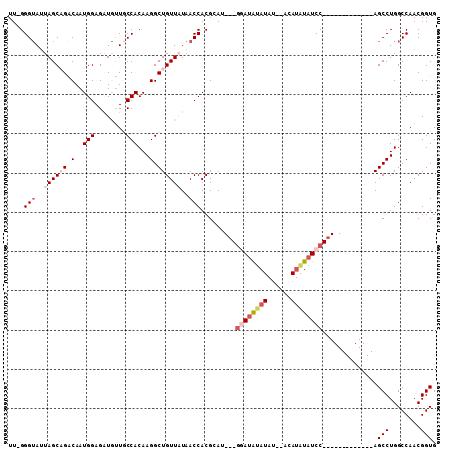
| Location | 9,453,533 – 9,453,626 |
|---|---|
| Length | 93 |
| Sequences | 5 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 77.07 |
| Mean single sequence MFE | -28.68 |
| Consensus MFE | -17.82 |
| Energy contribution | -19.00 |
| Covariance contribution | 1.18 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.87 |
| Structure conservation index | 0.62 |
| SVM decision value | 3.08 |
| SVM RNA-class probability | 0.998373 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 9453533 93 - 22224390 CACCGUUGGCCAGGCU-------------GGAUAUAUAUAUAUAUAUAUCC---AUGCGUGGUUAUAACAGCCUUGUGGCAACAUCUCCAUUGUCUGCUAAUACCCUAA ....((((((((.(((-------------(((((((((....)))))))))---).)).))))..)))).......((((((((.......))).)))))......... ( -28.30) >DroSec_CAF1 2143 90 - 1 CACCGUUGGCCAGGCU-------------GGAUAUAUGC--AUAUAUAUCC---AUGCGUGGUUAGAACAGCCUUGUGGCAACAUCUCCAUUGUCGGCUAAUACCC-AA .....(((((((.(((-------------((((((((..--..))))))))---).)).)))))))...((((..((((........))))....)))).......-.. ( -29.50) >DroSim_CAF1 1835 90 - 1 CACCGUUGGCCAGGCU-------------GGAUAUAUGC--AUAUAUAUCC---AUGCGUGGUUAGAACAGCCUUGUGGCAACAUCUCCAUUGUCGGCUAAUACCC-AA .....(((((((.(((-------------((((((((..--..))))))))---).)).)))))))...((((..((((........))))....)))).......-.. ( -29.50) >DroEre_CAF1 1856 107 - 1 CACCGUUGGCCAGGCUGUCACAACUGCAUGGAUACAUGUAU-UAUACAUACAUAAUCCGUGGUUAUAACAGCCUUGUGGUAGAACCUCCAUUGCCUGCUAAUUCCC-AA ....((.(((.(((((((.(.(((..(((((((..((((((-.....)))))).)))))))))).).))))))).((((........)))).))).))........-.. ( -30.60) >DroYak_CAF1 1865 98 - 1 CACCGUUGGCCAGGCUGUCACAACUGCCUGGAUA----------UUCGUAUAUAAUCCGUGGUUAUAACAGCCUUGUGGUAAUACCUCCAUUGCCUGCUAUUACCC-AA ....((.(((.(((((((.(.(((..(..(((((----------(......)).)))))..))).).))))))).((((........)))).))).))........-.. ( -25.50) >consensus CACCGUUGGCCAGGCU_____________GGAUAUAUGC__AUAUAUAUCC___AUGCGUGGUUAUAACAGCCUUGUGGCAACAUCUCCAUUGUCUGCUAAUACCC_AA ....((((((.((((..............(((((((((....))))))))).......((((........(((....))).......)))).))))))))))....... (-17.82 = -19.00 + 1.18)

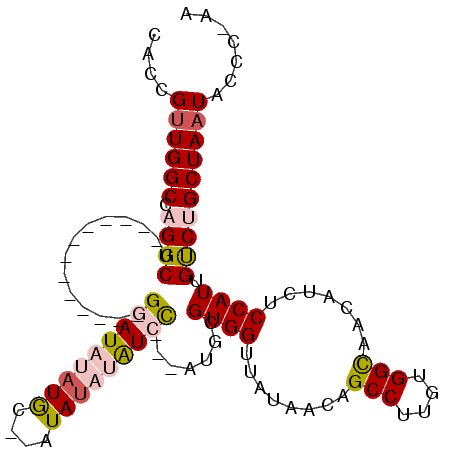
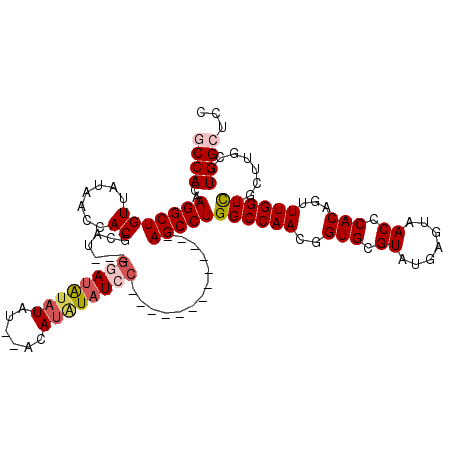
| Location | 9,453,562 – 9,453,666 |
|---|---|
| Length | 104 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.75 |
| Mean single sequence MFE | -35.84 |
| Consensus MFE | -20.03 |
| Energy contribution | -21.16 |
| Covariance contribution | 1.13 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.47 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.97 |
| SVM RNA-class probability | 0.891790 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 9453562 104 + 22224390 GCCACAAGGCUGUUAUAACCACGCAU---GGAUAUAUAUAUAUAUAUAUCC-------------AGCCUGGCCAACGGUGCGUAUUAGUAACCCACAGUUUGGUCUGCUUGCUUGGCUCC ((((....((((((....(((.((.(---(((((((((....)))))))))-------------))).)))..))))))(((....((((..(((.....)))..))))))).))))... ( -33.60) >DroSec_CAF1 2171 102 + 1 GCCACAAGGCUGUUCUAACCACGCAU---GGAUAUAUAU--GCAUAUAUCC-------------AGCCUGGCCAACGGUGCGUAUGAGUAACCCACAGUUUGGUCUGCUUGCUUGGCUCC ((((....((((((....(((.((.(---((((((((..--..))))))))-------------))).)))..))))))((....(((((..(((.....)))..))))))).))))... ( -32.70) >DroSim_CAF1 1863 102 + 1 GCCACAAGGCUGUUCUAACCACGCAU---GGAUAUAUAU--GCAUAUAUCC-------------AGCCUGGCCAACGGUGCGUAUGAGUAACCCACAGUUUGGUCUGCUUGCUUGGCUCC ((((....((((((....(((.((.(---((((((((..--..))))))))-------------))).)))..))))))((....(((((..(((.....)))..))))))).))))... ( -32.70) >DroEre_CAF1 1884 119 + 1 ACCACAAGGCUGUUAUAACCACGGAUUAUGUAUGUAUA-AUACAUGUAUCCAUGCAGUUGUGACAGCCUGGCCAACGGUGCGUAUGAGUAACCCACAGUUUGGUUUGCUUGCUUGGCUGC ......(((((((((((((((.(((((((((((.....-))))))).)))).))..)))))))))))))((((((....((....((((((.(((.....))).)))))))))))))).. ( -44.70) >DroYak_CAF1 1893 110 + 1 ACCACAAGGCUGUUAUAACCACGGAUUAUAUACGAA----------UAUCCAGGCAGUUGUGACAGCCUGGCCAACGGUGCGUAUGAGUAACCCACAGUUUGGUCUGCUUGCUUGGCUGC ......(((((((((((((..(((((..........----------.)))).)...)))))))))))))((((((....((....(((((..(((.....)))..))))))))))))).. ( -35.50) >consensus GCCACAAGGCUGUUAUAACCACGCAU___GGAUAUAUAU__ACAUAUAUCC_____________AGCCUGGCCAACGGUGCGUAUGAGUAACCCACAGUUUGGUCUGCUUGCUUGGCUCC ((((..(((((((.......)).......((((((((......)))))))).............)))))((((((..(((.((.......)).)))...))))))........))))... (-20.03 = -21.16 + 1.13)

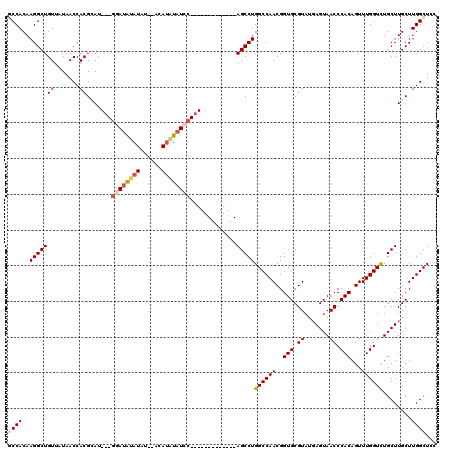
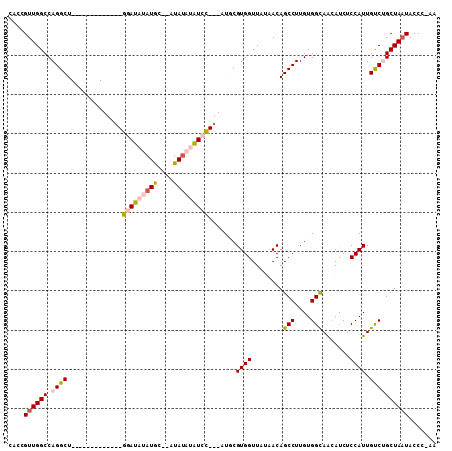
| Location | 9,453,562 – 9,453,666 |
|---|---|
| Length | 104 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.75 |
| Mean single sequence MFE | -34.15 |
| Consensus MFE | -23.76 |
| Energy contribution | -24.98 |
| Covariance contribution | 1.22 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.56 |
| SVM RNA-class probability | 0.782448 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 9453562 104 - 22224390 GGAGCCAAGCAAGCAGACCAAACUGUGGGUUACUAAUACGCACCGUUGGCCAGGCU-------------GGAUAUAUAUAUAUAUAUAUCC---AUGCGUGGUUAUAACAGCCUUGUGGC ...(((..((((((......(((.(((.((.......)).))).)))(((((.(((-------------(((((((((....)))))))))---).)).)))))......).)))))))) ( -35.60) >DroSec_CAF1 2171 102 - 1 GGAGCCAAGCAAGCAGACCAAACUGUGGGUUACUCAUACGCACCGUUGGCCAGGCU-------------GGAUAUAUGC--AUAUAUAUCC---AUGCGUGGUUAGAACAGCCUUGUGGC ...(((((((..((((......))))..)))........((....(((((((.(((-------------((((((((..--..))))))))---).)).)))))))....))....)))) ( -34.50) >DroSim_CAF1 1863 102 - 1 GGAGCCAAGCAAGCAGACCAAACUGUGGGUUACUCAUACGCACCGUUGGCCAGGCU-------------GGAUAUAUGC--AUAUAUAUCC---AUGCGUGGUUAGAACAGCCUUGUGGC ...(((((((..((((......))))..)))........((....(((((((.(((-------------((((((((..--..))))))))---).)).)))))))....))....)))) ( -34.50) >DroEre_CAF1 1884 119 - 1 GCAGCCAAGCAAGCAAACCAAACUGUGGGUUACUCAUACGCACCGUUGGCCAGGCUGUCACAACUGCAUGGAUACAUGUAU-UAUACAUACAUAAUCCGUGGUUAUAACAGCCUUGUGGU ((......))......(((.(((.(((.((.......)).))).))).((.(((((((.(.(((..(((((((..((((((-.....)))))).)))))))))).).))))))).))))) ( -34.30) >DroYak_CAF1 1893 110 - 1 GCAGCCAAGCAAGCAGACCAAACUGUGGGUUACUCAUACGCACCGUUGGCCAGGCUGUCACAACUGCCUGGAUA----------UUCGUAUAUAAUCCGUGGUUAUAACAGCCUUGUGGU ...(((..((((((......(((..(((((((...(((((.........((((((.((....)).))))))...----------..))))).)))))))..)))......).)))))))) ( -31.84) >consensus GGAGCCAAGCAAGCAGACCAAACUGUGGGUUACUCAUACGCACCGUUGGCCAGGCU_____________GGAUAUAUGC__AUAUAUAUCC___AUGCGUGGUUAUAACAGCCUUGUGGC ..(((((.((((((...((((...(((.((.......)).)))..))))....))).............(((((((((....)))))))))....))).)))))......(((....))) (-23.76 = -24.98 + 1.22)

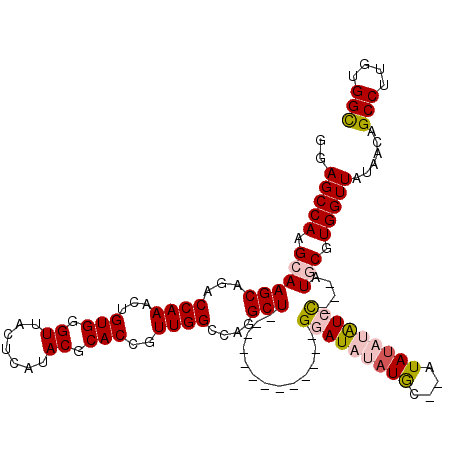

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:01:47 2006