
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 9,450,434 – 9,450,591 |
| Length | 157 |
| Max. P | 0.762394 |

| Location | 9,450,434 – 9,450,551 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.39 |
| Mean single sequence MFE | -41.21 |
| Consensus MFE | -23.36 |
| Energy contribution | -23.20 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.53 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.51 |
| SVM RNA-class probability | 0.762394 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
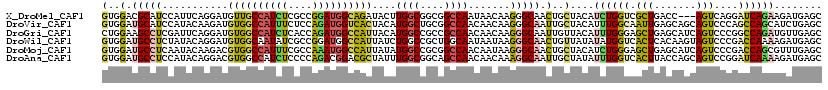
>X_DroMel_CAF1 9450434 117 - 22224390 GUGGACGCAUCCAUUCAGGAUGUUGCCAUCUCGCCGGAUGGCAGAUACUUGGCGGCGGCCAAUAACAAGGGCAACUGCUACAUCUGGUCGCUGACC---AGUCAGGAUCAGAAGAUGAGC (((...((((((.....))))))((((((((....))))))))..))).((((((..(((.........)))..))))))..(((((((.((((..---..)))))))))))........ ( -46.20) >DroVir_CAF1 3169 120 - 1 GUGGAUGCAUCCAUACAAGAUGUGGCCAUUUCUCCAGAUGGUCACUACAUGGCUGCGGCCAACAACAAGGGCAAUUGCUACAUUUGGCAAUUGAGCAGCAGUCCCAGCCAGCAUCUGAGC ..((((((.............(((((((((......)))))))))....((((((.((((........)).(((((((((....))))))))).........)))))))))))))).... ( -43.80) >DroGri_CAF1 3991 120 - 1 CUGGAAGCCUCGAUUCAGGAUGUGGCCAUCUCACCAGAUGGCCAUUACAUGGCCGCCGCCAACAACAAGGGCAAUUGUUACAUUUGGGAGCUGAGCAUCAGUCCCGGCCAGAUGUUGAGC ........((((((((.(...((((((((((....))))))))))..).(((((...(((.........))).............((((.(((.....)))))))))))))).)))))). ( -45.80) >DroWil_CAF1 4471 120 - 1 GUGGAUGCCUCUAUACAGGAUGUGGCAAUAUCGCCGGAUGGCCAUUAUCUGGCCGCUGGCAAUAAUAAGGGCAACUGUUAUAUAUGGUCACUCACAAGUAGUCCCGACCAAAAGAUGAGC ..((.((((..(((....(((((....)))))(((((.((((((.....)))))))))))....)))..)))).)).((((...(((((((((....).)))...)))))....)))).. ( -37.80) >DroMoj_CAF1 3421 120 - 1 GUGGAUGCCUCAAUACAAGACGUGGCCAUUUCGCCAAAUGGCCAUUAUAUGGCCGCGGCCAACAAUAAGGGCAACUGCUACAUCUGGGAGCUGAGCAUCAGUCCCGACCAGCGUUUGAGC ........(((((.((....(((((((((...(((....)))......)))))))))(((.........)))....(((...((.((((.(((.....)))))))))..)))))))))). ( -40.80) >DroAna_CAF1 5413 120 - 1 GUGGAUGCCUCCAUACAGGACGUGGCCAUCUCCCCAGACGGACGCUAUUUGGCGGCAGCCAACAACAAAGGCAAUUGCUAUAUUUGGUCACUUACCAGCAGUCCGGAUCAAAAGAUGAGC (((((....)))))..........(((((((.......((((((((...((((((..(((.........)))..)))))).....(((.....)))))).))))).......))))).)) ( -32.84) >consensus GUGGAUGCCUCCAUACAGGAUGUGGCCAUCUCGCCAGAUGGCCAUUACAUGGCCGCGGCCAACAACAAGGGCAACUGCUACAUUUGGUAACUGACCAGCAGUCCCGACCAGAAGAUGAGC (..(.(((((...........((((((((((....))))))))))....((((....)))).......))))).)..)....((((((.(((.......)))....))))))........ (-23.36 = -23.20 + -0.16)


| Location | 9,450,471 – 9,450,591 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.83 |
| Mean single sequence MFE | -42.63 |
| Consensus MFE | -27.73 |
| Energy contribution | -27.77 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.45 |
| Mean z-score | -1.68 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.47 |
| SVM RNA-class probability | 0.750631 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 9450471 120 - 22224390 GGACGUCAAGUCGGAGCGGCACGAACGCAUCGUGCCCGAGGUGGACGCAUCCAUUCAGGAUGUUGCCAUCUCGCCGGAUGGCAGAUACUUGGCGGCGGCCAAUAACAAGGGCAACUGCUA ....((((..((((...(((((((.....)))))))((((((((..((((((.....))))))..)))))))))))).)))).......((((((..(((.........)))..)))))) ( -54.10) >DroVir_CAF1 3209 120 - 1 GGACGUUAAGUCGGAAGUGCACGAGCAGCUUGUGCCCGAGGUGGAUGCAUCCAUACAAGAUGUGGCCAUUUCUCCAGAUGGUCACUACAUGGCUGCGGCCAACAACAAGGGCAAUUGCUA .(((.....)))......((....))(((...(((((..(.(((.((((.((((.......(((((((((......)))))))))...)))).)))).)))....)..)))))...))). ( -42.40) >DroGri_CAF1 4031 120 - 1 GGAUGUUAAGUCCGAACUGCACGAACAGCUUGUACCCGAACUGGAAGCCUCGAUUCAGGAUGUGGCCAUCUCACCAGAUGGCCAUUACAUGGCCGCCGCCAACAACAAGGGCAAUUGUUA ((((.....))))..........(((((.(((..(((...(((((........)))))...((((((((((....))))))))))....((((....)))).......))))))))))). ( -35.40) >DroWil_CAF1 4511 120 - 1 GGAUGUCAAGUCGGAGAAGCACGAGAGAAUUGUACCCGAAGUGGAUGCCUCUAUACAGGAUGUGGCAAUAUCGCCGGAUGGCCAUUAUCUGGCCGCUGGCAAUAAUAAGGGCAACUGUUA .....(((..((((.(....((((.....)))).)))))..))).((((..(((....(((((....)))))(((((.((((((.....)))))))))))....)))..))))....... ( -38.80) >DroMoj_CAF1 3461 120 - 1 GGACGUGAAGUCAGAGGUGCAUGAGCAGCUGGUGCCCGAGGUGGAUGCCUCAAUACAAGACGUGGCCAUUUCGCCAAAUGGCCAUUAUAUGGCCGCGGCCAACAAUAAGGGCAACUGCUA .(((.....)))...........(((((....((((((((((....))))).........(((((((((...(((....)))......)))))))))...........))))).))))). ( -47.40) >DroAna_CAF1 5453 120 - 1 GGACGUCAAGUCGGAGAAACACGAACGGAUUGUGCCCGAGGUGGAUGCCUCCAUACAGGACGUGGCCAUCUCCCCAGACGGACGCUAUUUGGCGGCAGCCAACAACAAAGGCAAUUGCUA ((.((((..(((((.....(((((.....)))))...(((((((..((.(((.....))).))..))))))).)).))).))))))..(((((....))))).......(((....))). ( -37.70) >consensus GGACGUCAAGUCGGAGAUGCACGAACAGAUUGUGCCCGAGGUGGAUGCCUCCAUACAGGAUGUGGCCAUCUCGCCAGAUGGCCAUUACAUGGCCGCGGCCAACAACAAGGGCAACUGCUA .(((.....)))......((((((.....))))))....((..(.(((((...........((((((((((....))))))))))....((((....)))).......))))).)..)). (-27.73 = -27.77 + 0.04)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:01:40 2006