
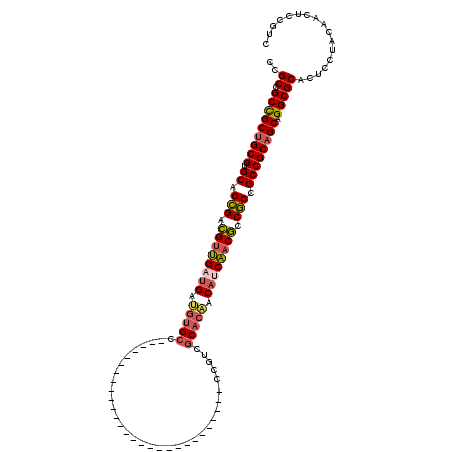
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 1,076,446 – 1,076,538 |
| Length | 92 |
| Max. P | 0.960123 |

| Location | 1,076,446 – 1,076,538 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.06 |
| Mean single sequence MFE | -48.53 |
| Consensus MFE | -39.44 |
| Energy contribution | -39.58 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.17 |
| Mean z-score | -4.22 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.51 |
| SVM RNA-class probability | 0.960123 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
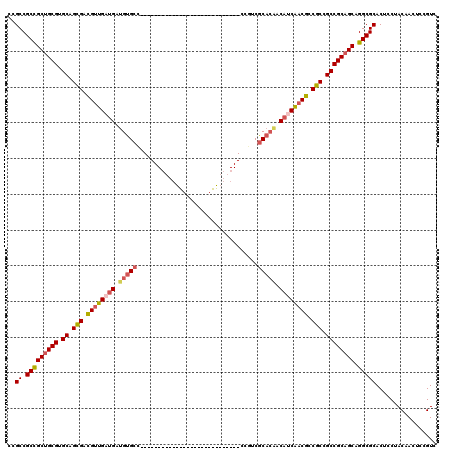
>X_DroMel_CAF1 1076446 92 + 22224390 UAGCCGCCGCCGCGUGCAGCGAUGUGGACGAUGCGCC----------------------------UCGGCGCACCACAUCCACACCGCCGCCGCAGCAGGCGCACUCCUACAACUCCGUC ..((.(((((.(((.((.(((.((((((.(.(((((.----------------------------...)))))...).)))))).))).))))).)).)))))................. ( -41.40) >DroPse_CAF1 796 92 + 1 CCGCCGCCGCUGCGUGCAGCGACGUUGAUGAUGUGCC----------------------------CCGUCGCACAACAUCAACGCCGCCGCCGCAGCAGGCGCACUCCUACAACUCCGUC ..((.(((((((((.((.(((.((((((((.(((((.----------------------------.....))))).)))))))).))).)))))))).)))))................. ( -48.00) >DroGri_CAF1 4995 116 + 1 CCGCCGCUGCUGCGUGCAGCGACGCUGAUGCUGUGCUCGUGCCCGUCGA----ACAGUUCGUGGCACGUCGCAAAACAUCAACGCCGCCGCCGCAGCAGGCGCACUCCUACAACUCCGUU .((((..(((((((.((.(((.((.(((((...(((.((((((...(((----.....))).))))))..)))...))))).)).))).))))))))))))).................. ( -52.00) >DroWil_CAF1 5886 92 + 1 UGGCCGCCGCUGCGUGCAGUGACGUUGUUGUGGUGCC----------------------------CCUACGCACUACAUCAACGCCACCGCCGCAGCAGGCGCACUCCUACAAUUCGGUA ..((.(((((((((.((.(((.(((((.((((((((.----------------------------.....)))))))).))))).))).)))))))).)))))................. ( -46.00) >DroMoj_CAF1 11639 116 + 1 CUGCCGCCGCUGCGUGCAGCGAUGUUGCUGCUGUGGCCGUGCC----AGUCGAACAGCUCGUGGCACGCCGCACAACAUCAACACCGCCGCCGCAGCAGGCGCACUCCUACAAUUCCGUC .(((.(((((((((.((.(((.(((((.((.((((((.(((((----(..(((.....)))))))))))))))...)).))))).))).)))))))).))))))................ ( -55.80) >DroPer_CAF1 796 92 + 1 CCGCCGCCGCUGCGUGCAGCGACGUUGAUGAUGUGCC----------------------------CCGUCGCACAACAUCAACGCCGCCGCCGCAGCAGGCGCACUCCUACAACUCCGUC ..((.(((((((((.((.(((.((((((((.(((((.----------------------------.....))))).)))))))).))).)))))))).)))))................. ( -48.00) >consensus CCGCCGCCGCUGCGUGCAGCGACGUUGAUGAUGUGCC____________________________CCGUCGCACAACAUCAACGCCGCCGCCGCAGCAGGCGCACUCCUACAACUCCGUC ..((.(((((((((.((.(((.((((((((.(((((..................................))))).)))))))).))).)))))))).)))))................. (-39.44 = -39.58 + 0.14)

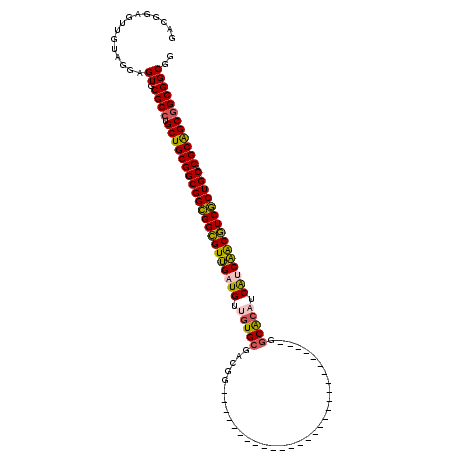
| Location | 1,076,446 – 1,076,538 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.06 |
| Mean single sequence MFE | -61.02 |
| Consensus MFE | -50.43 |
| Energy contribution | -50.05 |
| Covariance contribution | -0.38 |
| Combinations/Pair | 1.22 |
| Mean z-score | -5.26 |
| Structure conservation index | 0.83 |
| SVM decision value | 1.20 |
| SVM RNA-class probability | 0.929470 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
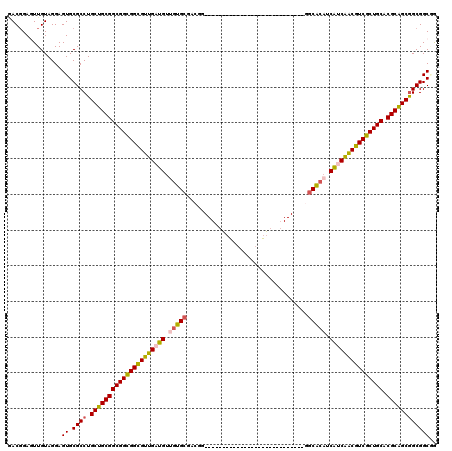
>X_DroMel_CAF1 1076446 92 - 22224390 GACGGAGUUGUAGGAGUGCGCCUGCUGCGGCGGCGGUGUGGAUGUGGUGCGCCGA----------------------------GGCGCAUCGUCCACAUCGCUGCACGCGGCGGCGGCUA ..............(((.((((.((((((((((((((((((((..(((((((...----------------------------.))))))))))))))))))))).))))))))))))). ( -60.10) >DroPse_CAF1 796 92 - 1 GACGGAGUUGUAGGAGUGCGCCUGCUGCGGCGGCGGCGUUGAUGUUGUGCGACGG----------------------------GGCACAUCAUCAACGUCGCUGCACGCAGCGGCGGCGG ...............((.((((.(((((((((((((((((((((.(((((.....----------------------------.))))).))))))))))))))).)))))))))))).. ( -57.60) >DroGri_CAF1 4995 116 - 1 AACGGAGUUGUAGGAGUGCGCCUGCUGCGGCGGCGGCGUUGAUGUUUUGCGACGUGCCACGAACUGU----UCGACGGGCACGAGCACAGCAUCAGCGUCGCUGCACGCAGCAGCGGCGG ..................((((((((((((((((((((((((((((.(((..((((((.(((.....----)))...)))))).))).))))))))))))))))).)))))))..)))). ( -69.20) >DroWil_CAF1 5886 92 - 1 UACCGAAUUGUAGGAGUGCGCCUGCUGCGGCGGUGGCGUUGAUGUAGUGCGUAGG----------------------------GGCACCACAACAACGUCACUGCACGCAGCGGCGGCCA ..((........))....((((.((((((((((((((((((.(((.((((.....----------------------------.)))).))).)))))))))))).)))))))))).... ( -52.40) >DroMoj_CAF1 11639 116 - 1 GACGGAAUUGUAGGAGUGCGCCUGCUGCGGCGGCGGUGUUGAUGUUGUGCGGCGUGCCACGAGCUGUUCGACU----GGCACGGCCACAGCAGCAACAUCGCUGCACGCAGCGGCGGCAG ................((((((.((((((((((((((((((.(((((((.(.(((((((((((...))))..)----)))))).)))))))).)))))))))))).))))))))).))). ( -69.20) >DroPer_CAF1 796 92 - 1 GACGGAGUUGUAGGAGUGCGCCUGCUGCGGCGGCGGCGUUGAUGUUGUGCGACGG----------------------------GGCACAUCAUCAACGUCGCUGCACGCAGCGGCGGCGG ...............((.((((.(((((((((((((((((((((.(((((.....----------------------------.))))).))))))))))))))).)))))))))))).. ( -57.60) >consensus GACGGAGUUGUAGGAGUGCGCCUGCUGCGGCGGCGGCGUUGAUGUUGUGCGACGG____________________________GGCACAUCAUCAACGUCGCUGCACGCAGCGGCGGCGG ...............((.((((.(((((((((((((((((((((.(((((..................................))))).))))))))))))))).)))))))))))).. (-50.43 = -50.05 + -0.38)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:42:21 2006