
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 9,131,748 – 9,131,850 |
| Length | 102 |
| Max. P | 0.987675 |

| Location | 9,131,748 – 9,131,850 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 72.43 |
| Mean single sequence MFE | -49.68 |
| Consensus MFE | -26.99 |
| Energy contribution | -26.33 |
| Covariance contribution | -0.66 |
| Combinations/Pair | 1.36 |
| Mean z-score | -2.28 |
| Structure conservation index | 0.54 |
| SVM decision value | 2.09 |
| SVM RNA-class probability | 0.987675 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
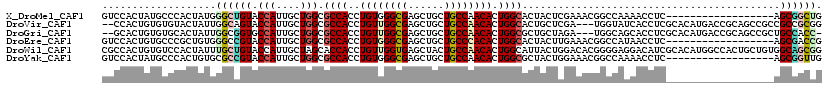
>X_DroMel_CAF1 9131748 102 + 22224390 CAGCCGCU------------------GAGGUUUUGGCCGUUUCGAGUAGUGCCAGUGUUGGCAGCAGCUCGCCCACAGGUGGCGCCAGCAAUGGUACAGCCCAUAGUGGGCAUAGUGGAC ...(((((------------------(.(((....)))..........((((((.(((((((....((.((((....))))))))))))).)))))).((((.....)))).)))))).. ( -45.20) >DroVir_CAF1 86 115 + 1 CCGCGGCGGCGGCUGCGGUCAUGUGCGAGGUGAUACCA---UCGAGCAGUGCCAGUGUUGGCAGCAGCUCGCCAACAGGUGGCGCCAGCAAUGGUACUGCCAAUAGUACACACAGUGG-- (((((((....)))))))...(((((..((((....))---))(.(((((((((.(((((((....((.((((....))))))))))))).))))))))))....)))))........-- ( -55.70) >DroGri_CAF1 6085 114 + 1 -GGUGGCAGCGGCUGCGGUCAUGUGCGAGGUGCUGCCA---UCUAGCAGCGCCAGUGUUGGCAGCAGCUCGCCAACAGGUGGCGCCAGCAAUGGCACCGCCAAUAGUGCACACAGUGC-- -.((((((((((((((.((((.(..(..((((((((..---....)))))))).)..))))).))))).))).....(((((.((((....)))).))))).....))).))).....-- ( -61.60) >DroEre_CAF1 3744 102 + 1 CGGUCGCU------------------GAGGUUAUGGCCGUUUCAAGUAGUGCCAGUGUGGGCAGCAGCUCGCCCACAGGUGGCGCCAGCAAUGGUACGGCCCACAGCGGGCACAGUGGAC .(.(((((------------------(.((.....(((((..........((((.(((((((........)))))))..))))((((....))))))))))).)))))).)......... ( -44.40) >DroWil_CAF1 95 120 + 1 CCGCUGCCACAGCAGUGGCCAUGUGCGAUGUCCUCCCCGUGUCCAGUAAUGCCAGUGUUGGCAGUAGCUCACCAACAGGUGGUGCUAGCAAUGGUACAGCAAAUAGUGGACACAGUGGCG .((((((....))))))(((((....((.....))...(((((((.((.((((......))))...(((((((....))))((((((....)))))))))...)).))))))).))))). ( -49.40) >DroYak_CAF1 1900 102 + 1 CAACCGCU------------------GAGGUUUUGGCCGUUUCCAGUAGCGCCAGUGUUGGCAGCAGCUCGCCCACAGGUGGCGCCAGCAAUGGUACGGCGCACAGUGGGCAUAGUGGAC ...(((((------------------(.(((....)))......(((.(((((......))).)).))).((((((..(((((((((....))))...)).))).)))))).)))))).. ( -41.80) >consensus CAGCCGCU__________________GAGGUUAUGCCCGUUUCCAGUAGUGCCAGUGUUGGCAGCAGCUCGCCAACAGGUGGCGCCAGCAAUGGUACAGCCAAUAGUGGACACAGUGGAC .............................((((............((.((((((.(((((((....((.((((....))))))))))))).)))))).)).......))))......... (-26.99 = -26.33 + -0.66)


| Location | 9,131,748 – 9,131,850 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 72.43 |
| Mean single sequence MFE | -44.47 |
| Consensus MFE | -20.60 |
| Energy contribution | -21.02 |
| Covariance contribution | 0.42 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.89 |
| Structure conservation index | 0.46 |
| SVM decision value | 0.68 |
| SVM RNA-class probability | 0.821086 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 9131748 102 - 22224390 GUCCACUAUGCCCACUAUGGGCUGUACCAUUGCUGGCGCCACCUGUGGGCGAGCUGCUGCCAACACUGGCACUACUCGAAACGGCCAAAACCUC------------------AGCGGCUG ((((((...((((.....)))).(..(((....)))..).....)))))).(((((((((((....))))............((......))..------------------))))))). ( -37.00) >DroVir_CAF1 86 115 - 1 --CCACUGUGUGUACUAUUGGCAGUACCAUUGCUGGCGCCACCUGUUGGCGAGCUGCUGCCAACACUGGCACUGCUCGA---UGGUAUCACCUCGCACAUGACCGCAGCCGCCGCCGCGG --.((.(((((((((((((((((((.(((....))).((((..((((((((......)))))))).))))))))).)))---)))))......)))))))).((((.((....)).)))) ( -48.00) >DroGri_CAF1 6085 114 - 1 --GCACUGUGUGCACUAUUGGCGGUGCCAUUGCUGGCGCCACCUGUUGGCGAGCUGCUGCCAACACUGGCGCUGCUAGA---UGGCAGCACCUCGCACAUGACCGCAGCCGCUGCCACC- --((((...)))).....(((((((((((....)))))))..(((((((((((.((((((((...(((((...))))).---)))))))).))))).)).....)))).....))))..- ( -53.70) >DroEre_CAF1 3744 102 - 1 GUCCACUGUGCCCGCUGUGGGCCGUACCAUUGCUGGCGCCACCUGUGGGCGAGCUGCUGCCCACACUGGCACUACUUGAAACGGCCAUAACCUC------------------AGCGACCG ............(((((..((((((..((..(.(((.((((..((((((((......)))))))).)))).)))).))..)))))).......)------------------)))).... ( -40.80) >DroWil_CAF1 95 120 - 1 CGCCACUGUGUCCACUAUUUGCUGUACCAUUGCUAGCACCACCUGUUGGUGAGCUACUGCCAACACUGGCAUUACUGGACACGGGGAGGACAUCGCACAUGGCCACUGCUGUGGCAGCGG (.((.(((((((((.....(((((((....))).)))).((((....))))......(((((....)))))....))))))))))).).....(((.....(((((....))))).))). ( -44.40) >DroYak_CAF1 1900 102 - 1 GUCCACUAUGCCCACUGUGCGCCGUACCAUUGCUGGCGCCACCUGUGGGCGAGCUGCUGCCAACACUGGCGCUACUGGAAACGGCCAAAACCUC------------------AGCGGUUG ........(((((((.((((((((((....))).)))).)))..)))))))(((((((((((....))))....(((....)))..........------------------))))))). ( -42.90) >consensus GUCCACUGUGCCCACUAUGGGCCGUACCAUUGCUGGCGCCACCUGUGGGCGAGCUGCUGCCAACACUGGCACUACUCGAAACGGCCAAAACCUC__________________AGCGGCCG ...................((((((.(((....))).((((..((((((((......)))))))).))))...........................................)))))). (-20.60 = -21.02 + 0.42)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:58:53 2006