
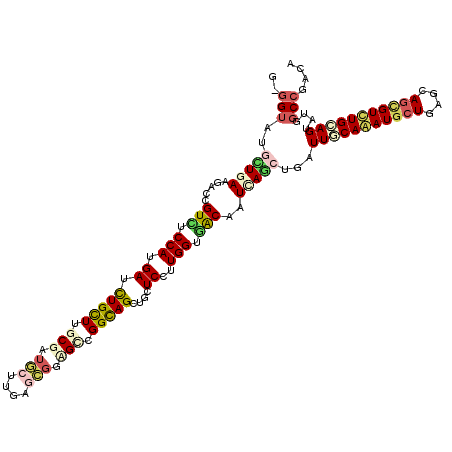
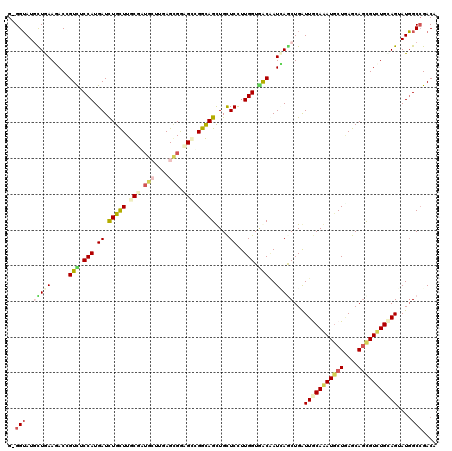
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 9,124,249 – 9,124,408 |
| Length | 159 |
| Max. P | 0.618295 |

| Location | 9,124,249 – 9,124,368 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.61 |
| Mean single sequence MFE | -42.83 |
| Consensus MFE | -25.67 |
| Energy contribution | -26.65 |
| Covariance contribution | 0.98 |
| Combinations/Pair | 1.39 |
| Mean z-score | -1.66 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.544282 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 9124249 119 + 22224390 G-GGGAUGUUGAAGACCGUCUCCAUGAUCUGCUUGCGAUGACGCAGAGGAGCCGGCAGCUGCUCCUUGGUGGCUAUCAACUGGUUGCAGAUGCUGAGCAGCGUCUGCAGGAUCGCCGACA (-(.((((((((.(.(((.((((....(((((..........))))))))).))))(((..((....))..))).)))))...(((((((((((....))))))))))).))).)).... ( -50.40) >DroVir_CAF1 73 119 + 1 G-GGUAUACUGAAGACAGUUUCCAUGAUUUGCUUGCGAUGCUUGAGCGGAGCUGGCAGUUGUUCUUUGGUGACAAUUAGCUGAUUACAAAUACUAAGCAGCGUUUGUAGUAUGGCCGACA (-(.(((((((.((((.(((.(((.((.(((((.((..(((....)))..)).)))))....))..))).))).....((((.(((.......))).)))))))).))))))).)).... ( -37.80) >DroGri_CAF1 73 119 + 1 G-GGUAUGCUAAAUACGGUUUCCAUGAUCUGUUUGCGAAGCUUGAGCGGCGUUGGCAGUUGCUCUUUGGUAACAAUAAGCUGAUUACAAAUGCUGAGCAGCGUUUGUAGUAUGGCCGACA (-(.(((((......((((((........(((..(((..((....))..)))..)))((((((....))))))...))))))..((((((((((....))))))))))))))).)).... ( -38.50) >DroMoj_CAF1 73 119 + 1 G-GGUAUGUUAAACACCGUUUCCAUGAUUUGUUUGCGAUGCUUUAGCGAUGCUGGCAGCUGCUCUUUGGUGACAAUAAGCUGGUUGCAAAUGCUGAGCAGUGUCUGGAGUAUGGCGGACA (-((.(((........))).)))......(((((((.(((((((((((.((((.(((((((((..(((....)))..))).))))))........)))).)).))))))))).))))))) ( -43.50) >DroAna_CAF1 73 119 + 1 G-GGUAUACUGAAGACAGUCUCCAUGAUCUGCUUGCGGUGGCGCAGCGGCGACGGCAGCUGCUCCUUGGUGGCGAUCAACUGGUUGCAGAUGCUCAGCAACGUCUGCAGGAUGGCCGACA (-(.(((.(((.((((........(((((.(((.((.(.((.(((((.((....)).))))).)).).))))))))))....(((((.(.....).))))))))).))).))).)).... ( -44.20) >DroPer_CAF1 75 120 + 1 GGGGUAUGCUGAAGACCGUCUCCAUGAUCUGCUUACGAUACUUGAGUGGUGCCGGUAGCUGCUCCUUGGUCACGAUCAGCUGAUUGCAGAUGCUAAGCAGCGUCUGAAGAAUGGCCGACA ((((((.((((..(((((.(((....(((.......)))....)))))))..)..))))))))))((((((((((((....)))))((((((((....)))))))).....))))))).. ( -42.60) >consensus G_GGUAUGCUGAAGACCGUCUCCAUGAUCUGCUUGCGAUGCUUGAGCGGAGCCGGCAGCUGCUCCUUGGUGACAAUCAGCUGAUUGCAAAUGCUGAGCAGCGUCUGCAGUAUGGCCGACA ..(((..(((((.....(((.(((.((.(((((.(((.(((....))).))).)))))....))..))).)))..)))))...(((((((((((....)))))))))))....))).... (-25.67 = -26.65 + 0.98)

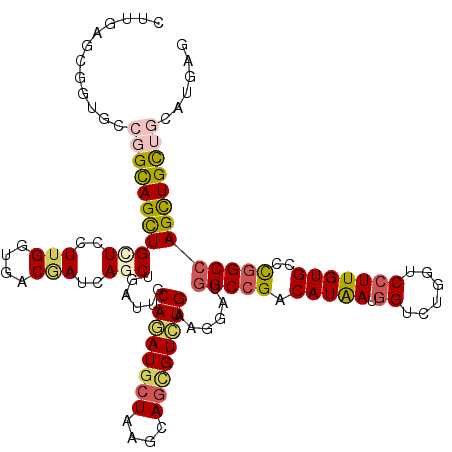
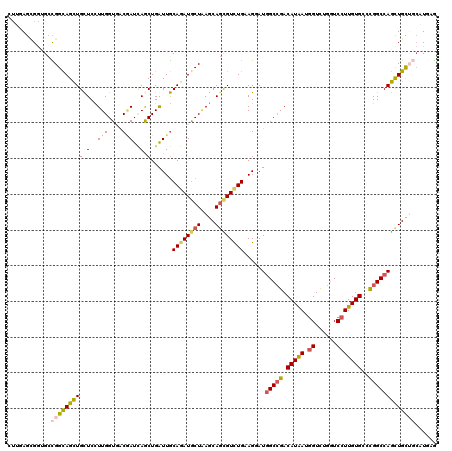
| Location | 9,124,288 – 9,124,408 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.56 |
| Mean single sequence MFE | -46.82 |
| Consensus MFE | -29.90 |
| Energy contribution | -29.35 |
| Covariance contribution | -0.55 |
| Combinations/Pair | 1.34 |
| Mean z-score | -1.75 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.17 |
| SVM RNA-class probability | 0.618295 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 9124288 120 + 22224390 ACGCAGAGGAGCCGGCAGCUGCUCCUUGGUGGCUAUCAACUGGUUGCAGAUGCUGAGCAGCGUCUGCAGGAUCGCCGACAUAAUUGUUUGGUCCUUGUGCACGGCCAGCUGUUGGAUGAG .(((.(((((((.(....).))))))).))).(((.((.(((((((((((((((....)))))))))(((((((..((((....))))))))))).......)))))).)).)))..... ( -50.30) >DroVir_CAF1 112 120 + 1 CUUGAGCGGAGCUGGCAGUUGUUCUUUGGUGACAAUUAGCUGAUUACAAAUACUAAGCAGCGUUUGUAGUAUGGCCGACAUAAUGGUCUGAUCCUUGUGACCGGCCAGUUGUAUCAUUAA ..(((..(.(((((((.((((....(((....)))...((((((((((((..((....))..)))))))).))))))))....(((((..........)))))))))))).).))).... ( -36.20) >DroPse_CAF1 112 120 + 1 CUUGAGUGGUGCCGGUAGCUGCUCCUUGGUCACGAUCAGCUGAUUGCAGAUGCUAAGCAGCGUCUGAAGGAUGGCCGACAUGAUGGUCUGGUCCUUGUGACCGGCCAGCUGCUGCAUGAG ......(.((((.((((((((..((..((((((((((....)))).((((((((....))))))))(((((((((((......)))))..)))))))))))))).)))))))))))).). ( -50.80) >DroMoj_CAF1 112 120 + 1 CUUUAGCGAUGCUGGCAGCUGCUCUUUGGUGACAAUAAGCUGGUUGCAAAUGCUGAGCAGUGUCUGGAGUAUGGCGGACAUAAUGGUUUGGUCCUUGUGGCCGGCCAGCUGUAUCAUUAG .......((((...(((((((....(((....)))...((((((..(((..((..(((.(((((((........)))))))....)))..))..)))..)))))))))))))..)))).. ( -48.50) >DroAna_CAF1 112 120 + 1 GCGCAGCGGCGACGGCAGCUGCUCCUUGGUGGCGAUCAACUGGUUGCAGAUGCUCAGCAACGUCUGCAGGAUGGCCGACAUGAUGGUCUGGUCCUUGUGCAUGGCCAGCUGCUGCAUGAG ..((((((((((((...((((..(.((....((((((....)))))).)).)..))))..))))((((.((.(((((((......))).)))).)).))))......))))))))..... ( -49.50) >DroPer_CAF1 115 120 + 1 CUUGAGUGGUGCCGGUAGCUGCUCCUUGGUCACGAUCAGCUGAUUGCAGAUGCUAAGCAGCGUCUGAAGAAUGGCCGACAUGAUGGUCUGGUCCUUGUGCCCGGCCAGCUGCUGCAUGAG ......(.((((.(((((((.....((((((((((((....)))))((((((((....)))))))).....)))))))......((((.(((......))).))))))))))))))).). ( -45.60) >consensus CUUGAGCGGUGCCGGCAGCUGCUCCUUGGUGACGAUCAGCUGAUUGCAGAUGCUAAGCAGCGUCUGAAGGAUGGCCGACAUAAUGGUCUGGUCCUUGUGCCCGGCCAGCUGCUGCAUGAG ............((((((((((...(..((........))..)..))(((((((....))))))).......(((((.(((((.((......)))))))..)))))))))))))...... (-29.90 = -29.35 + -0.55)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:58:38 2006