
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 9,122,454 – 9,122,650 |
| Length | 196 |
| Max. P | 0.844889 |

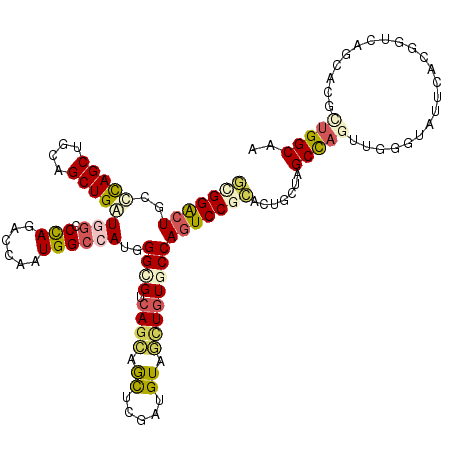
| Location | 9,122,454 – 9,122,574 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.17 |
| Mean single sequence MFE | -52.10 |
| Consensus MFE | -27.29 |
| Energy contribution | -27.88 |
| Covariance contribution | 0.59 |
| Combinations/Pair | 1.39 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.52 |
| SVM decision value | 0.77 |
| SVM RNA-class probability | 0.844889 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 9122454 120 + 22224390 GCGGACUUCCCAGCUGCAGCUGUUGACCUAGACCGAUGGCCAUCGGUGUCAGCAAUUCGAUGUAGUUGUGCCAGUCCGCGCUGCUCGCCAGUUGCGUAUUCACAGUCAGCACGCUGGCAA (((((((..(((((((((..(((((((....((((((....)))))))))))))......)))))))).)..))))))).......(((((((((.............))).)))))).. ( -51.32) >DroVir_CAF1 4825 120 + 1 CUGGGCUGGUUAGCUGCAGCUGAUUGGCCAGGCCAAUGGCCAUCGGGGUCAAUAGCUCAAUGUAACUGUGCCAGUCCGUGCUGGCAGCCAGUUGGGUAUUAACCGUCAGCACGGUGGCAA ...(((..((((((....))))))..))).((((...))))....((((.....))))..........(((((..((((((((((...(....)(((....)))))))))))))))))). ( -52.80) >DroPse_CAF1 3013 120 + 1 GUGGACUCCCCAGCUGCAGCUGGUGGCCCAGACCGAUGGCCAUGGGCGUCAGAAGCUCGAUGUAGCUGUGCCAGUCCGCACUGCUGGCUAGGUUGCUUGUCACGGUCAGCACAUUGGCGA (((((((..((((((((.(((.((((((.........)))))).)))(((........)))))))))).)..)))))))..((((((((..(........)..))))))))......... ( -50.20) >DroGri_CAF1 5725 120 + 1 ACGGGCUGCUCAGCUGCAGCUGAUUGGCCAUUCCAAUGGCCAUUGGGGUCAGCAACUCAAUGUAGCUGUGCCAAUCCGUGCUGGCAGCCAGUUGUGGAUUAAUCGUUAGCACGGUGGCAA ...(((((((((((....)))))..((((((....))))))((((((........)))))))))))).(((((..((((((((((..(((....))).......))))))))))))))). ( -52.60) >DroAna_CAF1 3720 120 + 1 GGGGACUGCCCAGCUGGAGCUGCUGGCCCAGGCCAAUGGCCAUGGGCGUGAGCAGUUCGAUGUAGCUGUGCCAGUCCACACUGCACGCCAGUUGCGAGUUGACGCUCAACACUCCGGCGA ..(((((((.((((((((((((((.(((((((((...)))).)))))...))))))))....)))))).).))))))........(((((((...((((....))))...)))..)))). ( -56.50) >DroPer_CAF1 4088 120 + 1 GAGGACUCCCCAGCUGCAGCUGGUGGCCCAGACCGAUGGCCAUGGGCGUCAGCAGCUCGAUGUAGCUGUGCCAGUCCGCACUGCUGGCUAGGUUGCUUGUCACGGUCAGCACAUUGGCGA ..(((((..(((((((((((((.(((((((..((...))...)))))..)).))))....)))))))).)..)))))((..((((((((..(........)..)))))))).....)).. ( -49.20) >consensus GCGGACUGCCCAGCUGCAGCUGAUGGCCCAGACCAAUGGCCAUGGGCGUCAGCAGCUCGAUGUAGCUGUGCCAGUCCGCACUGCUAGCCAGUUGGGUAUUCACGGUCAGCACGCUGGCAA (((((((..(((((....)))))(((.(((......))))))..((((.((((.((.....)).))))))))))))))).......(((((......................))))).. (-27.29 = -27.88 + 0.59)


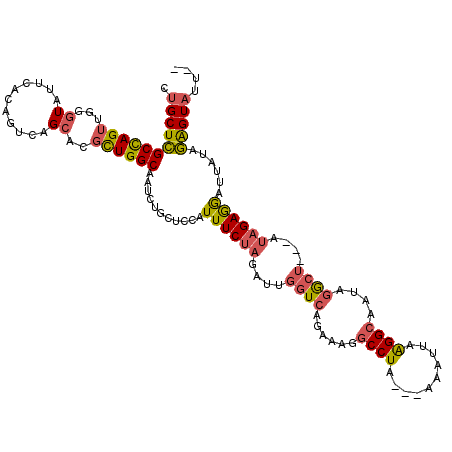
| Location | 9,122,534 – 9,122,650 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.66 |
| Mean single sequence MFE | -34.44 |
| Consensus MFE | -21.19 |
| Energy contribution | -21.28 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.30 |
| Mean z-score | -1.72 |
| Structure conservation index | 0.62 |
| SVM decision value | -0.00 |
| SVM RNA-class probability | 0.533033 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 9122534 116 + 22224390 CUGCUCGCCAGUUGCGUAUUCACAGUCAGCACGCUGGCAAUCUGCUCCAUUUCUAGAUUGGUCAGGAAGGCCUAA--AAAUUAAGGCAAUAGGCUAUUAUAGAGAAUUAUAGAGUAUU-- .((((((((((((((.............))).))))))...........((((((...(((((......((((..--......))))....)))))...))))))......)))))..-- ( -33.72) >DroSec_CAF1 3774 116 + 1 CUGCUCGCCAGUUGCGUAUUCACAGUCAGCACGCUGGCAAUUUGCUCCAUUUCUAGAUUGGUCAGGAAGGCCUAC--AAAUUAAGGCAAUUGGCUAUUAUAGAGGAUUAUAGAGUAUU-- .((((((((((((((.............))).)))))).......(((...((((...(((((((....((((..--......))))..)))))))...))))))).....)))))..-- ( -37.02) >DroSim_CAF1 3770 116 + 1 CUGCUCGCCAGUUGUGUAUUCACAGUCAGCACGCUGGCAAUUUGCUCCAUUUCUAGAUUGGUCAGGAAGGCCUAA--AAAUUAAGGCAAUUGGCUAUUAUAGAGGAUUAUAGAGUAUU-- .(((((((((((.((((...........)))))))))).......(((...((((...(((((((....((((..--......))))..)))))))...))))))).....)))))..-- ( -37.60) >DroEre_CAF1 3893 111 + 1 CUGCUCGCCAGUUUGGUAUUCACAGUCAGCACGCUGGCAAUCUGCUCCAUUUCUAGGUUGGUCAGAAAGGCCUA---AAAUUGGGGCAAUAGGCU---GUAGAGGGUUAUAGAGU-UA-- ...(((..(((((((.........(((((....)))))....(((((((.((.((((((.........))))))---.)).))))))).))))))---)..)))...........-..-- ( -34.50) >DroYak_CAF1 3907 112 + 1 CUGCUCGCCAGUUGGGUAUUUACAGUCAGCACGCUGGCAAUCUGCUCCAUUUCUAGGUUGGUCAGAAAGGCCUG---AAAUGGGGGCAAUAGGCU---AUAGAGGGUUAUGGGGUAUU-- .((((((((.((((..........(((((....)))))......(((((((((.(((((.........))))))---)))))))).)))).)))(---((((....))))))))))..-- ( -35.70) >DroPer_CAF1 4168 111 + 1 CUGCUGGCUAGGUUGCUUGUCACGGUCAGCACAUUGGCGAUCUGCUCCAUUUCGAGAUUGGUGAGAAAGGCCUAUUGAAAGAAAGGGAAA--AA------AGAAAGUCA-ACAGUAGUCC (((((((((.(..((.......))..))))...(((((..(((..(((.((((..(((.(((.......))).)))....)))).)))..--..------)))..))))-)))))))... ( -28.10) >consensus CUGCUCGCCAGUUGGGUAUUCACAGUCAGCACGCUGGCAAUCUGCUCCAUUUCUAGAUUGGUCAGAAAGGCCUA___AAAUUAAGGCAAUAGGCU___AUAGAGGAUUAUAGAGUAUU__ .(((((((((((...((...........))..))))))...........((((((....((((......((((..........))))....))))....))))))......))))).... (-21.19 = -21.28 + 0.09)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:58:35 2006