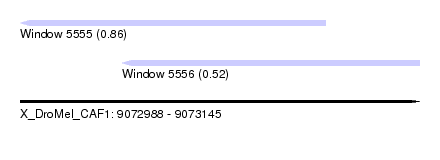
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 9,072,988 – 9,073,145 |
| Length | 157 |
| Max. P | 0.860115 |

| Location | 9,072,988 – 9,073,108 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 74.83 |
| Mean single sequence MFE | -42.50 |
| Consensus MFE | -21.83 |
| Energy contribution | -21.03 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.42 |
| Mean z-score | -1.94 |
| Structure conservation index | 0.51 |
| SVM decision value | 0.82 |
| SVM RNA-class probability | 0.860115 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 9072988 120 - 22224390 AUUUCGCGUUCCAAGCCGGUCAGCUGCUCCCUGAGCAGCUCCAAGUGCAGUACGAACUGCAGAUUGCGCAGAUACGUCAGCCACACGAGCGGCUCAUAGGUGCUCCAAAAGAGCAACAUA .....((((.....(((((((((((((((...)))))))).....((((((....))))))))))).))....)))).((((.(....).))))....(.(((((.....))))).)... ( -40.30) >DroVir_CAF1 4053 120 - 1 AGCUCGCAUUCCAAUUGCACCAGCUGUUGCCGCAACAGCUCCAGGUGCAGCAAAAAUUGCGUGUUGCGCAAAUGCACCAGAAACACAAACGGCUGAAAGGUGCUCCAAAAGAGCAGAAUA .((((((((.....(((((((((((((((...))))))))...)))))))......(((((.....)))))))))................(((....))).........))))...... ( -38.90) >DroPse_CAF1 499 120 - 1 AGCUCCCGUUCGAGCUGGGCCAGCUGCUGGCGGAGCAGCUCCAUGUGCAGCACAAACUGUACGUUCCGCAUGUACAUGAGGAAAACGAGCGGCUCGAAUGUGCUCCAGAAGAGUAUCAAG ......(((((((((((((..(((((((.....)))))))))((((((((......))))))))(((.(((....))).))).......)))))))))))(((((.....)))))..... ( -46.40) >DroYak_CAF1 478 120 - 1 AUUUCGCGUUCCAAAGCGUUCAGGUGCUCCCGAAGCAUCUCCAGGUGCAGCACAAACUGCAGAUUUCGCAGAUACGUCAGCCACACGAGCGGCUCGUAGGUGCUCCAAAAGAGCAACAUG ....(((........)))....(.(((((.....(((((....)))))(((((...((((.......)))).((((..((((.(....).)))))))).)))))......))))).)... ( -36.60) >DroAna_CAF1 2547 120 - 1 AGCUCCUGUUCCAGCUGGGCCAGCUGCUGACGGAGUAGCUCCAUGUGAAGGAUAAACUGGAUGUUUCGGAGGUAGGUCAGGACCACCAGGGGCUCGUAGGUGGUCCAGAAGAGCAGAAAC .....((((((...((((((((.((((.......(.((((((.(.(((((.((.......)).))))).)(((.(((....)))))).))))))))))).))))))))..)))))).... ( -46.41) >DroPer_CAF1 499 120 - 1 AGCUCCCGUUCGAGCUGGGCCAGCUGCUGGCGGAGCAGCUCCAUGUGCAGCACAAACUGUACGUUCCGCAUGUACAUGAGGAAAACGAGCGGCUCGAAUGUGCUCCAGAAGAGUAUCAAG ......(((((((((((((..(((((((.....)))))))))((((((((......))))))))(((.(((....))).))).......)))))))))))(((((.....)))))..... ( -46.40) >consensus AGCUCCCGUUCCAACUGGGCCAGCUGCUGCCGGAGCAGCUCCAUGUGCAGCACAAACUGCACGUUCCGCAGAUACAUCAGGAACACGAGCGGCUCGAAGGUGCUCCAAAAGAGCAACAAG ..........((.....(((((((((((.....)))))))....((((((......))))))............................))))....))(((((.....)))))..... (-21.83 = -21.03 + -0.80)


| Location | 9,073,028 – 9,073,145 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 74.12 |
| Mean single sequence MFE | -43.83 |
| Consensus MFE | -18.77 |
| Energy contribution | -18.13 |
| Covariance contribution | -0.63 |
| Combinations/Pair | 1.44 |
| Mean z-score | -1.84 |
| Structure conservation index | 0.43 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.521062 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 9073028 117 - 22224390 UUCGCU---AGCAAAUCGCGAGUACUCCGCCAGCAGACCCAUUUCGCGUUCCAAGCCGGUCAGCUGCUCCCUGAGCAGCUCCAAGUGCAGUACGAACUGCAGAUUGCGCAGAUACGUCAG ...(((---.((....(....)......)).))).(((..((((((((.((......((..((((((((...))))))))))...((((((....)))))))).)))).))))..))).. ( -37.50) >DroVir_CAF1 4093 117 - 1 AACGCU---GGCAAAGCUCGAGUACUCAACGAGCAGCCCCAGCUCGCAUUCCAAUUGCACCAGCUGUUGCCGCAACAGCUCCAGGUGCAGCAAAAAUUGCGUGUUGCGCAAAUGCACCAG ...(((---((....(((((.........)))))....)))))..((((.....(((((((((((((((...))))))))...)))))))......(((((.....)))))))))..... ( -44.50) >DroPse_CAF1 539 117 - 1 UAGGCU---GGCAAAUCGCGAGUACUCUGCCAGUAGGGAAAGCUCCCGUUCGAGCUGGGCCAGCUGCUGGCGGAGCAGCUCCAUGUGCAGCACAAACUGUACGUUCCGCAUGUACAUGAG ..((((---(((.....)).....((((((((((((((..(((((......)))))...))..))))))))))))))))).((((((((((...(((.....)))..)).)))))))).. ( -50.20) >DroYak_CAF1 518 117 - 1 UUCGCU---GGCGAAUCGCGAGUACUCCGCCAGGAGUCCCAUUUCGCGUUCCAAAGCGUUCAGGUGCUCCCGAAGCAUCUCCAGGUGCAGCACAAACUGCAGAUUUCGCAGAUACGUCAG ....((---((((.((((((((.(((((....))))).....)))))........(((.(((((((((.....))))))).....(((((......)))))))...))).))).)))))) ( -41.60) >DroAna_CAF1 2587 120 - 1 UCCGCUGCAGGCGAAUCUCGAAUACUCGGCCAGGAGACGGAGCUCCUGUUCCAGCUGGGCCAGCUGCUGACGGAGUAGCUCCAUGUGAAGGAUAAACUGGAUGUUUCGGAGGUAGGUCAG .........(((..((((((((((..(((((.(....)((((((((((((.((((((...))))))..))))).).)))))).......)).....)))..)))))..)))))..))).. ( -39.00) >DroPer_CAF1 539 117 - 1 UAGGCU---GGCAAAUCGCGAGUACUCUGCCAGUAGGGAAAGCUCCCGUUCGAGCUGGGCCAGCUGCUGGCGGAGCAGCUCCAUGUGCAGCACAAACUGUACGUUCCGCAUGUACAUGAG ..((((---(((.....)).....((((((((((((((..(((((......)))))...))..))))))))))))))))).((((((((((...(((.....)))..)).)))))))).. ( -50.20) >consensus UACGCU___GGCAAAUCGCGAGUACUCCGCCAGCAGACCCAGCUCCCGUUCCAACUGGGCCAGCUGCUGCCGGAGCAGCUCCAUGUGCAGCACAAACUGCACGUUCCGCAGAUACAUCAG ...((.....)).....(((.........................................(((((((.....)))))))....((((((......))))))....)))........... (-18.77 = -18.13 + -0.63)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:58:17 2006