
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 8,989,273 – 8,989,409 |
| Length | 136 |
| Max. P | 0.799980 |

| Location | 8,989,273 – 8,989,384 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 73.80 |
| Mean single sequence MFE | -44.30 |
| Consensus MFE | -17.45 |
| Energy contribution | -19.37 |
| Covariance contribution | 1.92 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.12 |
| Structure conservation index | 0.39 |
| SVM decision value | 0.61 |
| SVM RNA-class probability | 0.799980 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8989273 111 - 22224390 AUCAUCGCUGAUAUCGAUGACGGCGUCGGGUAGUUUGCUUGUCGCAACAAUCGGACGAUUGUUGCGAAAAUUCGGCUUCGUUUUGCGACA---AU-UGUCGGAAUUG-----GUGGUGGC .(((((((..((..((((....(((.(((((.....))))).)))....))))((((((((((((((((...((....))))))))))))---))-))))...))..-----))))))). ( -45.80) >DroPse_CAF1 1135 102 - 1 GUCAUCGCUAAUGUCGAUGACAGUUUCAGGAGCACCGC------------UGCCUCGAUUGUUGCGAAAAUUCGGCU------UACGGCGGACAUGGGGCGGCCCCGACGAAGGGGUGGC ((((((((....).)))))))...........((((((------------(((((((..((((.(((....)))((.------....)).))))))))))))).((......)))))).. ( -38.00) >DroSec_CAF1 1192 111 - 1 AUCAUCGCUGAUAUCGAUGACGGCGUCGGCUAGCUUGCUUAUCGCACCAAUCGGACGAUUGCUGCGGAAGUUUGGCUUCGGCUUGCGACC---AU-UGUCGGCAUUG-----GUGGUGGC .(((((((..((..(((..(.(((((.((((((((.((((.(((((.(((((....))))).)))))))))..))))..)))).))).))---.)-..)))..))..-----))))))). ( -51.10) >DroSim_CAF1 2936 111 - 1 AUCAUCGCUGAUAUCGAUGACGGCGUCGGCUAGCUUGCUUACCGCACCAAUCGGACGAUUGCUGCGGAAGUUUGGCUUCGGCUUGCGACC---AU-UGUCGGCAUUG-----GUGGUGGC .(((((((..((..(((..(.(((((.((((((((.((((.(((((.(((((....))))).)))))))))..))))..)))).))).))---.)-..)))..))..-----))))))). ( -54.00) >DroEre_CAF1 1204 105 - 1 AUCAUCGCUGAUAUCGAUGACGGCGUCGGGUGCUCCGCCUGCUGCACCAAUGGGACGAUUGUUGCGGAAGUU------CGGCUUGCGGCG---AU-UGACGGCAUUG-----GUGGUGGC .(((((((..((..((......(((.((((((...)))))).))).((....)).(((((((((((..((..------...)))))))))---))-)).))..))..-----))))))). ( -38.20) >DroYak_CAF1 2817 105 - 1 AUCAUCGCUGAUAUCAAUGACGGCUUCGGGUGCUCCGCCUGCCGCACCAAUUGGACGAUUGCUGCGGAAGUU------CGGCUUGCGACG---AU-UGGCGACAUUG-----GUGGGGGC ......(((..((((((((.((.(.(((..(((.(((.((.(((((.(((((....))))).))))).))..------)))...))).))---).-.).)).)))))-----)))..))) ( -38.70) >consensus AUCAUCGCUGAUAUCGAUGACGGCGUCGGGUACUCCGCUUGCCGCACCAAUCGGACGAUUGCUGCGGAAGUU_GGCU_CGGCUUGCGACG___AU_UGUCGGCAUUG_____GUGGUGGC .(((((((......(((((.((.((.......(.((((...(((((.(((((....))))).)))))(((((.......))))))))).)......)).)).))))).....))))))). (-17.45 = -19.37 + 1.92)

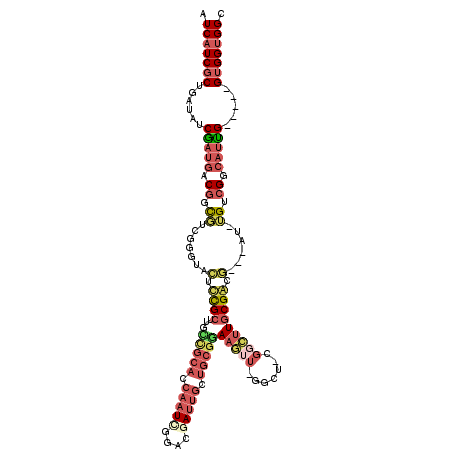
| Location | 8,989,304 – 8,989,409 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 74.22 |
| Mean single sequence MFE | -32.40 |
| Consensus MFE | -17.02 |
| Energy contribution | -18.88 |
| Covariance contribution | 1.86 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.04 |
| SVM RNA-class probability | 0.553709 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8989304 105 + 22224390 CGAAGCCGAAUUUUCGCAACAAUCGUCCGAUUGUUGCGACAAGCAAACUACCCGACGCCGUCAUCGAUAUCAGCGAUGAUGAUGUUGAUAGUCCUCCAUCUCGUA ......(((....(((((((((((....))))))))))).......((((..(((((.((((((((.......)))))))).))))).))))........))).. ( -34.80) >DroPse_CAF1 1172 90 + 1 ---AGCCGAAUUUUCGCAACAAUCGAGGCA------------GCGGUGCUCCUGAAACUGUCAUCGACAUUAGCGAUGACGAAACACCCAAUCCAUCAUCGCGAA ---.........(((((.....((.(((.(------------((...))))))))...((((((((.(....)))))))))...................))))) ( -21.20) >DroSec_CAF1 1223 105 + 1 CGAAGCCAAACUUCCGCAGCAAUCGUCCGAUUGGUGCGAUAAGCAAGCUAGCCGACGCCGUCAUCGAUAUCAGCGAUGAUGAUGUUGACACUCCUCCAUCUCGUG ((((((....(((.((((.(((((....))))).))))..)))...))).(.(((((.((((((((.......)))))))).))))).)...........))).. ( -31.60) >DroSim_CAF1 2967 105 + 1 CGAAGCCAAACUUCCGCAGCAAUCGUCCGAUUGGUGCGGUAAGCAAGCUAGCCGACGCCGUCAUCGAUAUCAGCGAUGAUGAUGUUGACACUCCUCCGUCUCGUG ((((((....((((((((.(((((....))))).))))).)))...))).(.(((((.((((((((.......)))))))).))))).)...........))).. ( -36.50) >DroEre_CAF1 1235 99 + 1 CG------AACUUCCGCAACAAUCGUCCCAUUGGUGCAGCAGGCGGAGCACCCGACGCCGUCAUCGAUAUCAGCGAUGAUGAGGUCGACAGUCCUCCGUCUCGUG ..------.......(((.((((......)))).))).((((((((((.((.((((..((((((((.......))))))))..))))...)).)))))))).)). ( -36.00) >DroYak_CAF1 2848 99 + 1 CG------AACUUCCGCAGCAAUCGUCCAAUUGGUGCGGCAGGCGGAGCACCCGAAGCCGUCAUUGAUAUCAGCGAUGAUGAGGCCGAAAGUCCUCCAUCUCGUG .(------(....(((((.((((......)))).)))))..(((((..(....)...))))).......)).((((.(((((((.(....).))).)))))))). ( -34.30) >consensus CG_AGCC_AACUUCCGCAACAAUCGUCCGAUUGGUGCGGCAAGCAAACCACCCGACGCCGUCAUCGAUAUCAGCGAUGAUGAUGUUGACAGUCCUCCAUCUCGUG ..........((((((((.((((......)))).))))).))).........((((..((((((((.......))))))))..)))).................. (-17.02 = -18.88 + 1.86)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:57:44 2006