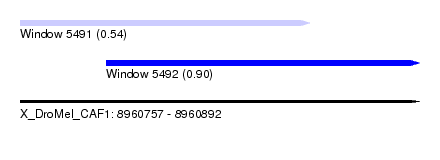
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 8,960,757 – 8,960,892 |
| Length | 135 |
| Max. P | 0.903291 |

| Location | 8,960,757 – 8,960,855 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | forward |
| Mean pairwise identity | 82.35 |
| Mean single sequence MFE | -39.10 |
| Consensus MFE | -28.90 |
| Energy contribution | -29.30 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.30 |
| Mean z-score | -1.38 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.544839 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8960757 98 + 22224390 ---CAGCAUACCCACAAACGGCAACGAUUUGGCCAGCAUGCUGGGUGGCCUGGGUGCC-CCGCCGGCUGCAUCACCGUCUCUGGCGGCAUCAGCUCCCGUUG ---((((....((((...((....))......((((....))))))))...(((.((.-(((((((..((......))..))))))).....)).))))))) ( -38.00) >DroVir_CAF1 7155 95 + 1 CAACAGCAUACCUACAAACGGCAAUGAUCUGGCGAGCAUGCUGGGACGCCUAAAUGUUGCC---GG----UGGUGCGCCAGUCCAUGCACUGGCGCCGCUUG ....(((..(((......(((((((.....((((..(......)..)))).....))))))---).----.)))((((((((......)))))))).))).. ( -35.60) >DroSec_CAF1 6520 98 + 1 ---CAGCAUACCCACAAACGGCAACGAUUUGGCCAGCAUGCUGGGUGGCCUGGGAGCC-CCACCGACUGCAUCAGCGCCUCUGGCGGCUCCAGCUUCCGUUG ---((((((.(...((((((....)).))))....).))))))((.(((...((((((-(((.((.(((...)))))....))).)))))).))).)).... ( -37.10) >DroSim_CAF1 7285 98 + 1 ---CAGCAUACCCACAAACGGCAACGAUUUGGCCAGCAUGCUGGGUGGCCUGGGAGCC-CCACCGGCUGCAUCAGCGCCUCUGGCGGCUCCAGCUUCCGUUG ---((((((.(...((((((....)).))))....).))))))((.(((...((((((-(((..(((.((....)))))..))).)))))).))).)).... ( -43.40) >DroEre_CAF1 6787 98 + 1 ---CAGCAUACCCACAAACGGCAACGAUUUGGCCAGCAUGCUGGGUGGACUGGGAGCC-CCAACGGCUGCAUCAGCGCCUCUGUCGGCUCCAGCUCCAGUUG ---((((..(((((((((((....)).))))((......)))))))(((((((.((((-.((..(((.((....)))))..))..)))))))).))).)))) ( -38.70) >DroYak_CAF1 6325 98 + 1 ---CAGCAUACCCACAAACGGCAACGAUUUGGCCAGCAUGUUGGGUGGACUGGGAGCC-CCGCCGGCUGCAUCAGCGCCACUGGCGGCUCCAGCUCCCGUUG ---((((....((((...((....))......((((....))))))))...((((((.-(((((((..((......))..))))))).....)))))))))) ( -41.80) >consensus ___CAGCAUACCCACAAACGGCAACGAUUUGGCCAGCAUGCUGGGUGGCCUGGGAGCC_CCACCGGCUGCAUCAGCGCCUCUGGCGGCUCCAGCUCCCGUUG ...((((((.(...((((((....)).))))....).))))))((.((.((((.((((.(((..(((.((....)))))..))).)))))))))).)).... (-28.90 = -29.30 + 0.40)


| Location | 8,960,786 – 8,960,892 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 107 |
| Reading direction | forward |
| Mean pairwise identity | 79.69 |
| Mean single sequence MFE | -50.27 |
| Consensus MFE | -37.44 |
| Energy contribution | -38.25 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.89 |
| Structure conservation index | 0.74 |
| SVM decision value | 1.03 |
| SVM RNA-class probability | 0.903291 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8960786 106 + 22224390 CCAGCAUGCUGGGUGGCCUGGGUGCC-CCGCCGGCUGCAUCACCGUCUCUGGCGGCAUCAGCUCCCGUUGGCAACCUUAUCGAUGGCCAUGCCGGCAGUCUGCUGGC ((((((((((((((((((.(((.((.-(((((((..((......))..))))))).....)).)))(((((........))))))))))).))))))...)))))). ( -50.80) >DroVir_CAF1 7187 94 + 1 CGAGCAUGCUGGGACGCCUAAAUGUUGCC---GG----UGGUGCGCCAGUCCAUGCACUGGCGCCGCUUGGCAG------CGAUGUCAACAGCAGCAGCCUGCUGGG (.((((.((((...........(((((((---((----((..((((((((......))))))))))).))))))------)).(((.....))).)))).)))).). ( -41.40) >DroSec_CAF1 6549 106 + 1 CCAGCAUGCUGGGUGGCCUGGGAGCC-CCACCGACUGCAUCAGCGCCUCUGGCGGCUCCAGCUUCCGUUGGCAACCUCAUCGAUGGCCAUGCCGGCAGUCUGCUGGC ((((((((((((((((((..((((((-(((.((.(((...)))))....))).)))))).(((......)))............)))))).))))))...)))))). ( -49.70) >DroSim_CAF1 7314 106 + 1 CCAGCAUGCUGGGUGGCCUGGGAGCC-CCACCGGCUGCAUCAGCGCCUCUGGCGGCUCCAGCUUCCGUUGGCAACCUCAUCGAUGGCCAUGCCGGCAGUCUGCUGGC ((((((((((((((((((..((((((-(((..(((.((....)))))..))).)))))).(((......)))............)))))).))))))...)))))). ( -56.00) >DroEre_CAF1 6816 106 + 1 CCAGCAUGCUGGGUGGACUGGGAGCC-CCAACGGCUGCAUCAGCGCCUCUGUCGGCUCCAGCUCCAGUUGGCAACCUCAUCGAUGGCCAUGCCGGCGGUCUGCUGGG (((((((((..(.((((((((.((((-.((..(((.((....)))))..))..)))))))).)))).)..)))........(((.(((.....))).))))))))). ( -52.80) >DroYak_CAF1 6354 106 + 1 CCAGCAUGUUGGGUGGACUGGGAGCC-CCGCCGGCUGCAUCAGCGCCACUGGCGGCUCCAGCUCCCGUUGGCAACCUCAUCGAUGGCCAUGCCGGCGGUCUGCUGGG ((((((....((((......((((((-(((..(((.((....)))))..))).)))))).))))(((((((((.((........))...)))))))))..)))))). ( -50.90) >consensus CCAGCAUGCUGGGUGGCCUGGGAGCC_CCACCGGCUGCAUCAGCGCCUCUGGCGGCUCCAGCUCCCGUUGGCAACCUCAUCGAUGGCCAUGCCGGCAGUCUGCUGGC ((((((.((((((.((.((((.((((.(((..(((.((....)))))..))).)))))))))))))(((((((.((........))...)))))))))).)))))). (-37.44 = -38.25 + 0.81)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:57:16 2006