
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 8,936,446 – 8,936,550 |
| Length | 104 |
| Max. P | 0.996378 |

| Location | 8,936,446 – 8,936,550 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 85.30 |
| Mean single sequence MFE | -35.73 |
| Consensus MFE | -32.49 |
| Energy contribution | -31.97 |
| Covariance contribution | -0.52 |
| Combinations/Pair | 1.23 |
| Mean z-score | -3.98 |
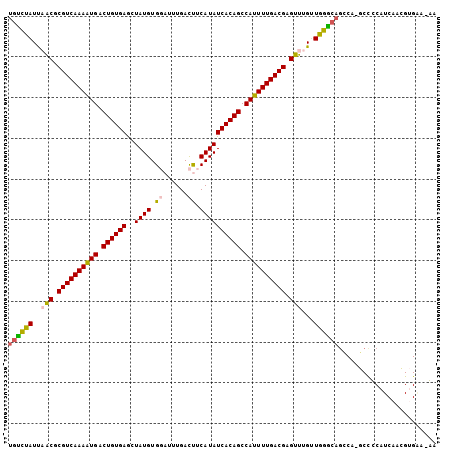
| Structure conservation index | 0.91 |
| SVM decision value | 2.69 |
| SVM RNA-class probability | 0.996378 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
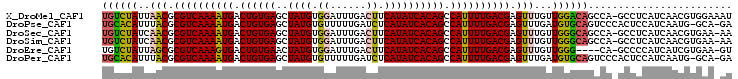
>X_DroMel_CAF1 8936446 104 + 22224390 UGUCUAUUAACGCGUCAAAAUGACUGUGAGCUAUGUGGAUUUGACUUCAUAUCACAGCCAUUUUGACGAGUUUGUUGGACAGCCA-GCCUCAUCAACGUGGAAAU ((((((..(((.((((((((((.((((((....((((((......)))))))))))).)))))))))).)))...))))))....-.((.(......).)).... ( -35.10) >DroPse_CAF1 32058 103 + 1 UGCACAUUUACGCGUCAAAAUGACUGUGAGCUAUGUGUUUUUGAUCUCAUAUCACAGCCAUUUUGACGAGUUUGAUGUGCAGUCCCACUCCAUCAAUG-GCA-GA ((((((((.((.((((((((((.((((((..((((.(........).)))))))))).)))))))))).))..))))))))......(((((....))-).)-). ( -39.00) >DroSec_CAF1 994 103 + 1 UGUCUAUCAACGCGUCAAAAUGACUGUGAGCUAUGUGGAUUUGACUUCAUAUCACAGCCAUUUUGACGAGUUUGUUGGGCAGCCA-GCCUCAUCAACGUGAA-AA ((((((.((((.((((((((((.((((((....((((((......)))))))))))).)))))))))).).))).))))))....-...((((....)))).-.. ( -33.90) >DroSim_CAF1 485 103 + 1 UGUCUAUCAACGCGUCAAAAUGACUGUGAGCUAUGUGGAUUUGACUUCAUAUCACAGCCAUUUUGACGAGUUUGUUGGGCAGCCA-GCCUCAUCAACGUGAA-AA ((((((.((((.((((((((((.((((((....((((((......)))))))))))).)))))))))).).))).))))))....-...((((....)))).-.. ( -33.90) >DroEre_CAF1 775 99 + 1 UGUCUAUUAGCGCGUCAAAGUGACUGUGAACUAUGUGGAUUUGACUUCAUAUCACAGCCAUUUUGACGAGUUUGUUGGG----CA-GCCCCAUCAUCGUGAA-GU ((((((..(((.((((((((((.((((((....((((((......)))))))))))).)))))))))).)))...))))----))-................-.. ( -33.50) >DroPer_CAF1 39923 103 + 1 UGCACAUUUACGCGUCAAAAUGACUGUGAGCUAUGUGUUUUUGAUCUCAUAUCACAGCCAUUUUGACGAGUUUGAUGUGCAGUCCCACUCCAUCAAUG-GCA-GA ((((((((.((.((((((((((.((((((..((((.(........).)))))))))).)))))))))).))..))))))))......(((((....))-).)-). ( -39.00) >consensus UGUCUAUUAACGCGUCAAAAUGACUGUGAGCUAUGUGGAUUUGACUUCAUAUCACAGCCAUUUUGACGAGUUUGUUGGGCAGCCA_GCCCCAUCAACGUGAA_AA ((((((..(((.((((((((((.((((((..((((.((......)).)))))))))).)))))))))).)))...))))))........................ (-32.49 = -31.97 + -0.52)

| Location | 8,936,446 – 8,936,550 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 85.30 |
| Mean single sequence MFE | -35.88 |
| Consensus MFE | -27.57 |
| Energy contribution | -28.27 |
| Covariance contribution | 0.69 |
| Combinations/Pair | 1.06 |
| Mean z-score | -4.39 |
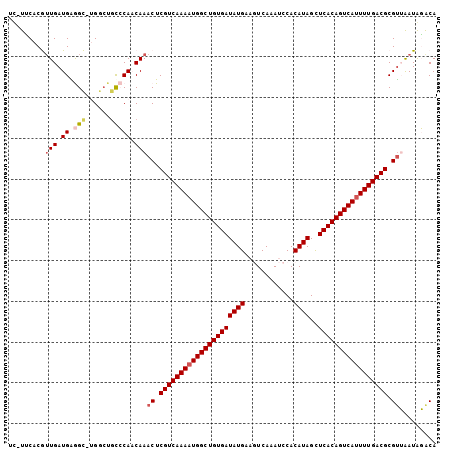
| Structure conservation index | 0.77 |
| SVM decision value | 1.66 |
| SVM RNA-class probability | 0.970454 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
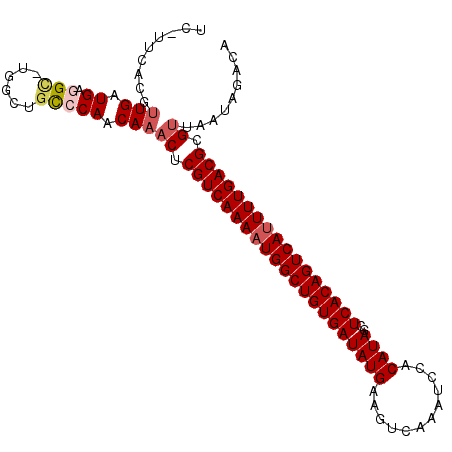
>X_DroMel_CAF1 8936446 104 - 22224390 AUUUCCACGUUGAUGAGGC-UGGCUGUCCAACAAACUCGUCAAAAUGGCUGUGAUAUGAAGUCAAAUCCACAUAGCUCACAGUCAUUUUGACGCGUUAAUAGACA ....(((.(((.....)))-)))..(((.....(((.(((((((((((((((((((((..(......)..))))..))))))))))))))))).)))....))). ( -33.60) >DroPse_CAF1 32058 103 - 1 UC-UGC-CAUUGAUGGAGUGGGACUGCACAUCAAACUCGUCAAAAUGGCUGUGAUAUGAGAUCAAAAACACAUAGCUCACAGUCAUUUUGACGCGUAAAUGUGCA ..-..(-((((.....)))))....((((((...((.(((((((((((((((((((((.(........).))))..))))))))))))))))).))..)))))). ( -40.80) >DroSec_CAF1 994 103 - 1 UU-UUCACGUUGAUGAGGC-UGGCUGCCCAACAAACUCGUCAAAAUGGCUGUGAUAUGAAGUCAAAUCCACAUAGCUCACAGUCAUUUUGACGCGUUGAUAGACA ..-.....((((....(((-.....))))))).(((.(((((((((((((((((((((..(......)..))))..))))))))))))))))).)))........ ( -35.70) >DroSim_CAF1 485 103 - 1 UU-UUCACGUUGAUGAGGC-UGGCUGCCCAACAAACUCGUCAAAAUGGCUGUGAUAUGAAGUCAAAUCCACAUAGCUCACAGUCAUUUUGACGCGUUGAUAGACA ..-.....((((....(((-.....))))))).(((.(((((((((((((((((((((..(......)..))))..))))))))))))))))).)))........ ( -35.70) >DroEre_CAF1 775 99 - 1 AC-UUCACGAUGAUGGGGC-UG----CCCAACAAACUCGUCAAAAUGGCUGUGAUAUGAAGUCAAAUCCACAUAGUUCACAGUCACUUUGACGCGCUAAUAGACA ..-.......((.((((..-..----)))).))..(.(((((((.(((((((((((((..(......)..))))..))))))))).))))))).).......... ( -28.70) >DroPer_CAF1 39923 103 - 1 UC-UGC-CAUUGAUGGAGUGGGACUGCACAUCAAACUCGUCAAAAUGGCUGUGAUAUGAGAUCAAAAACACAUAGCUCACAGUCAUUUUGACGCGUAAAUGUGCA ..-..(-((((.....)))))....((((((...((.(((((((((((((((((((((.(........).))))..))))))))))))))))).))..)))))). ( -40.80) >consensus UC_UUCACGUUGAUGAGGC_UGGCUGCCCAACAAACUCGUCAAAAUGGCUGUGAUAUGAAGUCAAAUCCACAUAGCUCACAGUCAUUUUGACGCGUUAAUAGACA .........(((.((.(((......))))).)))((.(((((((((((((((((((((............))))..))))))))))))))))).))......... (-27.57 = -28.27 + 0.69)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:57:00 2006