
| Sequence ID | X_DroMel_CAF1 |
|---|---|
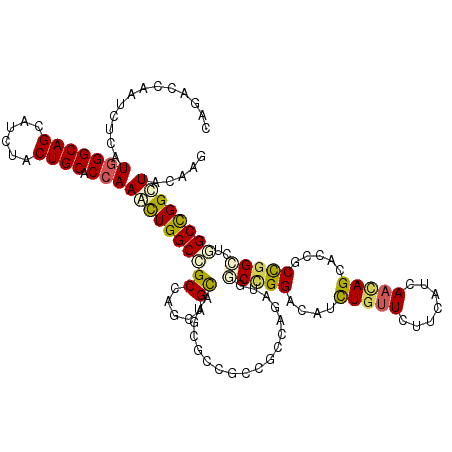
| Location | 8,818,993 – 8,819,153 |
| Length | 160 |
| Max. P | 0.741626 |

| Location | 8,818,993 – 8,819,113 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.44 |
| Mean single sequence MFE | -46.26 |
| Consensus MFE | -26.16 |
| Energy contribution | -26.13 |
| Covariance contribution | -0.02 |
| Combinations/Pair | 1.37 |
| Mean z-score | -2.26 |
| Structure conservation index | 0.57 |
| SVM decision value | -0.01 |
| SVM RNA-class probability | 0.529337 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8818993 120 - 22224390 CAGACGAAUCUCAUGGGCAGCAUUUACUGCACCAAAUUGGCCGCCAGCAGCAUGAGACGCCGCCAGGUGGCUGGACAUCUGAUCUUUGUGAAUAGCACGGCCGGAGUGGCCGGUUAUAAG ...(((((..((((((((((......)))).)))..(..((((((.((.((.......)).))..))))))..).....)))..)))))..(((((.(((((.....))))))))))... ( -44.40) >DroVir_CAF1 91057 120 - 1 CAGACCAAUCUGAUGGGCAGCAUCUACUGCACCAAACUGGCGGCCAACAGCAUGCGCCGCCGCCAGAUAGCCGGCCAUCUGUUCUUCAUCAACAGCACCGCCGGCCUGGCUGGCUAUAAU ((((....)))).(((((((......)))).)))....((((((((......)).))))))(((((...((((((...(((((.......)))))....))))))....)))))...... ( -50.40) >DroPse_CAF1 61840 120 - 1 CAAACGAAUCUCAUGGGCAGCAUCUACUGCACGAAACUGGCCGCUGGCAGCAUGCGGCGUCGCCAGGUCCCAGGACAUUUGUUCUUUGUGAACAGCACCGCCGGUUUGGCCGGCUACAAG ..(((((((.((.(((((.(((.....)))......((((((((((........)))))..)))))).)))).)).)))))))....(((.....))).(((((.....)))))...... ( -41.10) >DroGri_CAF1 64132 120 - 1 CAAACCAACCUAAUGGGCAGCAUCUACUGCACCAAACUGGCGGCAAACAGUAUGCGCCGCCGCCAGAUGGCCGGGCAUUUGUUUUUCAUCAACAGCACCGCUGGCCUUGCCGGUUAUAAU ..((((.......(((((((......)))).)))..((((((((..............))))))))..(((.((((..(((........)))((((...)))))))).)))))))..... ( -44.94) >DroMoj_CAF1 95437 120 - 1 CAGACCAACCUAAUGGGCAGCAUCUACUGCACCAAGCUGGCCGCCAGCAGCAUGCGUCGCCGCCAAAUGGCUGGCCAUCUGUUCUUCAUCAACAGCACCGCCGGCCUGGCUGGCUACAAU ..(((((......(((((((......)))).))).(((((...)))))....)).)))(((((((...(((((((...(((((.......)))))....))))))))))).)))...... ( -47.60) >DroAna_CAF1 48309 120 - 1 CAGACGAAUCUCAUGGGCAGCAUCUACUGCACCAAGCUGGCGGCGAGCAGCAUGCGCCGGCGGCAGGUGCCCGGACACCUAUUCUUCGUGAACAGCACGGCCGGGGUGGCCGGCUACAAG ...(((((.....(((((((......)))).))).((((.(((((.........))))).))))(((((......)))))....)))))....(((.(((((.....))))))))..... ( -49.10) >consensus CAGACCAAUCUCAUGGGCAGCAUCUACUGCACCAAACUGGCCGCCAGCAGCAUGCGCCGCCGCCAGAUGGCCGGACAUCUGUUCUUCAUCAACAGCACCGCCGGCCUGGCCGGCUACAAG .............(((((((......)))).)))((((((((((.....))..................(((((....(((((.......))))).....)))))..))))))))..... (-26.16 = -26.13 + -0.02)


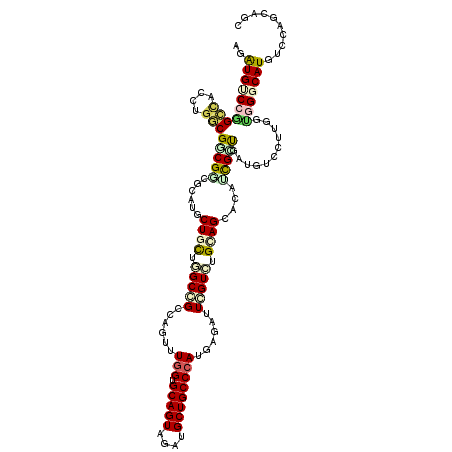
| Location | 8,819,033 – 8,819,153 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.50 |
| Mean single sequence MFE | -50.55 |
| Consensus MFE | -27.83 |
| Energy contribution | -28.45 |
| Covariance contribution | 0.62 |
| Combinations/Pair | 1.50 |
| Mean z-score | -2.06 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.741626 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8819033 120 + 22224390 AGAUGUCCAGCCACCUGGCGGCGUCUCAUGCUGCUGGCGGCCAAUUUGGUGCAGUAAAUGCUGCCCAUGAGAUUCGUCUGUAGUAUGUUGUUGAUGUCCUUGGUGGGCAUAUCGAUUAGC .(((((....(((((.((.((((((.((((((((.(((((......(((.(((((....))))))))......))))).)))))))).....)))))))).)))))..)))))....... ( -48.40) >DroVir_CAF1 91097 120 + 1 AGAUGGCCGGCUAUCUGGCGGCGGCGCAUGCUGUUGGCCGCCAGUUUGGUGCAGUAGAUGCUGCCCAUCAGAUUGGUCUGGAGCACAUCGCUGAUAUCCUUGAUAGGCAUGUCCAGCAAA .((((.((((((((((((.((((((.((......))))))))....(((.(((((....))))))))))))).))).))))....))))(((((((((((....))).)))).))))... ( -53.20) >DroPse_CAF1 61880 120 + 1 AAAUGUCCUGGGACCUGGCGACGCCGCAUGCUGCCAGCGGCCAGUUUCGUGCAGUAGAUGCUGCCCAUGAGAUUCGUUUGCAGGACAUUGCCGAUCUCCUGGGUGGGCAUAUCGAUCAGC .(((((((((.((....((((.(((((.((....)))))))..((((((((((((....)))))..))))))))))))).)))))))))(((.(((.....))).)))............ ( -45.00) >DroGri_CAF1 64172 120 + 1 AAAUGCCCGGCCAUCUGGCGGCGGCGCAUACUGUUUGCCGCCAGUUUGGUGCAGUAGAUGCUGCCCAUUAGGUUGGUUUGCAGCACAUCGCUGAUGUCCUUGACAGGCAUGUCCAGUAAG ..((((((((((..((((((((((.((.....))))))))))))..(((.(((((....))))))))...))))).....((((.....)))).((((...))))))))).......... ( -47.60) >DroMoj_CAF1 95477 120 + 1 AGAUGGCCAGCCAUUUGGCGGCGACGCAUGCUGCUGGCGGCCAGCUUGGUGCAGUAGAUGCUGCCCAUUAGGUUGGUCUGGAGCACAUCGGUGAUGUCCUUGGUGGGCAUGUCCAGCAGA .((((((((((......(((....))).....))))))(((((((((((.(((((....))))))))...)))))))).......))))((..((((((.....))))))..))...... ( -55.30) >DroAna_CAF1 48349 120 + 1 AGGUGUCCGGGCACCUGCCGCCGGCGCAUGCUGCUCGCCGCCAGCUUGGUGCAGUAGAUGCUGCCCAUGAGAUUCGUCUGCAGGGUGCUGGCGAUGUCUUUUGUGGGCAUGUCGAUCAGC ((((((....)))))).....((((((((.((((((((((......))))).)))))))))(((((((((((((((((.((.....)).))))).)))))..))))))).))))...... ( -53.80) >consensus AGAUGUCCGGCCACCUGGCGGCGGCGCAUGCUGCUGGCCGCCAGUUUGGUGCAGUAGAUGCUGCCCAUGAGAUUCGUCUGCAGCACAUCGCUGAUGUCCUUGGUGGGCAUGUCCAGCAGC ..((((((((((....)))(((((......((((.(((((......(((.(((((....))))))))......))))).))))....)))))...........))))))).......... (-27.83 = -28.45 + 0.62)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:55:58 2006