
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 8,440,163 – 8,440,323 |
| Length | 160 |
| Max. P | 0.579348 |

| Location | 8,440,163 – 8,440,283 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.22 |
| Mean single sequence MFE | -42.96 |
| Consensus MFE | -29.25 |
| Energy contribution | -27.28 |
| Covariance contribution | -1.96 |
| Combinations/Pair | 1.57 |
| Mean z-score | -1.42 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.579348 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
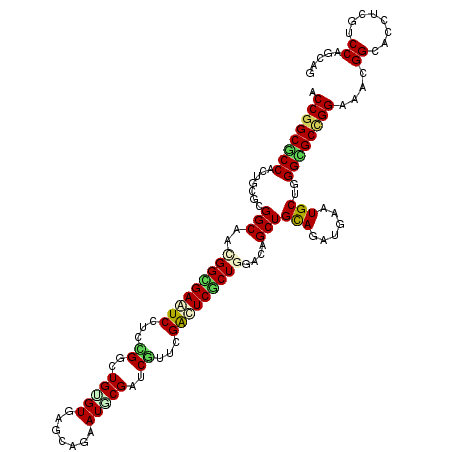
>X_DroMel_CAF1 8440163 120 - 22224390 GUGAGCAGUAUGCGAUCGUUGGACUCGCUGGACAGCUGCAGGUGAAUGCUGGGCGCCGGAAACGGUACUUCGUCCAGCAGCUGGGUGAUGUAGCGCUCCACGGGAAUGGGUAUAUUAACG ((((((.((..((((((....)).))))...)).((((((......((((((((((((....))).....))))))))).........))))))))).)))(..((((....))))..). ( -45.76) >DroVir_CAF1 1210 120 - 1 GUGAGCAGAAUACGAUCAUUCGACUCGCUGGACAGCUGUAGAUGUAUGCUAGGCGCCGGGAACGGAACCUCGUCCAGCAGCUGGAUAAUGUAACGCUCCACGGGUAUUGGUAUAUUGACG (((((..((((......))))..))))).........((....(((((((((((.((((((.((..((.(..(((((...)))))..).))..)).))).))))).)))))))))..)). ( -37.80) >DroGri_CAF1 58567 120 - 1 GUGAGCAAAAUGCGAUCAUUUGAUUCGCUGGACAGCUGCAAAUGGAUGCUGGGUGCCGGGAACGGCACCUCGUCCAGCAGCUGUAUAAUAUACCGUUCUACGGGUAUCGGAAUAUUGCCG (((((..(((((....)))))..))))).((((((((((...((((((..((((((((....)))))))))))))))))))))).((((((.(((.((....))...))).)))))))). ( -55.40) >DroWil_CAF1 1226 120 - 1 GUUAACAAUAUUCGAUCCUUUGGUUCGCUCGAUAGCUGUAGAUGAAUGCUGGGUGCCGGAAAUGGCACCUCAUCCAACAGUUGGGUUAUAUAACGUUCAACCGGAAUGGGUAUAUUUACG .......(((((((.(((...((((.((....(((((((.(((((......(((((((....))))))))))))..))))))).(((....)))))..))))))).)))))))....... ( -32.90) >DroMoj_CAF1 90854 120 - 1 GUGAGCAGAAUUCGAUCGUUCGACUCACUCGACAGCUGCAGGUGUAUGCUGGGCGCUGGGAAGGGCACCUCGUCCAGCAGCUGGAUAAUGUAACGCUCCACGGGUAUCGGUAUAUUGACG (((((..((((......))))..)))))....(((((((....)))(((((((((..((........)).)))))))))))))..(((((((.((..((...))...)).)))))))... ( -39.10) >DroAna_CAF1 1491 118 - 1 GUGAGCAGGAUGCGGUCGUUCGACUCGCUGGACAGCUGCAGGUGAAUGCUCGGAGCCGGAAACGGCACUUCGUCCAGUAGCUGCGUAAUGUAGCGCUCCACGGGAAUGGGGAUAUUCC-- ..(((((...((((((.(((((......))))).))))))......)))))(((((((....)))).((((((((.((.(..((((......))))..))).)).))))))....)))-- ( -46.80) >consensus GUGAGCAGAAUGCGAUCGUUCGACUCGCUGGACAGCUGCAGAUGAAUGCUGGGCGCCGGAAACGGCACCUCGUCCAGCAGCUGGAUAAUGUAACGCUCCACGGGAAUGGGUAUAUUGACG ((((((......(((((....)).))).....(((((((...((.(((..(((.((((....)))).)))))).)))))))))...........))).)))................... (-29.25 = -27.28 + -1.96)



| Location | 8,440,203 – 8,440,323 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.72 |
| Mean single sequence MFE | -50.77 |
| Consensus MFE | -35.40 |
| Energy contribution | -35.18 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.41 |
| Mean z-score | -1.36 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.541268 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8440203 120 - 22224390 ACCGGCGCCACUUCGCGGCAGCGGGGAAUCUUCCGGCUGUGUGAGCAGUAUGCGAUCGUUGGACUCGCUGGACAGCUGCAGGUGAAUGCUGGGCGCCGGAAACGGUACUUCGUCCAGCAG .....((((...(((((.((((.((((...)))).)))))))))(((((..((((((....)).))))......))))).))))..((((((((((((....))).....))))))))). ( -54.70) >DroVir_CAF1 1250 120 - 1 ACCGGCGCCACUGCGCGGCAAUGGCGAAUCCUCUGGCUGUGUGAGCAGAAUACGAUCAUUCGACUCGCUGGACAGCUGUAGAUGUAUGCUAGGCGCCGGGAACGGAACCUCGUCCAGCAG .((((((((..(((...))).(((((.(((..(.(((((((((((..((((......))))..)))))...)))))))..)))...)))))))))))))....(((......)))..... ( -46.50) >DroGri_CAF1 58607 120 - 1 ACCGGCGCCGCUGCGUGGCAACGGCGAAUCCUCUGGCUGUGUGAGCAAAAUGCGAUCAUUUGAUUCGCUGGACAGCUGCAAAUGGAUGCUGGGUGCCGGGAACGGCACCUCGUCCAGCAG .(((((((((.(((...))).))))(((((...((..(((((.......)))))..))...))))))))))....((((...((((((..((((((((....)))))))))))))))))) ( -57.00) >DroWil_CAF1 1266 120 - 1 ACCAGCACCACUUCGUGGCAAUGGUGAAUCUUCCGGUUGGGUUAACAAUAUUCGAUCCUUUGGUUCGCUCGAUAGCUGUAGAUGAAUGCUGGGUGCCGGAAAUGGCACCUCAUCCAACAG ..((((.((((...))))....((((((((....((((((((.......))))))))....)))))))).....))))..(((((......(((((((....))))))))))))...... ( -38.00) >DroMoj_CAF1 90894 120 - 1 GCCGGCGCCGCUGCGCGGCAGUGGUGAGUCCUCUGGCUGCGUGAGCAGAAUUCGAUCGUUCGACUCACUCGACAGCUGCAGGUGUAUGCUGGGCGCUGGGAAGGGCACCUCGUCCAGCAG .(((((((((((((((.(((((((((((((......((((....)))).....((....)))))))))).....)))))..))))).))..))))))))...((((.....))))..... ( -56.90) >DroAna_CAF1 1529 120 - 1 GCCGGCACCACUCCUCGGCAGCGGCGAGUCCUCCGGCUGGGUGAGCAGGAUGCGGUCGUUCGACUCGCUGGACAGCUGCAGGUGAAUGCUCGGAGCCGGAAACGGCACUUCGUCCAGUAG .((((((...(.(((((((..(((((((((...((((((.((.......)).))))))...)))))))))....)))).))).)..))).))).((((....)))).............. ( -51.50) >consensus ACCGGCGCCACUGCGCGGCAACGGCGAAUCCUCCGGCUGUGUGAGCAGAAUGCGAUCGUUCGACUCGCUGGACAGCUGCAGAUGAAUGCUGGGCGCCGGAAACGGCACCUCGUCCAGCAG .((((((((.......(((..(((((((((...((..(((((.......)))))..))...)))))))))....)))(((......)))..))))))))....((........))..... (-35.40 = -35.18 + -0.22)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:53:04 2006