
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 8,402,586 – 8,402,946 |
| Length | 360 |
| Max. P | 0.999919 |

| Location | 8,402,586 – 8,402,706 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.00 |
| Mean single sequence MFE | -44.46 |
| Consensus MFE | -38.06 |
| Energy contribution | -38.38 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.64 |
| Structure conservation index | 0.86 |
| SVM decision value | 1.19 |
| SVM RNA-class probability | 0.928340 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8402586 120 + 22224390 CAGUACGCACCUCGAGGAACUCAUCCUCUAGGCCAUGUAUAAGGGCGCCACAGGCGCCGCUGGCUAUAAUUGAGAUGCUCUGAGCAUCGAGGUGCUCUUGCGGCGCAUCGUCCGCCCACC ......((((((((((((.....)))))..(((((.((.....((((((...)))))))))))))........(((((.....))))))))))))....((((((...)).))))..... ( -51.40) >DroSec_CAF1 1792 120 + 1 CAGUACGCACCUCCAGGAACUCAUCUUCUAGACCAUGUAUAAGGGCGCCACAGGCGCCGCUGGCUAUAAUUGAGAUGCUCUGAGCAUCGAGGUGCUCUUGCGGAGCAUCGUCUGCCCACC (((.(((...((((.....((((.....(((.(((.((.....((((((...))))))))))))))....))))..((...((((((....))))))..))))))...))))))...... ( -42.10) >DroSim_CAF1 5593 120 + 1 CAGUACGCACCUCCAGGAACUCAUCCUCUAGGCCAUGUAUAAGGGCGCCACAGGCGCCGCUGGCUAUAAUUGAGAUGCUCUGAGCAUCGAGGUGCUCUUGCGGCGCAUCGUCCGCCCACC ......(((((((.((((.....))))...(((((.((.....((((((...)))))))))))))........(((((.....))))))))))))....((((((...)).))))..... ( -46.50) >DroEre_CAF1 5239 120 + 1 CAGUACGCACCUCCAGGAACUCAUCCUCCAGGCCAUGGAUAAGAGCGCCGCAGGCUCCGCUGGCUAUUAUCGAGAUGCUCCGAGCAUCUAGAUGCUCUUGCGGCGCAUCGUCUGCCCACC (((.(((..(((..((((.....))))..)))............((((((((((((.....))))..((((.((((((.....)))))).))))....))))))))..))))))...... ( -41.20) >DroYak_CAF1 4877 120 + 1 CAGUACGCACCUCCAGGAACUCAUCCUCCAGGCCAUGGAUAAGGGCGCCACAGGCGCCGCUGGCUAUAAUCGAAAUGCUCCGGGCAUCUAGGUGCUCUUGUGGCGCAUCGUCCGCCCACC ......((.(((..((((.....))))..)))....((((....((((((((((..(((..(((............))).)))((((....)))).))))))))))...))))))..... ( -41.10) >consensus CAGUACGCACCUCCAGGAACUCAUCCUCUAGGCCAUGUAUAAGGGCGCCACAGGCGCCGCUGGCUAUAAUUGAGAUGCUCUGAGCAUCGAGGUGCUCUUGCGGCGCAUCGUCCGCCCACC ..(((((..(((..((((.....))))..)))...)))))..(((((......(((((((.(((............)))..((((((....))))))..)))))))......)))))... (-38.06 = -38.38 + 0.32)

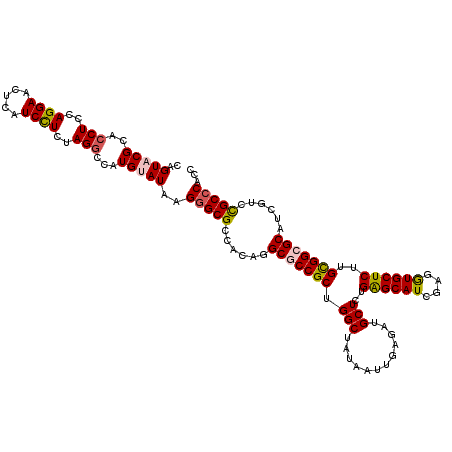
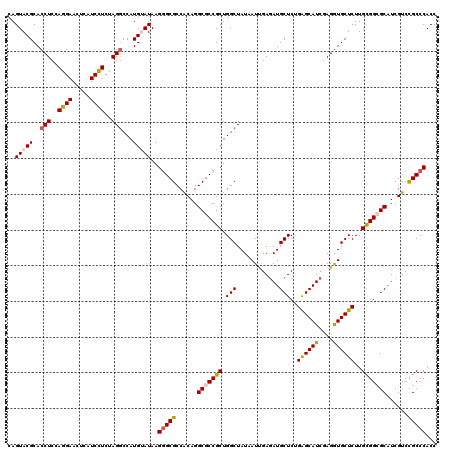
| Location | 8,402,626 – 8,402,746 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.17 |
| Mean single sequence MFE | -54.32 |
| Consensus MFE | -51.94 |
| Energy contribution | -51.26 |
| Covariance contribution | -0.68 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.60 |
| Structure conservation index | 0.96 |
| SVM decision value | 2.20 |
| SVM RNA-class probability | 0.990179 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8402626 120 - 22224390 GCUCGCCUGGUGGCCAGCGGUGAGUGGUCAUCGGGUAAACGGUGGGCGGACGAUGCGCCGCAAGAGCACCUCGAUGCUCAGAGCAUCUCAAUUAUAGCCAGCGGCGCCUGUGGCGCCCUU ....((((((((((((.(.....))))))))))))).......(((((..((..(((((((..(((...)))(((((.....))))).............)))))))...)).))))).. ( -54.40) >DroSec_CAF1 1832 120 - 1 GCUCGCCUGGUGGCCAGCGGAGAGUGGUCAUCGGGUAAACGGUGGGCAGACGAUGCUCCGCAAGAGCACCUCGAUGCUCAGAGCAUCUCAAUUAUAGCCAGCGGCGCCUGUGGCGCCCUU ((((((((((((((((.(.....)))))))))))))......((((((..(((((((.(....)))))..))).))))))))))...............((.((((((...)))))))). ( -54.40) >DroSim_CAF1 5633 120 - 1 GCUCGCCUGGUGGCCAGCGGAGAGUGGUCAUCGGGUAAACGGUGGGCGGACGAUGCGCCGCAAGAGCACCUCGAUGCUCAGAGCAUCUCAAUUAUAGCCAGCGGCGCCUGUGGCGCCCUU ....((((((((((((.(.....))))))))))))).......(((((..((..(((((((..(((...)))(((((.....))))).............)))))))...)).))))).. ( -54.40) >DroEre_CAF1 5279 120 - 1 GCUCGCCUGGUGGCCAGCGGUGAGUGGUCAUCGGGUAAACGGUGGGCAGACGAUGCGCCGCAAGAGCAUCUAGAUGCUCGGAGCAUCUCGAUAAUAGCCAGCGGAGCCUGCGGCGCUCUU ....((((((((((((.(.....))))))))))))).....(((((((.....))).))))(((((((((.((((((.....)))))).)))....(((.((((...))))))))))))) ( -55.20) >DroYak_CAF1 4917 120 - 1 GCUCGCCUGGUGGCCAGCGGUGAGUGGUCAUCGGGCAAACGGUGGGCGGACGAUGCGCCACAAGAGCACCUAGAUGCCCGGAGCAUUUCGAUUAUAGCCAGCGGCGCCUGUGGCGCCCUU ((((((((((((((((.(.....))))))))))))).....(((((((.....))).))))..))))..(.((((((.....)))))).).........((.((((((...)))))))). ( -53.20) >consensus GCUCGCCUGGUGGCCAGCGGUGAGUGGUCAUCGGGUAAACGGUGGGCGGACGAUGCGCCGCAAGAGCACCUCGAUGCUCAGAGCAUCUCAAUUAUAGCCAGCGGCGCCUGUGGCGCCCUU ((((((((((((((((.(.....))))))))))))).....(((((((.....))).))))..)))).....(((((.....)))))............((.((((((...)))))))). (-51.94 = -51.26 + -0.68)

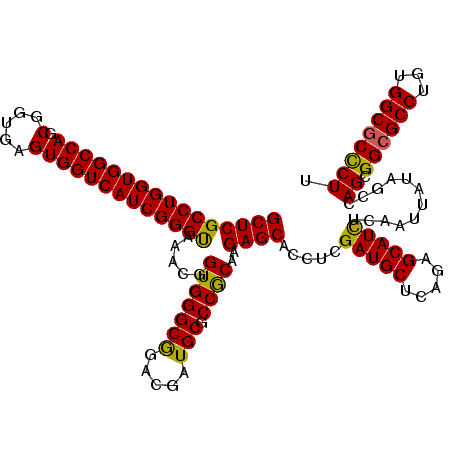
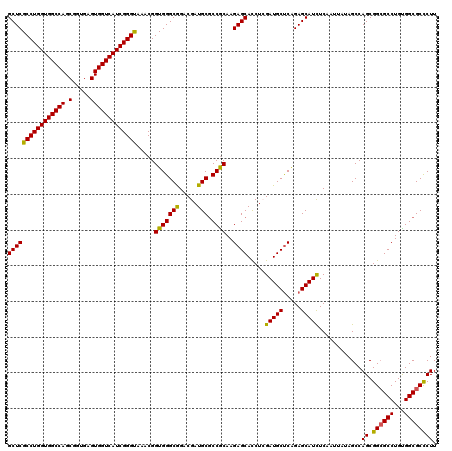
| Location | 8,402,666 – 8,402,786 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.08 |
| Mean single sequence MFE | -49.15 |
| Consensus MFE | -46.00 |
| Energy contribution | -46.48 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.06 |
| Structure conservation index | 0.94 |
| SVM decision value | 3.32 |
| SVM RNA-class probability | 0.999001 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8402666 120 + 22224390 UGAGCAUCGAGGUGCUCUUGCGGCGCAUCGUCCGCCCACCGUUUACCCGAUGACCACUCACCGCUGGCCACCAGGCGAGCACCUCUCUCGUGCGCCGUUCGCUUCCGGCGCAGAUUGCUC .((((((((((((((((..((((.((((((.................)))))......).))))..(((....)))))))))))).....(((((((........))))))))).))))) ( -50.83) >DroSec_CAF1 1872 120 + 1 UGAGCAUCGAGGUGCUCUUGCGGAGCAUCGUCUGCCCACCGUUUACCCGAUGACCACUCUCCGCUGGCCACCAGGCGAGCACCUCUCUCGUGCGCCGUCCGCUUUCGGCGCAGAUUGCUC .((((((((((((((((..(((((((((((...((.....)).....)))))......))))))..(((....)))))))))))).....(((((((........))))))))).))))) ( -56.00) >DroSim_CAF1 5673 120 + 1 UGAGCAUCGAGGUGCUCUUGCGGCGCAUCGUCCGCCCACCGUUUACCCGAUGACCACUCUCCGCUGGCCACCAGGCGAGCACCUCUCUCGUGCGCCGUCCGCUUCCGGCGGAGAUUGCUC .((((((((.((((..(.((.((((.......))))))..)..))))))))......((((((((((....(.((((.((((.......))))..)))).)...))))))))))..)))) ( -47.50) >DroEre_CAF1 5319 120 + 1 CGAGCAUCUAGAUGCUCUUGCGGCGCAUCGUCUGCCCACCGUUUACCCGAUGACCACUCACCGCUGGCCACCAGGCGAGCACCUCUCUCGUGCGCCGUCCGCUUCCGGCGCAGAUUGCUC .((((((((((.(((((..((((.((((((...((.....)).....)))))......).))))..(((....)))))))).)).......((((((........))))))))).))))) ( -40.80) >DroYak_CAF1 4957 120 + 1 CGGGCAUCUAGGUGCUCUUGUGGCGCAUCGUCCGCCCACCGUUUGCCCGAUGACCACUCACCGCUGGCCACCAGGCGAGCACCUCUCUCGUGCGCCGUCCGCUUGCGGCGCAGAUUGCUC .((((((((((((((((..((((.((.......)))))).....(((.(.((.(((........))).)).).))))))))))).......(((((((......)))))))))).))))) ( -50.60) >consensus UGAGCAUCGAGGUGCUCUUGCGGCGCAUCGUCCGCCCACCGUUUACCCGAUGACCACUCACCGCUGGCCACCAGGCGAGCACCUCUCUCGUGCGCCGUCCGCUUCCGGCGCAGAUUGCUC .((((((((((((((((..((((.((((((...((.....)).....)))))......).))))..(((....)))))))))))).....(((((((........))))))))).))))) (-46.00 = -46.48 + 0.48)

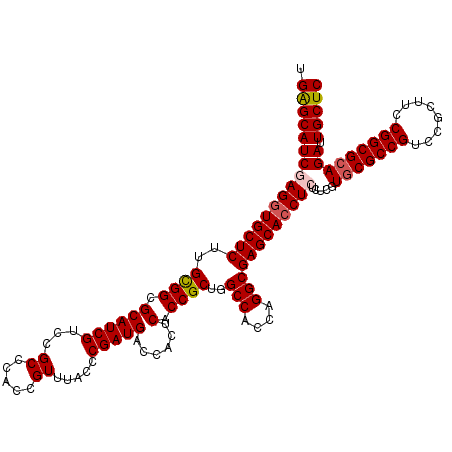

| Location | 8,402,666 – 8,402,786 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.08 |
| Mean single sequence MFE | -66.64 |
| Consensus MFE | -65.58 |
| Energy contribution | -65.66 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.09 |
| Mean z-score | -4.92 |
| Structure conservation index | 0.98 |
| SVM decision value | 4.55 |
| SVM RNA-class probability | 0.999919 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8402666 120 - 22224390 GAGCAAUCUGCGCCGGAAGCGAACGGCGCACGAGAGAGGUGCUCGCCUGGUGGCCAGCGGUGAGUGGUCAUCGGGUAAACGGUGGGCGGACGAUGCGCCGCAAGAGCACCUCGAUGCUCA (((((.(((((((((........))))))).))..(((((((((((((((((((((.(.....))))))))))))).....(((((((.....))).))))..)))))))))..))))). ( -69.70) >DroSec_CAF1 1872 120 - 1 GAGCAAUCUGCGCCGAAAGCGGACGGCGCACGAGAGAGGUGCUCGCCUGGUGGCCAGCGGAGAGUGGUCAUCGGGUAAACGGUGGGCAGACGAUGCUCCGCAAGAGCACCUCGAUGCUCA (((((.(((((((((........))))))).))..(((((((((((((((((((((.(.....))))))))))))).....(((((((.....))).))))..)))))))))..))))). ( -69.30) >DroSim_CAF1 5673 120 - 1 GAGCAAUCUCCGCCGGAAGCGGACGGCGCACGAGAGAGGUGCUCGCCUGGUGGCCAGCGGAGAGUGGUCAUCGGGUAAACGGUGGGCGGACGAUGCGCCGCAAGAGCACCUCGAUGCUCA (((((.(((((((((........)))))...))))(((((((((((((((((((((.(.....))))))))))))).....(((((((.....))).))))..)))))))))..))))). ( -67.20) >DroEre_CAF1 5319 120 - 1 GAGCAAUCUGCGCCGGAAGCGGACGGCGCACGAGAGAGGUGCUCGCCUGGUGGCCAGCGGUGAGUGGUCAUCGGGUAAACGGUGGGCAGACGAUGCGCCGCAAGAGCAUCUAGAUGCUCG (((((.(((((((((........))))))).))...((((((((((((((((((((.(.....))))))))))))).....(((((((.....))).))))..))))))))...))))). ( -64.30) >DroYak_CAF1 4957 120 - 1 GAGCAAUCUGCGCCGCAAGCGGACGGCGCACGAGAGAGGUGCUCGCCUGGUGGCCAGCGGUGAGUGGUCAUCGGGCAAACGGUGGGCGGACGAUGCGCCACAAGAGCACCUAGAUGCCCG (.(((.(((((((((........))))))).))...((((((((((((((((((((.(.....))))))))))))).....(((((((.....))).))))..))))))))...))).). ( -62.70) >consensus GAGCAAUCUGCGCCGGAAGCGGACGGCGCACGAGAGAGGUGCUCGCCUGGUGGCCAGCGGUGAGUGGUCAUCGGGUAAACGGUGGGCGGACGAUGCGCCGCAAGAGCACCUCGAUGCUCA (((((.(((((((((........))))))).))..(((((((((((((((((((((.(.....))))))))))))).....(((((((.....))).))))..)))))))))..))))). (-65.58 = -65.66 + 0.08)

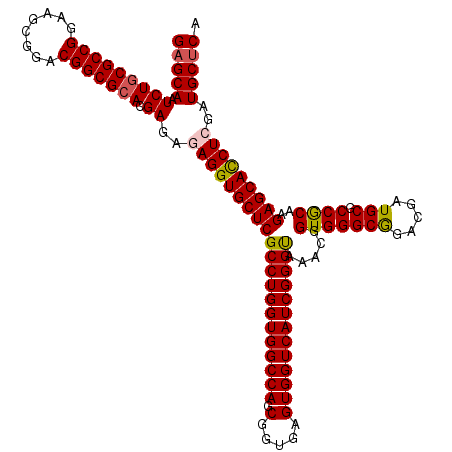

| Location | 8,402,706 – 8,402,826 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.33 |
| Mean single sequence MFE | -42.30 |
| Consensus MFE | -37.82 |
| Energy contribution | -38.42 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.32 |
| Structure conservation index | 0.89 |
| SVM decision value | 0.41 |
| SVM RNA-class probability | 0.726409 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8402706 120 + 22224390 GUUUACCCGAUGACCACUCACCGCUGGCCACCAGGCGAGCACCUCUCUCGUGCGCCGUUCGCUUCCGGCGCAGAUUGCUCAUCAGCUUUUUGUCGAGUGUCUGGUGUAGGAACUCCCUGC ....(((....(((.((((((.(((((((....)))(((((......((.(((((((........))))))))).)))))..)))).....)).))))))).)))(((((.....))))) ( -41.90) >DroSec_CAF1 1912 120 + 1 GUUUACCCGAUGACCACUCUCCGCUGGCCACCAGGCGAGCACCUCUCUCGUGCGCCGUCCGCUUUCGGCGCAGAUUGCUCAUCAGCUUUUUGUCGAGUGUCUGGUGUAGGAACUCCCUGC ....(((....(((.((((..((((((((....)))(((((......((.(((((((........))))))))).)))))..)))).....)..))))))).)))(((((.....))))) ( -41.70) >DroSim_CAF1 5713 120 + 1 GUUUACCCGAUGACCACUCUCCGCUGGCCACCAGGCGAGCACCUCUCUCGUGCGCCGUCCGCUUCCGGCGGAGAUUGCUCAUCAGCUUUUUGUCGAGUGUCUGGUGUAGGAACUCCCUGC ....(((.(((((.((.((((((((((....(.((((.((((.......))))..)))).)...)))))))))).)).))))).((((......))))....)))(((((.....))))) ( -45.50) >DroEre_CAF1 5359 120 + 1 GUUUACCCGAUGACCACUCACCGCUGGCCACCAGGCGAGCACCUCUCUCGUGCGCCGUCCGCUUCCGGCGCAGAUUGCUCAUCAGCUUUUUGUCUAGUGUCUGGUGCAGAAACUCCCUGC ........(((((.((.((...(((.(((....))).)))..........(((((((........))))))))).)).)))))((.(((((((((((...)))).)))))))))...... ( -38.90) >DroYak_CAF1 4997 120 + 1 GUUUGCCCGAUGACCACUCACCGCUGGCCACCAGGCGAGCACCUCUCUCGUGCGCCGUCCGCUUGCGGCGCAGAUUGCUCAUCAGCUUUUUGUCCAGUGUCUGGUGCAGAAACUCCCUGC ....((..(((((.((.((...(((.(((....))).)))..........((((((((......)))))))))).)).))))).))(((((((((((...)))).)))))))........ ( -43.50) >consensus GUUUACCCGAUGACCACUCACCGCUGGCCACCAGGCGAGCACCUCUCUCGUGCGCCGUCCGCUUCCGGCGCAGAUUGCUCAUCAGCUUUUUGUCGAGUGUCUGGUGUAGGAACUCCCUGC ....(((.(((((.((.((...(((.(((....))).)))..........(((((((........))))))))).)).))))).((((......))))....)))(((((.....))))) (-37.82 = -38.42 + 0.60)

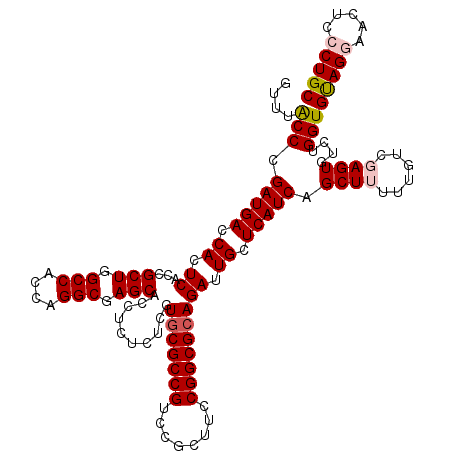

| Location | 8,402,706 – 8,402,826 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.33 |
| Mean single sequence MFE | -47.72 |
| Consensus MFE | -43.90 |
| Energy contribution | -43.70 |
| Covariance contribution | -0.20 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.92 |
| SVM decision value | 2.02 |
| SVM RNA-class probability | 0.985694 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8402706 120 - 22224390 GCAGGGAGUUCCUACACCAGACACUCGACAAAAAGCUGAUGAGCAAUCUGCGCCGGAAGCGAACGGCGCACGAGAGAGGUGCUCGCCUGGUGGCCAGCGGUGAGUGGUCAUCGGGUAAAC .....((((.(((.(....)...((((.......(((....)))....(((((((........)))))))))))..))).))))((((((((((((.(.....))))))))))))).... ( -47.60) >DroSec_CAF1 1912 120 - 1 GCAGGGAGUUCCUACACCAGACACUCGACAAAAAGCUGAUGAGCAAUCUGCGCCGAAAGCGGACGGCGCACGAGAGAGGUGCUCGCCUGGUGGCCAGCGGAGAGUGGUCAUCGGGUAAAC .....((((.(((.(....)...((((.......(((....)))....(((((((........)))))))))))..))).))))((((((((((((.(.....))))))))))))).... ( -46.30) >DroSim_CAF1 5713 120 - 1 GCAGGGAGUUCCUACACCAGACACUCGACAAAAAGCUGAUGAGCAAUCUCCGCCGGAAGCGGACGGCGCACGAGAGAGGUGCUCGCCUGGUGGCCAGCGGAGAGUGGUCAUCGGGUAAAC .....((((..((.....))..)))).........(((((((.((.(((((((.((.(.(((.((((((.(....)..)))).)).))).)..)).))))))).)).)))))))...... ( -46.30) >DroEre_CAF1 5359 120 - 1 GCAGGGAGUUUCUGCACCAGACACUAGACAAAAAGCUGAUGAGCAAUCUGCGCCGGAAGCGGACGGCGCACGAGAGAGGUGCUCGCCUGGUGGCCAGCGGUGAGUGGUCAUCGGGUAAAC (((((.....)))))....((((((.........(((....)))..(((((((((........))))))).))....)))).))((((((((((((.(.....))))))))))))).... ( -47.00) >DroYak_CAF1 4997 120 - 1 GCAGGGAGUUUCUGCACCAGACACUGGACAAAAAGCUGAUGAGCAAUCUGCGCCGCAAGCGGACGGCGCACGAGAGAGGUGCUCGCCUGGUGGCCAGCGGUGAGUGGUCAUCGGGCAAAC (((((.....))))).((((...)))).............(((((.(((((((((........)))))).(....)))))))))((((((((((((.(.....))))))))))))).... ( -51.40) >consensus GCAGGGAGUUCCUACACCAGACACUCGACAAAAAGCUGAUGAGCAAUCUGCGCCGGAAGCGGACGGCGCACGAGAGAGGUGCUCGCCUGGUGGCCAGCGGUGAGUGGUCAUCGGGUAAAC .....((((.(((.....................(((....)))..(((((((((........))))))).))...))).))))((((((((((((.(.....))))))))))))).... (-43.90 = -43.70 + -0.20)


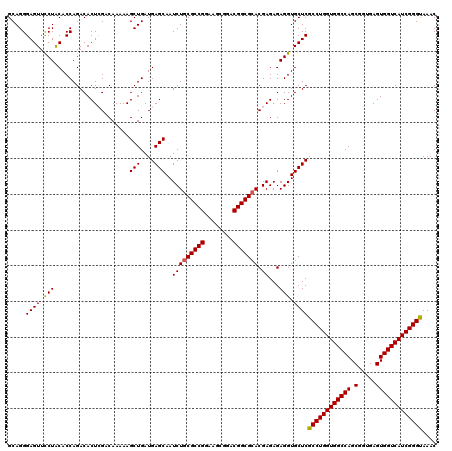
| Location | 8,402,746 – 8,402,866 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.33 |
| Mean single sequence MFE | -42.61 |
| Consensus MFE | -37.54 |
| Energy contribution | -37.86 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.42 |
| Structure conservation index | 0.88 |
| SVM decision value | 0.72 |
| SVM RNA-class probability | 0.831792 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8402746 120 + 22224390 ACCUCUCUCGUGCGCCGUUCGCUUCCGGCGCAGAUUGCUCAUCAGCUUUUUGUCGAGUGUCUGGUGUAGGAACUCCCUGCUUACGGCGGUCAUCCCACCGAGCAAUUCCGCUGCGAGCAC ......(((((((((((........)))))).((((((((....((((......))))....((((..(((...(((((....))).))...))))))))))))))).....)))))... ( -41.40) >DroSec_CAF1 1952 120 + 1 ACCUCUCUCGUGCGCCGUCCGCUUUCGGCGCAGAUUGCUCAUCAGCUUUUUGUCGAGUGUCUGGUGUAGGAACUCCCUGCUUACGGCGGUCAUCCCACCCAGCAGUUCCGCUGCGAGCAC .......((.(((((((........))))))))).(((((..............((.((.(((..(((((.....)))))...))))).))..........((((.....))))))))). ( -41.10) >DroSim_CAF1 5753 120 + 1 ACCUCUCUCGUGCGCCGUCCGCUUCCGGCGGAGAUUGCUCAUCAGCUUUUUGUCGAGUGUCUGGUGUAGGAACUCCCUGCUUACGGCGGUCAUCCCACCCAGCAGUUCCGCUGCGAGCAC .......((.(((((((..((((..(((((((((..(((....))))))))))))))))..))))))).))......((((..((((((..................))))))..)))). ( -41.77) >DroEre_CAF1 5399 120 + 1 ACCUCUCUCGUGCGCCGUCCGCUUCCGGCGCAGAUUGCUCAUCAGCUUUUUGUCUAGUGUCUGGUGCAGAAACUCCCUGCUGACGGCGGUCAUCCCACCCAGCAGCUCCGCUGCGAGCAC .......((.(((((((........))))))))).(((((..(((((((((((((((...)))).)))))))....((((((..((.((.....)).))))))))....)))).))))). ( -43.50) >DroYak_CAF1 5037 120 + 1 ACCUCUCUCGUGCGCCGUCCGCUUGCGGCGCAGAUUGCUCAUCAGCUUUUUGUCCAGUGUCUGGUGCAGAAACUCCCUGCUUACGGCGGUCAUUCCACCCAGCAGCUCCGCUGCGAGCAC .......((.((((((((......)))))))))).(((((..(((((((((((((((...)))).)))))))....(((((...((.((.....)).)).)))))....)))).))))). ( -45.30) >consensus ACCUCUCUCGUGCGCCGUCCGCUUCCGGCGCAGAUUGCUCAUCAGCUUUUUGUCGAGUGUCUGGUGUAGGAACUCCCUGCUUACGGCGGUCAUCCCACCCAGCAGUUCCGCUGCGAGCAC .......((.(((((((........))))))))).(((((..((((........((((.((((...)))).)))).(((((...((.((.....)).)).)))))....)))).))))). (-37.54 = -37.86 + 0.32)


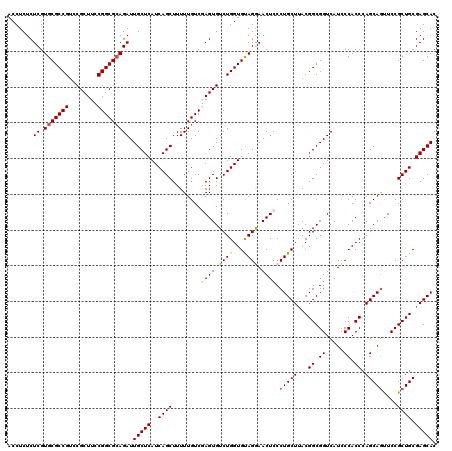
| Location | 8,402,746 – 8,402,866 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.33 |
| Mean single sequence MFE | -49.44 |
| Consensus MFE | -46.44 |
| Energy contribution | -46.24 |
| Covariance contribution | -0.20 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.23 |
| Structure conservation index | 0.94 |
| SVM decision value | 3.75 |
| SVM RNA-class probability | 0.999581 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8402746 120 - 22224390 GUGCUCGCAGCGGAAUUGCUCGGUGGGAUGACCGCCGUAAGCAGGGAGUUCCUACACCAGACACUCGACAAAAAGCUGAUGAGCAAUCUGCGCCGGAAGCGAACGGCGCACGAGAGAGGU .((((((((((....(((((((((((.....))))))..))))).((((..((.....))..))))........)))).)))))).(((((((((........))))))).))....... ( -49.20) >DroSec_CAF1 1952 120 - 1 GUGCUCGCAGCGGAACUGCUGGGUGGGAUGACCGCCGUAAGCAGGGAGUUCCUACACCAGACACUCGACAAAAAGCUGAUGAGCAAUCUGCGCCGAAAGCGGACGGCGCACGAGAGAGGU .((((((((((....(((((.(((((.....)))))...))))).((((..((.....))..))))........)))).)))))).(((((((((........))))))).))....... ( -49.20) >DroSim_CAF1 5753 120 - 1 GUGCUCGCAGCGGAACUGCUGGGUGGGAUGACCGCCGUAAGCAGGGAGUUCCUACACCAGACACUCGACAAAAAGCUGAUGAGCAAUCUCCGCCGGAAGCGGACGGCGCACGAGAGAGGU .((((((((((....(((((.(((((.....)))))...))))).((((..((.....))..))))........)))).)))))).(((((((((........)))))...))))..... ( -48.00) >DroEre_CAF1 5399 120 - 1 GUGCUCGCAGCGGAGCUGCUGGGUGGGAUGACCGCCGUCAGCAGGGAGUUUCUGCACCAGACACUAGACAAAAAGCUGAUGAGCAAUCUGCGCCGGAAGCGGACGGCGCACGAGAGAGGU .((((((((((....(((((((((((.....)))))..))))))..(((.((((...)))).))).........)))).)))))).(((((((((........))))))).))....... ( -52.50) >DroYak_CAF1 5037 120 - 1 GUGCUCGCAGCGGAGCUGCUGGGUGGAAUGACCGCCGUAAGCAGGGAGUUUCUGCACCAGACACUGGACAAAAAGCUGAUGAGCAAUCUGCGCCGCAAGCGGACGGCGCACGAGAGAGGU .((((((((((....(((((.(((((.....)))))...)))))..(((.((((...)))).))).........)))).)))))).(((((((((........))))))).))....... ( -48.30) >consensus GUGCUCGCAGCGGAACUGCUGGGUGGGAUGACCGCCGUAAGCAGGGAGUUCCUACACCAGACACUCGACAAAAAGCUGAUGAGCAAUCUGCGCCGGAAGCGGACGGCGCACGAGAGAGGU .((((((((((....(((((.(((((.....)))))...)))))((.((....)).))................)))).)))))).(((((((((........))))))).))....... (-46.44 = -46.24 + -0.20)

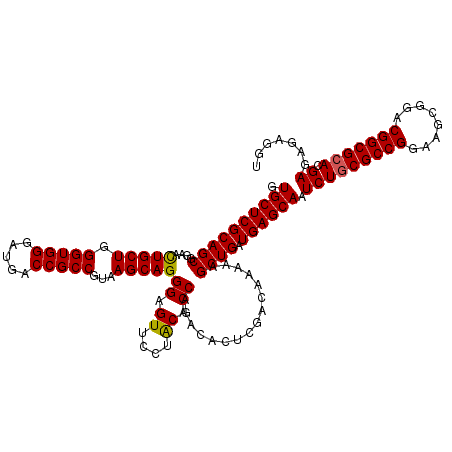
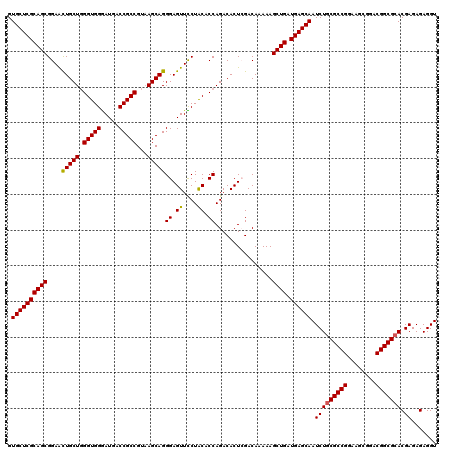
| Location | 8,402,826 – 8,402,946 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.08 |
| Mean single sequence MFE | -49.26 |
| Consensus MFE | -45.12 |
| Energy contribution | -44.80 |
| Covariance contribution | -0.32 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.41 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.921894 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8402826 120 - 22224390 AGCAGGAGGAGCUGCGCAUGAUAGACGCUGCGUUCAGCAAGGUCUGCGAAGCCCUGUGCGAUCUAUCGCUGCAAAUUCGCGUGCUCGCAGCGGAAUUGCUCGGUGGGAUGACCGCCGUAA (((((.....(((((((((...((((..(((.....)))..))))(((((....(((((((....)))).)))..)))))))))..)))))....)))))((((((.....))))))... ( -48.00) >DroSec_CAF1 2032 120 - 1 AGCAGGAGGAGCUGCGCAUGAUAGACGCUGCGUUCAGCAAGGUCUGCGAAGCCCUGUGCGAUCUAUCGCUGCAAAUUCGCGUGCUCGCAGCGGAACUGCUGGGUGGGAUGACCGCCGUAA (((((.....(((((((((...((((..(((.....)))..))))(((((....(((((((....)))).)))..)))))))))..)))))....))))).(((((.....))))).... ( -49.40) >DroSim_CAF1 5833 120 - 1 AGCAGGAGGAGCUGCGCAUGAUAGACGCUGCGUUCAGCAAGGUCUGCGAAGCCCUGUGCGAUCUAUCGCUGCAAAUUCGCGUGCUCGCAGCGGAACUGCUGGGUGGGAUGACCGCCGUAA (((((.....(((((((((...((((..(((.....)))..))))(((((....(((((((....)))).)))..)))))))))..)))))....))))).(((((.....))))).... ( -49.40) >DroEre_CAF1 5479 120 - 1 AGCAGGAGGAGCUCCGCAUGAUAGAUGCUGCCUUCAGCAAGGUCUGCGAAGCCCUGUGCGAUCUUUCGCUGCAGAUACGUGUGCUCGCAGCGGAGCUGCUGGGUGGGAUGACCGCCGUCA ......((.((((((((..(((...(((((....)))))..)))(((((.(((((((((((....)))).))))......).))))))))))))))).)).(((((.....))))).... ( -51.60) >DroYak_CAF1 5117 120 - 1 AGCAGGAGGAGCUGCGCAUGAUAGAUGCUGCCUUUAGCAAGGUCUGCGAAGCACUGUGUGAUCUCUCGCUGCAGAUUCGCGUGCUCGCAGCGGAGCUGCUGGGUGGAAUGACCGCCGUAA .((...((.((((.(((..(((...(((((....)))))..)))(((((.((((.(((.((((((.....).)))))))))))))))))))).)))).)).(((((.....))))))).. ( -47.90) >consensus AGCAGGAGGAGCUGCGCAUGAUAGACGCUGCGUUCAGCAAGGUCUGCGAAGCCCUGUGCGAUCUAUCGCUGCAAAUUCGCGUGCUCGCAGCGGAACUGCUGGGUGGGAUGACCGCCGUAA (((((.....(((((((((...((((..(((.....)))..))))(((((....(((((((....)))).)))..)))))))))..)))))....))))).(((((.....))))).... (-45.12 = -44.80 + -0.32)


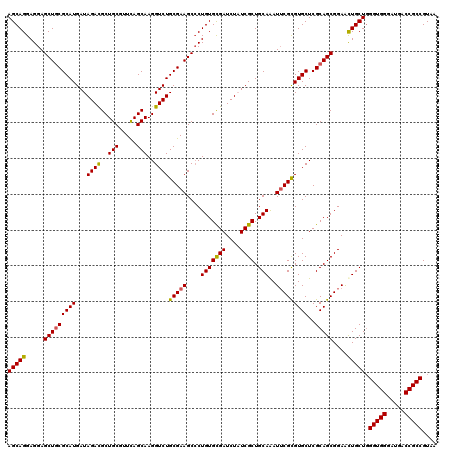
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:52:42 2006