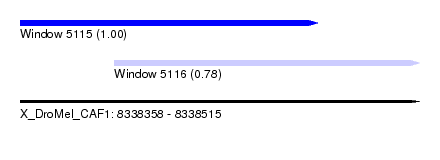
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 8,338,358 – 8,338,515 |
| Length | 157 |
| Max. P | 0.998646 |

| Location | 8,338,358 – 8,338,475 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.30 |
| Mean single sequence MFE | -31.56 |
| Consensus MFE | -28.76 |
| Energy contribution | -29.04 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.11 |
| Structure conservation index | 0.91 |
| SVM decision value | 3.17 |
| SVM RNA-class probability | 0.998646 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8338358 117 + 22224390 --U-ACAGCUAUGCUAUUAAUCGCACCCAAGCUGUAUGGUCGAGUCACGUCAUUGGUGCAUAUUGAGCAACAUAAUCGAUAUCUUCUAUAGGUUAUGACUUGCCCAAUGAAUCGUGAAUU --(-((((((.(((........)))....)))))))........(((((((((((((((.......))).(((((((.(((.....))).)))))))......)))))))..)))))... ( -31.90) >DroSec_CAF1 3221 119 + 1 CCU-GCAGCUAUGCUAUAAACCGCACCCAAGCUGUAUGGUCGAAUUACGUCAUUGGUGCAUAUUGAGCAACAUAAUCGAUAUCUUCUAUAGGUUAUGACUUGCCCAAUGAAUCGUGAAUU (((-((((((.(((........)))....))))))).)).....(((((((((((((((.......))).(((((((.(((.....))).)))))))......)))))))..)))))... ( -31.90) >DroSim_CAF1 3302 119 + 1 CCU-GCAGCUAUGCUAGUAACCGCACCCAAGCUGUAUGGUCGUAUCACGUCAUUGGUGCAUAUUGAGCAACAUAAUCGAUAUCUUCUAUAGGUUAUGACUUACCCAAUGAAUCGUGAAUU (((-((((((.(((........)))....))))))).)).....(((((((((((((((.......))).(((((((.(((.....))).)))))))......)))))))..)))))... ( -34.10) >DroEre_CAF1 1355 119 + 1 CCU-GCAGCUAUGCUAUUAACUGCAACCAAGCUGUAUGAUCGAAUCACGUCAUUGGUGCAUAUUGAGCAACAUAAUCGAUAUCUUCUAUAGGUUAUGACUUGCCCAAUGAAUCGUGCAUU ..(-((((((.(((........)))....))))))).........((((((((((((((.......))).(((((((.(((.....))).)))))))......)))))))..)))).... ( -30.20) >DroYak_CAF1 1946 120 + 1 CCUGGCAGCAAUGCUAUUAACUGCACCCAAGCUGUAUGGUCGAAUCACGUCAUUGGUGCAUAUUGAGCAACAUAAUCGAUAUCUUCUAUAGGUUAUGACUUGCCCAAUGAAUCGUGCAUU ..(((((....))))).....(((((....((...((((.((.....))))))....)).(((((.(((((((((((.(((.....))).)))))))..)))).)))))....))))).. ( -29.70) >consensus CCU_GCAGCUAUGCUAUUAACCGCACCCAAGCUGUAUGGUCGAAUCACGUCAUUGGUGCAUAUUGAGCAACAUAAUCGAUAUCUUCUAUAGGUUAUGACUUGCCCAAUGAAUCGUGAAUU ....((((((.(((........)))....)))))).........(((((((((((((((.......))).(((((((.(((.....))).)))))))......)))))))..)))))... (-28.76 = -29.04 + 0.28)



| Location | 8,338,395 – 8,338,515 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.17 |
| Mean single sequence MFE | -28.38 |
| Consensus MFE | -23.88 |
| Energy contribution | -25.56 |
| Covariance contribution | 1.68 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.48 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.779545 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8338395 120 + 22224390 CGAGUCACGUCAUUGGUGCAUAUUGAGCAACAUAAUCGAUAUCUUCUAUAGGUUAUGACUUGCCCAAUGAAUCGUGAAUUGAAUAACUUAGCCUAGUAACAUUUAGUUUGAUGUUAUUGG (((.(((((((((((((((.......))).(((((((.(((.....))).)))))))......)))))))..))))).)))...........(((((((((((......))))))))))) ( -33.30) >DroSec_CAF1 3260 120 + 1 CGAAUUACGUCAUUGGUGCAUAUUGAGCAACAUAAUCGAUAUCUUCUAUAGGUUAUGACUUGCCCAAUGAAUCGUGAAUUAAAUAACUUAGCCUAGUAACAUUCAGUUGGAUGUUGUUGG ....(((((((((((((((.......))).(((((((.(((.....))).)))))))......)))))))..)))))...............((((((((((((....)))))))))))) ( -28.90) >DroSim_CAF1 3341 120 + 1 CGUAUCACGUCAUUGGUGCAUAUUGAGCAACAUAAUCGAUAUCUUCUAUAGGUUAUGACUUACCCAAUGAAUCGUGAAUUGAAUAACUUAGCCUAGUAACAUUUAGUUUGUUGUUGUUGG ....(((((((((((((((.......))).(((((((.(((.....))).)))))))......)))))))..)))))....((((((.(((.((((......)))).)))..)))))).. ( -27.30) >DroEre_CAF1 1394 120 + 1 CGAAUCACGUCAUUGGUGCAUAUUGAGCAACAUAAUCGAUAUCUUCUAUAGGUUAUGACUUGCCCAAUGAAUCGUGCAUUGAAUAACUUAACCUAGUAACAUUUAGUUUGAAAUUGUUGG (((..((((((((((((((.......))).(((((((.(((.....))).)))))))......)))))))..))))..)))(((((.((((.((((......)))).))))..))))).. ( -23.20) >DroYak_CAF1 1986 120 + 1 CGAAUCACGUCAUUGGUGCAUAUUGAGCAACAUAAUCGAUAUCUUCUAUAGGUUAUGACUUGCCCAAUGAAUCGUGCAUUGAAUAACUCAACCUAGUAACAUUUAGUUGGUUAUUGUCGG (((..((((((((((((((.......))).(((((((.(((.....))).)))))))......)))))))..)))).....((((((.((((.(((......))))))))))))).))). ( -29.20) >consensus CGAAUCACGUCAUUGGUGCAUAUUGAGCAACAUAAUCGAUAUCUUCUAUAGGUUAUGACUUGCCCAAUGAAUCGUGAAUUGAAUAACUUAGCCUAGUAACAUUUAGUUUGAUGUUGUUGG (((.(((((((((((((((.......))).(((((((.(((.....))).)))))))......)))))))..))))).)))...........(((((((((((......))))))))))) (-23.88 = -25.56 + 1.68)

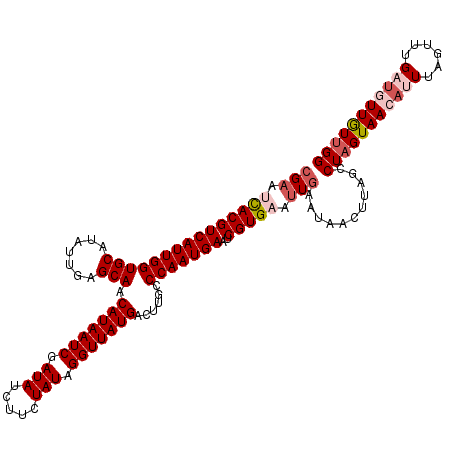
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:51:38 2006