
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 8,328,147 – 8,328,243 |
| Length | 96 |
| Max. P | 0.580453 |

| Location | 8,328,147 – 8,328,243 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 96 |
| Reading direction | forward |
| Mean pairwise identity | 80.11 |
| Mean single sequence MFE | -38.55 |
| Consensus MFE | -23.01 |
| Energy contribution | -22.68 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.67 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.580453 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
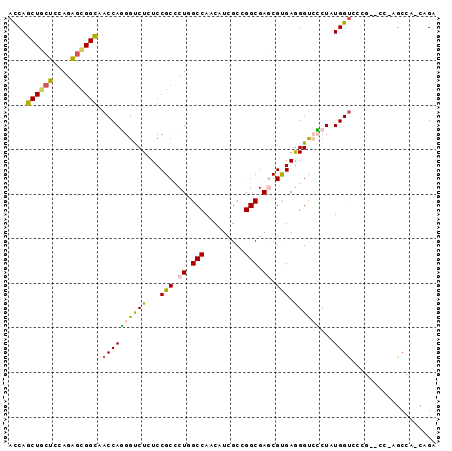
>X_DroMel_CAF1 8328147 96 + 22224390 AUCAGCUGCUCCAGAGCGGCAACCAGGGUCUGUCCGCUCUUGCCAACAUCGCCGGCGAGCGUGAGGGUCCCUAUGGUCCCGCUCCAAGCCAUCAGC ....((((((...((((((..(((((((.((.(((((((..(((.........)))))))).)).)).)))..)))).))))))..)))....))) ( -40.00) >DroVir_CAF1 734 84 + 1 ACCAGCUGCUCCAGGGUGGCAACCAUGGUCUCUCCGCUCUGGCCAACAUUGCCGGCGAGCGUGAAGGUAACUAUGGUA---------GC---CAGA (((..(((...))))))(((.(((((((((..(((((((((((.......))))..))))).))..)..)))))))).---------))---)... ( -32.10) >DroSec_CAF1 308 96 + 1 AUCAGCUGCUCCAGAGCGGCAACCAGGGUCUGUCCGCUCUCGCCAACAUUGCCGGCGAGCGUGAGGGUCCCUAUGGUCCCGCUCCCAGCCAUCAGC ....((((.....((((((..(((((((.((.(((((((..(((.........)))))))).)).)).)))..)))).)))))).))))....... ( -40.50) >DroEre_CAF1 716 96 + 1 ACCAGCUGCUCCAGAGCGGUAACCAGGGUCUGUCCGCCCUCGCCAGCAUCGCCGGCGAGCGUGAGGGUCCCUAUGGUCCUGGCCCAAGCCAGCAGC ((((((((((....))))))....((((.((.(((((..(((((.((...)).)))))))).)).)).)))).)))).(((((....))))).... ( -43.40) >DroMoj_CAF1 722 84 + 1 ACCAGCUGCUCCAGGGCGGCAACCAUGGUCUCUCCGCCCUGGCCAACAUUGCUGGCGACCGCGAGGGCAGCUAUGGCG---------GC---CAGA ....(((((.(((((((((..((....))....)))))))))....(((.((((....((....)).)))).))))))---------))---.... ( -39.10) >DroAna_CAF1 743 96 + 1 ACCAGCUCCUCCAGAACGGCAACCAGGGCCUCUCCGCCCUGGCCAGCAUCGCCGGCGAUCGUGAGGGUCCCUAUGGUGCCGGACCUAGCCACCAGA .................(((..(((((((......)))))))((.((((((..((.((((.....))))))..)))))).)).....)))...... ( -36.20) >consensus ACCAGCUGCUCCAGAGCGGCAACCAGGGUCUCUCCGCCCUGGCCAACAUCGCCGGCGAGCGUGAGGGUCCCUAUGGUCCCG__CC_AGCCA_CAGA ....((((((....)))))).((((((((((...(((.((.(((.........))).)).))).))))))...))))................... (-23.01 = -22.68 + -0.33)


| Location | 8,328,147 – 8,328,243 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 96 |
| Reading direction | reverse |
| Mean pairwise identity | 80.11 |
| Mean single sequence MFE | -42.02 |
| Consensus MFE | -26.05 |
| Energy contribution | -25.97 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.30 |
| Mean z-score | -1.43 |
| Structure conservation index | 0.62 |
| SVM decision value | -0.07 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
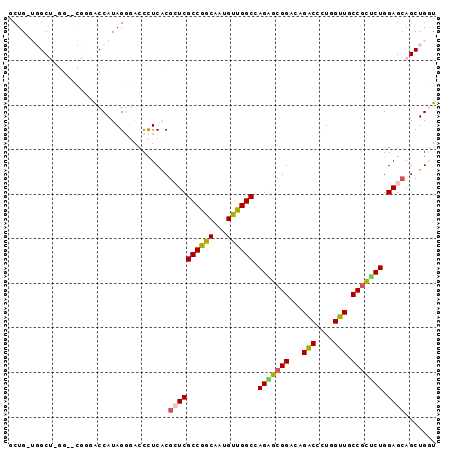
>X_DroMel_CAF1 8328147 96 - 22224390 GCUGAUGGCUUGGAGCGGGACCAUAGGGACCCUCACGCUCGCCGGCGAUGUUGGCAAGAGCGGACAGACCCUGGUUGCCGCUCUGGAGCAGCUGAU ((((.....(..(((((((((((..(((.(..((.(((((((((((...))))))..)))))))..).)))))))).))))))..)..)))).... ( -44.90) >DroVir_CAF1 734 84 - 1 UCUG---GC---------UACCAUAGUUACCUUCACGCUCGCCGGCAAUGUUGGCCAGAGCGGAGAGACCAUGGUUGCCACCCUGGAGCAGCUGGU ..((---((---------.(((((.((...(((..(((((((((((...))))))..)))))..))))).))))).))))..(..(.....)..). ( -31.30) >DroSec_CAF1 308 96 - 1 GCUGAUGGCUGGGAGCGGGACCAUAGGGACCCUCACGCUCGCCGGCAAUGUUGGCGAGAGCGGACAGACCCUGGUUGCCGCUCUGGAGCAGCUGAU ((((....(..(.((((((((((..(((.(..((.(((((((((((...))))))..)))))))..).)))))))).))))))..)..)))).... ( -44.30) >DroEre_CAF1 716 96 - 1 GCUGCUGGCUUGGGCCAGGACCAUAGGGACCCUCACGCUCGCCGGCGAUGCUGGCGAGGGCGGACAGACCCUGGUUACCGCUCUGGAGCAGCUGGU ((((((..((.((((...(((((..(((.(..((.(.(((((((((...))))))))).)..))..).))))))))...)))).)))))))).... ( -44.50) >DroMoj_CAF1 722 84 - 1 UCUG---GC---------CGCCAUAGCUGCCCUCGCGGUCGCCAGCAAUGUUGGCCAGGGCGGAGAGACCAUGGUUGCCGCCCUGGAGCAGCUGGU ...(---((---------(((.............))))))((((((..(((...(((((((((...(((....))).))))))))).))))))))) ( -43.92) >DroAna_CAF1 743 96 - 1 UCUGGUGGCUAGGUCCGGCACCAUAGGGACCCUCACGAUCGCCGGCGAUGCUGGCCAGGGCGGAGAGGCCCUGGUUGCCGUUCUGGAGGAGCUGGU .(..((..(((((..((((((((..(((.(((((.((..(((((((...))))))..)..))))).)))))))).))))).)))))....))..). ( -43.20) >consensus GCUG_UGGCU_GG__CGGGACCAUAGGGACCCUCACGCUCGCCGGCAAUGUUGGCCAGAGCGGACAGACCCUGGUUGCCGCUCUGGAGCAGCUGGU ....................................((((((((((...)))))).(((((((...(((....))).))))))).))))....... (-26.05 = -25.97 + -0.08)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:51:27 2006