
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 984,669 – 984,842 |
| Length | 173 |
| Max. P | 0.999551 |

| Location | 984,669 – 984,766 |
|---|---|
| Length | 97 |
| Sequences | 4 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 83.23 |
| Mean single sequence MFE | -35.12 |
| Consensus MFE | -26.27 |
| Energy contribution | -25.77 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.67 |
| Structure conservation index | 0.75 |
| SVM decision value | 2.03 |
| SVM RNA-class probability | 0.986049 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 984669 97 + 22224390 GUGGCUGGUGGCUGGUCCCCUUGUUUAUGUGUCUGCUAUAUAUG------------U----AUGUACAUAUGUGAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGCC ..(((.((.(((((((...((((((((((((.(((.((((((((------------(----....)))))))))..))).))))))).)))))..))))))).)).....))) ( -36.00) >DroSec_CAF1 67362 101 + 1 GUGGCUGGUGGCUGGUCCCCUUGUUUAUGUGUCUGCUGUGUAUGUAUG--------U----AUGUACAUAUGAGAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGGC ...((..(((((((((...((((((((((((...(((((((((((((.--------.----..))))))))....)))))))))))).)))))..))))))).....))..)) ( -36.70) >DroSim_CAF1 46630 97 + 1 GUGGCUGGUGGCUGGUCCCCUUGUUUAUGUGUCUGCUGUGUAUG------------U----AUGUACAUAUGCGAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGGC ...((..(((((((((...((((((((((((.(((.((((((((------------(----....)))))))))..))).))))))).)))))..))))))).....))..)) ( -36.10) >DroEre_CAF1 80745 102 + 1 GUG-------GCUGGUCCCCUUGUUUAUGUGUCUGCUAUGGAU----GUGUAUAUGUAUGGAUAUGUGUUUGUGAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGGC (.(-------((((((...((((((((((((.(((.((..(((----(..(((........)))..))))..))..))).))))))).)))))..))))))).)......... ( -31.70) >consensus GUGGCUGGUGGCUGGUCCCCUUGUUUAUGUGUCUGCUAUGUAUG____________U____AUGUACAUAUGUGAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGGC ..........((((((...((((((((((((.(((.((((((((......................))))))))..))).))))))).)))))..))))))............ (-26.27 = -25.77 + -0.50)



| Location | 984,669 – 984,766 |
|---|---|
| Length | 97 |
| Sequences | 4 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 83.23 |
| Mean single sequence MFE | -24.70 |
| Consensus MFE | -18.24 |
| Energy contribution | -18.24 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.68 |
| SVM RNA-class probability | 0.819498 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 984669 97 - 22224390 GGCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUCACAUAUGUACAU----A------------CAUAUAUAGCAGACACAUAAACAAGGGGACCAGCCACCAGCCAC (((...((.(..((((((..((((...((((((.(.(((...((((((....----)------------)))))..))).).))))))..))))..).)))))..)))))).. ( -29.30) >DroSec_CAF1 67362 101 - 1 GCCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUCUCAUAUGUACAU----A--------CAUACAUACACAGCAGACACAUAAACAAGGGGACCAGCCACCAGCCAC .....(((.(..((((((..((((...((((((.(.(((....((((((...----.--------..))))))...))).).))))))..))))..).)))))..)))).... ( -25.10) >DroSim_CAF1 46630 97 - 1 GCCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUCGCAUAUGUACAU----A------------CAUACACAGCAGACACAUAAACAAGGGGACCAGCCACCAGCCAC .....(((.(..((((((..((((...((((((.(.(((.(..(((((....----)------------))))..)))).).))))))..))))..).)))))..)))).... ( -24.80) >DroEre_CAF1 80745 102 - 1 GCCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUCACAAACACAUAUCCAUACAUAUACAC----AUCCAUAGCAGACACAUAAACAAGGGGACCAGC-------CAC ............((((((..((((...((((((...(((....)))..((((......))))....----............))))))..))))..).)))))-------... ( -19.60) >consensus GCCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUCACAUAUGUACAU____A____________CAUACACAGCAGACACAUAAACAAGGGGACCAGCCACCAGCCAC ............((((((..((((...((((((.(.(((.....................................))).).))))))..))))..).))))).......... (-18.24 = -18.24 + 0.00)



| Location | 984,709 – 984,803 |
|---|---|
| Length | 94 |
| Sequences | 4 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 82.41 |
| Mean single sequence MFE | -30.30 |
| Consensus MFE | -20.92 |
| Energy contribution | -22.30 |
| Covariance contribution | 1.38 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.37 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.43 |
| SVM RNA-class probability | 0.953266 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 984709 94 + 22224390 UAUG------------U----AUGUACAUAUGUGAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGCCAACGCACACCGUUGCACACCUUUGUGUGUGCUAGCUU ..((------------(----.(((((.((((((......))))))))))).((((.(((........))).)))))))....(((((((((......)))))).))).. ( -26.60) >DroSec_CAF1 67402 98 + 1 UAUGUAUG--------U----AUGUACAUAUGAGAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGGCAACGCACACCGUUGCACACCUCUGUGUGUGCUAGCUU (((((((.--------.----..))))))).(((...((((((((((.(...((((((((........))))))))(((......)))....).))))))).)))..))) ( -31.40) >DroSim_CAF1 46670 94 + 1 UAUG------------U----AUGUACAUAUGCGAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGGCAACGCACACCGUUGCACACCUCUGUGUGUGCUAGCUU ((((------------(----....))))).((.(((((.......((((..((((((((........)))))))))))).)))))((((((......))))))..)).. ( -30.30) >DroEre_CAF1 80778 101 + 1 GAU----GUGUAUAUGUAUGGAUAUGUGUUUGUGAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGGCAACGCACACCAUUGC-----ACCGUGUGUACUAGCUU ...----((...((..(((((.((((((..((....))..))))))((((..((((((((........))))))))))))........-----.)))))..))...)).. ( -32.90) >consensus UAUG____________U____AUGUACAUAUGUGAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGGCAACGCACACCGUUGCACACCUCUGUGUGUGCUAGCUU ......................(((((.((((((......))))))))))).((((((((........))))))))((((((....(((......)))))))))...... (-20.92 = -22.30 + 1.38)

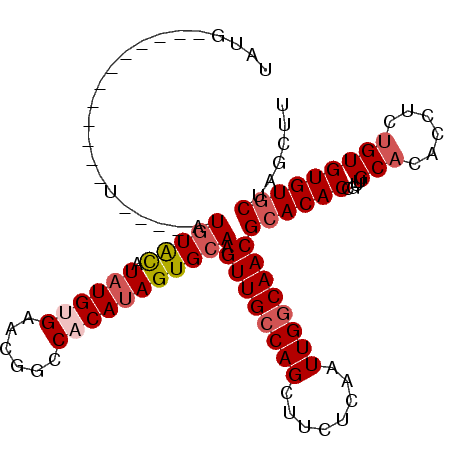
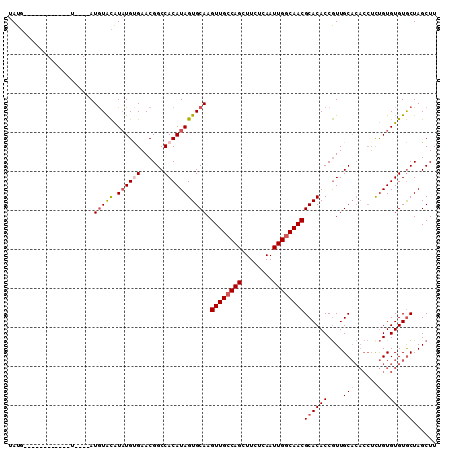
| Location | 984,709 – 984,803 |
|---|---|
| Length | 94 |
| Sequences | 4 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 82.41 |
| Mean single sequence MFE | -30.40 |
| Consensus MFE | -24.51 |
| Energy contribution | -25.57 |
| Covariance contribution | 1.06 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.33 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.73 |
| SVM RNA-class probability | 0.974315 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 984709 94 - 22224390 AAGCUAGCACACACAAAGGUGUGCAACGGUGUGCGUUGGCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUCACAUAUGUACAU----A------------CAUA ..((((((.(((((....)))))(((((.....)))))...........))))))....((((.(((((((....)))))))))))...----.------------.... ( -29.40) >DroSec_CAF1 67402 98 - 1 AAGCUAGCACACACAGAGGUGUGCAACGGUGUGCGUUGCCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUCUCAUAUGUACAU----A--------CAUACAUA ..((((((((((......))))))...((((((.(((((((.((....)).))))))).))))))...))))........((((((...----.--------..)))))) ( -32.50) >DroSim_CAF1 46670 94 - 1 AAGCUAGCACACACAGAGGUGUGCAACGGUGUGCGUUGCCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUCGCAUAUGUACAU----A------------CAUA ..((..((((((......))))))(((((((((((((((((.((....)).)))))))..))))......)))))).)).(((((....----)------------)))) ( -33.40) >DroEre_CAF1 80778 101 - 1 AAGCUAGUACACACGGU-----GCAAUGGUGUGCGUUGCCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUCACAAACACAUAUCCAUACAUAUACAC----AUC ............(((((-----...((((((((.(((((((.((....)).))))))).))))))))...)))))............................----... ( -26.30) >consensus AAGCUAGCACACACAGAGGUGUGCAACGGUGUGCGUUGCCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUCACAUAUGUACAU____A____________CAUA ..((((((((((......))))))...((((((.(((((((.((....)).))))))).))))))...))))...................................... (-24.51 = -25.57 + 1.06)

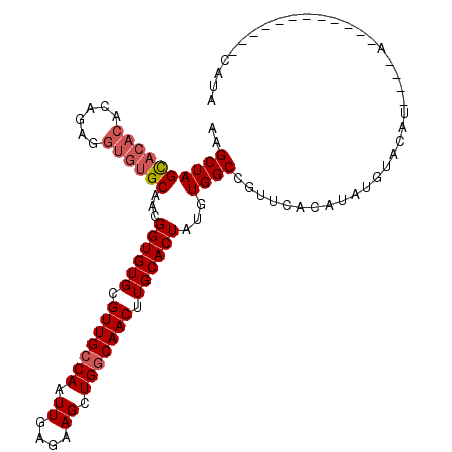
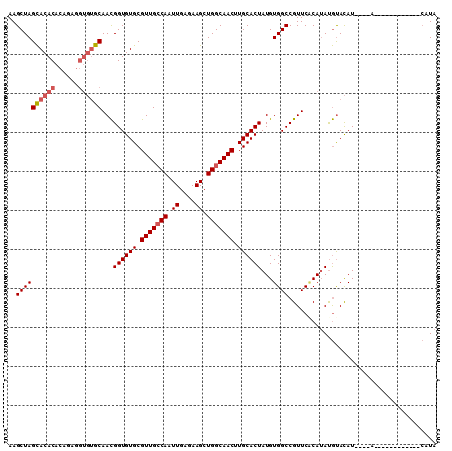
| Location | 984,726 – 984,842 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 93.25 |
| Mean single sequence MFE | -47.20 |
| Consensus MFE | -39.83 |
| Energy contribution | -40.03 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.34 |
| Structure conservation index | 0.84 |
| SVM decision value | 3.71 |
| SVM RNA-class probability | 0.999551 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 984726 116 + 22224390 GAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGCCAACGCACACC---GUUGCACACCUUUGUGUGUGCUAGCUUGUGGCAUAUCUACGGUGAAAUGUCUGCGCGUGUGGCACG ...(((((((((.(((((.((((.(((........))).))))...((((---((.((((((......))))))..((.....))......)))))).......)))))))))))).)) ( -42.70) >DroSec_CAF1 67423 116 + 1 GAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGGCAACGCACACC---GUUGCACACCUCUGUGUGUGCUAGCUUGUGGCAUAUCUACGGUGAAAUGUCUGCGCGUGUGGCACG ...(((((((((.(((((.((((((((........))))))))...((((---((.((((((......))))))..((.....))......)))))).......)))))))))))).)) ( -49.80) >DroSim_CAF1 46687 116 + 1 GAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGGCAACGCACACC---GUUGCACACCUCUGUGUGUGCUAGCUUGUGGCAUAUCUACGGUGAAAUGUCUGCGCGUGUGGCACG ...(((((((((.(((((.((((((((........))))))))...((((---((.((((((......))))))..((.....))......)))))).......)))))))))))).)) ( -49.80) >DroEre_CAF1 80807 111 + 1 GAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGGCAACGCACACC---AUUGC-----ACCGUGUGUACUAGCUUGUGGCAUAUCUACGGUGAAAUGUCUGCGCGUGUGGCACG ...(((((((((.(((((.((((((((........))))))))......(---(((.(-----((((((.(((...((.....)).))).))))))).))))..)))))))))))).)) ( -45.00) >DroYak_CAF1 73017 118 + 1 GAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGGCAACGCACACCACCAUUGCU-AGUACCGUGUGUACUAGCUUAUGGCAUAUCUACGGUGAAAUGUCUGCGCGUGUGGCACG ...(((((((((.(((((.((((((((........))))))))...((((.((((.(((-(((((.....))))))))..)))).........)))).......)))))))))))).)) ( -48.70) >consensus GAACGGCCACAUAGUGCAAGUUGCCAGCUUCUCAAUUGGCAACGCACACC___GUUGCACACCUCUGUGUGUGCUAGCUUGUGGCAUAUCUACGGUGAAAUGUCUGCGCGUGUGGCACG ...(((((((((.(((((.((((((((........))))))))((((((.................))))))..........(((((.((......)).))))))))))))))))).)) (-39.83 = -40.03 + 0.20)



| Location | 984,726 – 984,842 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 93.25 |
| Mean single sequence MFE | -42.89 |
| Consensus MFE | -33.56 |
| Energy contribution | -34.76 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.47 |
| Structure conservation index | 0.78 |
| SVM decision value | 1.54 |
| SVM RNA-class probability | 0.962743 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 984726 116 - 22224390 CGUGCCACACGCGCAGACAUUUCACCGUAGAUAUGCCACAAGCUAGCACACACAAAGGUGUGCAAC---GGUGUGCGUUGGCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUC ((.((((((.(.((((......((((((......((.....))..((((((......)))))).))---))))...((..((.........))..))...)))).)..))))))))... ( -39.80) >DroSec_CAF1 67423 116 - 1 CGUGCCACACGCGCAGACAUUUCACCGUAGAUAUGCCACAAGCUAGCACACACAGAGGUGUGCAAC---GGUGUGCGUUGCCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUC ((.((((((.(.((((......((((((......((.....))..((((((......)))))).))---))))...(((((((.((....)).))))))))))).)..))))))))... ( -43.50) >DroSim_CAF1 46687 116 - 1 CGUGCCACACGCGCAGACAUUUCACCGUAGAUAUGCCACAAGCUAGCACACACAGAGGUGUGCAAC---GGUGUGCGUUGCCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUC ((.((((((.(.((((......((((((......((.....))..((((((......)))))).))---))))...(((((((.((....)).))))))))))).)..))))))))... ( -43.50) >DroEre_CAF1 80807 111 - 1 CGUGCCACACGCGCAGACAUUUCACCGUAGAUAUGCCACAAGCUAGUACACACGGU-----GCAAU---GGUGUGCGUUGCCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUC ((.((((((.(((((..((((.((((((......((.....))........)))))-----).)))---).)))))(((((((.((....)).)))))))........))))))))... ( -41.44) >DroYak_CAF1 73017 118 - 1 CGUGCCACACGCGCAGACAUUUCACCGUAGAUAUGCCAUAAGCUAGUACACACGGUACU-AGCAAUGGUGGUGUGCGUUGCCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUC ((.((((((.(.((((.(((.((((((((....))).....((((((((.....)))))-)))...))))).))))(((((((.((....)).))))))).))).)..))))))))... ( -46.20) >consensus CGUGCCACACGCGCAGACAUUUCACCGUAGAUAUGCCACAAGCUAGCACACACAGAGGUGUGCAAC___GGUGUGCGUUGCCAAUUGAGAAGCUGGCAACUUGCACUAUGUGGCCGUUC .((((.....))))....................((((((.....((((((......))))))......((((((.(((((((.((....)).))))))).)))))).))))))..... (-33.56 = -34.76 + 1.20)

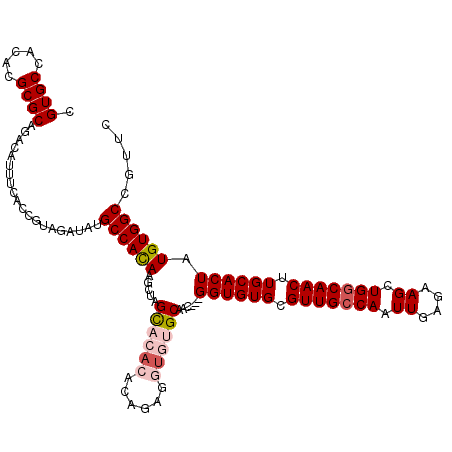
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:41:23 2006