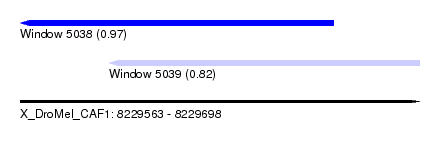
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 8,229,563 – 8,229,698 |
| Length | 135 |
| Max. P | 0.971916 |

| Location | 8,229,563 – 8,229,669 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 78.12 |
| Mean single sequence MFE | -24.98 |
| Consensus MFE | -16.70 |
| Energy contribution | -16.37 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.67 |
| SVM decision value | 1.68 |
| SVM RNA-class probability | 0.971916 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 8229563 106 - 22224390 UAG--CUAAAUGCAUGUGCAUGUGCGCACGA--AUGCAAAAAUUGAUUGUAAUUCAAUUACAGUGCGUAAUUUAUGCAUUGCCAAAAUCGACCACAAGACCU-AGUAAAAC ..(--(((...(((.(((((((((((((((.--...)...((((((.......))))))...)))))))...)))))))))).....((........))..)-)))..... ( -23.20) >DroSec_CAF1 130494 106 - 1 UGG--CUAAAUGCAUGUGCAUGUGCGCACGA--AUGCAAAAAUUGAUUUUAAUUCAAUUACAGUGCGUAAUUUAUGCAUUGCCAAAAGCGACCACAAGCCCU-AGUAAAAC (((--(..(((((((((((......))))..--(((((..((((((.......))))))....))))).....)))))))))))...((........))...-........ ( -26.90) >DroEre_CAF1 119711 104 - 1 UGG--CUAAAUGCAUUC---AGUGCGCACGA--AUGCAAAAAUUGAUUUUAAUUCAAUUACCGUGCGUAAUUUAUGCAUUGCUAAAAGCGACCACAAAACCCGGUGAAUCG (((--(..(((((((..---..((((((((.--.......((((((.......))))))..))))))))....)))))))))))....(((.(((........)))..))) ( -26.70) >DroWil_CAF1 129504 97 - 1 ACU--CUA-CUGUGCGUGUAUGUGUGUAUGGCCAUCCGAAAAUUGGUUUUAAUUCAAUUACAGUGCGUAAUUUAUGCAUUGCUUAGAGC-ACCGCA---CCU-AU------ ...--...-..((((((((...((..((..((((.........))))..))...))....(((((((((...)))))))))......))-).))))---)..-..------ ( -22.80) >DroYak_CAF1 121304 104 - 1 UGG--CUAAAUGCAUUC---AGUGCGCACGA--AUGCAAAAAUUGAUUUUAAUUCAAUUACAGUGCGUAAUUUAUGCAUUGCCAAAAGCGACCACAAAACUCUAGUAAAAU (((--(..(((((((..---..((((((((.--...)...((((((.......))))))...)))))))....)))))))))))........................... ( -26.30) >DroAna_CAF1 182586 97 - 1 GGGCACUAAAUGCAUUC---UGUGCGCACGA--AUGCAAAAAUUGAUUUUAAUUCAAUUACAGUGCGUAAUUUAUGCAUUGCUAAAAUCCACCACACAAUUC--------- (((((((...(((((((---((....)).))--)))))..((((((.......))))))..)))))(((((......))))).........)).........--------- ( -24.00) >consensus UGG__CUAAAUGCAUGC___UGUGCGCACGA__AUGCAAAAAUUGAUUUUAAUUCAAUUACAGUGCGUAAUUUAUGCAUUGCCAAAAGCGACCACAAAACCC_AGUAAAA_ ........(((((((.......((((((((......)...((((((.......))))))...)))))))....)))))))............................... (-16.70 = -16.37 + -0.33)

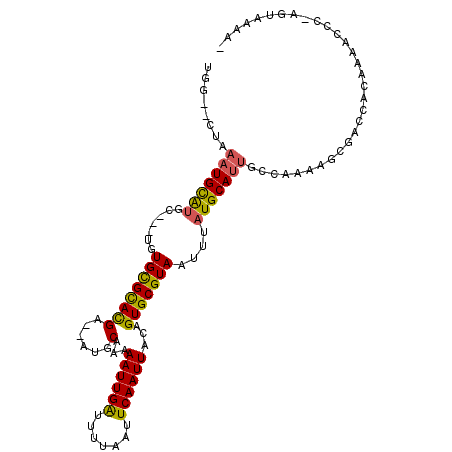
| Location | 8,229,593 – 8,229,698 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.27 |
| Mean single sequence MFE | -28.38 |
| Consensus MFE | -22.32 |
| Energy contribution | -22.10 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.25 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.67 |
| SVM RNA-class probability | 0.819099 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
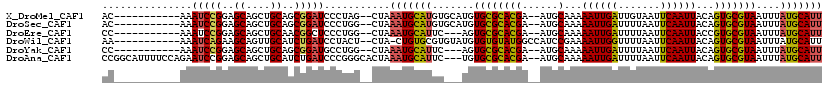
>X_DroMel_CAF1 8229593 105 - 22224390 AC-----------AAAUCCGGAGCAGCUGCAGCGGAUCCCUAG--CUAAAUGCAUGUGCAUGUGCGCACGA--AUGCAAAAAUUGAUUGUAAUUCAAUUACAGUGCGUAAUUUAUGCAUU ..-----------..(((((..((....))..))))).....(--(.....))..(((((((((((((((.--...)...((((((.......))))))...)))))))...))))))). ( -27.80) >DroSec_CAF1 130524 105 - 1 AC-----------AAAUCCGGAGCAGCUGCAGCGGAUCCCUGG--CUAAAUGCAUGUGCAUGUGCGCACGA--AUGCAAAAAUUGAUUUUAAUUCAAUUACAGUGCGUAAUUUAUGCAUU ..-----------..(((((..((....))..))))).....(--(.....))..(((((((((((((((.--...)...((((((.......))))))...)))))))...))))))). ( -27.60) >DroEre_CAF1 119742 102 - 1 CC-----------AAAUCCGGAGCAGCUGCAACGGCUCCCUGG--CUAAAUGCAUUC---AGUGCGCACGA--AUGCAAAAAUUGAUUUUAAUUCAAUUACCGUGCGUAAUUUAUGCAUU ..-----------....((((.(.(((((...))))).)))))--...(((((((..---..((((((((.--.......((((((.......))))))..))))))))....))))))) ( -30.00) >DroWil_CAF1 129524 106 - 1 AA-----------AAAUCAGAAGCAGUUGCAUCUGAUCCUACU--CUA-CUGUGCGUGUAUGUGUGUAUGGCCAUCCGAAAAUUGGUUUUAAUUCAAUUACAGUGCGUAAUUUAUGCAUU ..-----------..((((((.((....)).))))))......--...-..(((((((((((((((((.(((((.........)))))..........)))).))))))...))))))). ( -21.80) >DroYak_CAF1 121335 102 - 1 CC-----------AAAUCCGGAGCAGCUGCAGCGGAUGCCUGG--CUAAAUGCAUUC---AGUGCGCACGA--AUGCAAAAAUUGAUUUUAAUUCAAUUACAGUGCGUAAUUUAUGCAUU ((-----------(.(((((..((....))..)))))...)))--...(((((((..---..((((((((.--...)...((((((.......))))))...)))))))....))))))) ( -30.90) >DroAna_CAF1 182608 115 - 1 CCGGCAUUUUCCAGAAUCCGGAGCAGCUGCAUCUGAUCCCGGGCACUAAAUGCAUUC---UGUGCGCACGA--AUGCAAAAAUUGAUUUUAAUUCAAUUACAGUGCGUAAUUUAUGCAUU ((((.((((....)))))))).(((..(((..(((....)))(((((...(((((((---((....)).))--)))))..((((((.......))))))..)))))))).....)))... ( -32.20) >consensus AC___________AAAUCCGGAGCAGCUGCAGCGGAUCCCUGG__CUAAAUGCAUGC___UGUGCGCACGA__AUGCAAAAAUUGAUUUUAAUUCAAUUACAGUGCGUAAUUUAUGCAUU ...............(((((..((....))..)))))...........(((((((.......((((((((......)...((((((.......))))))...)))))))....))))))) (-22.32 = -22.10 + -0.22)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:50:30 2006