| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 8,179,058 – 8,179,172 |
| Length | 114 |
| Max. P | 0.786660 |

| Location | 8,179,058 – 8,179,172 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 85.79 |
| Mean single sequence MFE | -32.00 |
| Consensus MFE | -25.72 |
| Energy contribution | -25.08 |
| Covariance contribution | -0.64 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.33 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.57 |
| SVM RNA-class probability | 0.786660 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
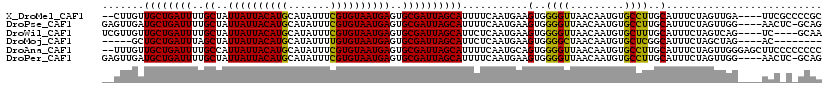
>X_DroMel_CAF1 8179058 114 + 22224390 --CUUGUUGCUGAUUUUGCUAUUAUUACAUGCAUAUUUCGUGUAAUGAGUGCGAUUAGCAUUUUCAAUGAAGUGGGGUUAACAAUGUGCCUUGCAUUUCUAGUUGA----UUCGCCCCGC --.(((.((((((((..((..((((((((((.......))))))))))..))))))))))....)))....(((((((.((((((((((...)))).....)))).----)).))))))) ( -33.20) >DroPse_CAF1 84803 115 + 1 GAGUUGAUGCUGAUUUUGCUAUUAUUACAUGCAUAUUUCGUGUAAUGAGUGCGAUUAGCAUUUUCAAUGAAGUGGGGUUAACAAUGUGCCUUGCAUUUCUAGUUGG----AACUC-GCAG (((((((((((((((..((..((((((((((.......))))))))))..)))))))))))).(((((((((((((((.........))))..))))))..)))))----)))))-.... ( -34.90) >DroWil_CAF1 72316 112 + 1 UCGUUGUUGCUGAUUUUGCUAUUAUUACAUGCAUAUUUCGUGUAAUGAGUGCGAUUAGCAUUCUCAAUGAAGUGGGGUUAACAAUGUGCUUUGCAUUUCUAGUCAG----UC----GCAA ((((((.((((((((..((..((((((((((.......))))))))))..))))))))))....)))))).(((.(..((..(((((.....)))))..))..)..----.)----)).. ( -30.50) >DroMoj_CAF1 89566 103 + 1 -----GCUGCUGAUUUAGCUAUUAUUACAUGCAUAUUUUGUGUAAUGAGUGCGAUUAGCAUUCUCAAUGAAGUGGGGCUAACAAUGUGCUCGGCAUUUCUAGCUAG----AC-------- -----........((((((((..(((((((.........)))))))((((((.....)))))).....((((((((((.........))))..)))))))))))))----).-------- ( -27.40) >DroAna_CAF1 111815 118 + 1 --UUUGUUGCUGAUUUUGCCAUUAUUACAUGCAUAUUUCGUGUAAUGAGUGCGAUUAGCAUUUUCAAUGCAGUGGGGUUAACAAUGUGCCUUGCAUUUCUAGUUGGGAGCUUCCCCCCCC --.(((.((((((((..((..((((((((((.......))))))))))..))))))))))....)))......((((.....(((((.....))))).......(((.....))))))). ( -31.10) >DroPer_CAF1 84450 115 + 1 GAGUUGAUGCUGAUUUUGCUAUUAUUACAUGCAUAUUUCGUGUAAUGAGUGCGAUUAGCAUUUUCAAUGAAGUGGGGUUAACAAUGUGCCUUGCAUUUCUAGUUGG----AACUC-GCAG (((((((((((((((..((..((((((((((.......))))))))))..)))))))))))).(((((((((((((((.........))))..))))))..)))))----)))))-.... ( -34.90) >consensus __GUUGUUGCUGAUUUUGCUAUUAUUACAUGCAUAUUUCGUGUAAUGAGUGCGAUUAGCAUUUUCAAUGAAGUGGGGUUAACAAUGUGCCUUGCAUUUCUAGUUGG____AAC_C_GCAC .......((((((((..((..((((((((((.......))))))))))..))))))))))...........(..((((.........))))..).......................... (-25.72 = -25.08 + -0.64)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:49:58 2006