
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 7,981,898 – 7,982,058 |
| Length | 160 |
| Max. P | 0.624777 |

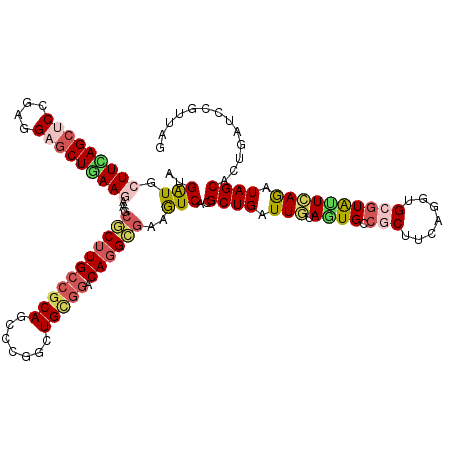
| Location | 7,981,898 – 7,982,018 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.94 |
| Mean single sequence MFE | -45.28 |
| Consensus MFE | -29.46 |
| Energy contribution | -30.30 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.30 |
| Mean z-score | -1.53 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.12 |
| SVM RNA-class probability | 0.593775 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
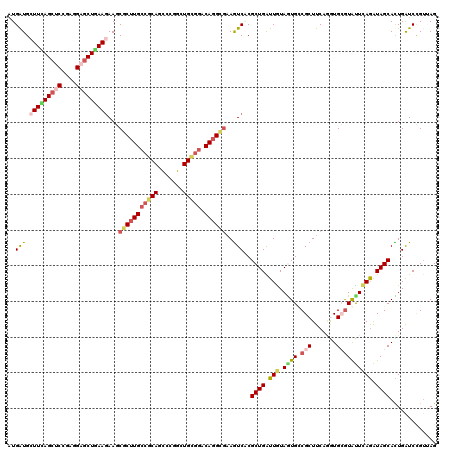
>X_DroMel_CAF1 7981898 120 - 22224390 GUGGUGCUUCAGCUCCGAGGCACUAAAGAAUCUCUUGCCGCACUCCGGCUGUGGGCAUGGGAAGUCGCGCUGAUUGUAGUGCCGCUUCAGGUGGAUGCUAAGAUAGCACUGAUCCGUUAG .(((((((((......)))))))))....((((..((((.(((.......)))))))...(((((.((((((....)))))).)))))))))((((((((...)))))....)))..... ( -41.40) >DroVir_CAF1 4882 120 - 1 AUGAUGGUUCAGCUCCGAGGAGCUGAAAAAGCGCUUGCCGCAGCCCGGUUGCGGACAGGCGAAGUCUCGCUGAUUAUAGUGUCGCUUCAAAUGCGUAUUUAGAUAGCACUGAUCCGUUAG .((((((((((((((....))))))))...((((((((((((((...))))))).)))))(((((..(((((....)))))..))))).................))......)))))). ( -49.30) >DroGri_CAF1 6287 120 - 1 AUGAUGCUUUAGCUCCGAUGAGCUGAAGAAACGCUUGCCGCAGCCCGGCUGCGUACAGGCGAAAUCACGCUGAUUAUAGUGACGCUUCAGAUGCGUAUUUAGAUAGCAUUGAUCUGACAG .((((.(((((((((....)))))))))...((((((.((((((...))))))..))))))..))))(((((....)))))((((.......))))..((((((.......))))))... ( -41.30) >DroMoj_CAF1 5681 120 - 1 AUGAUGGUUUAGCUCCGAGGAGCUAAAGAAGCGCUUGCCACAGCCCGGAUGCAGGCAGGCGAAAUCGCGCUGGUUGUAAUGGCGCUUCAAGUGCGUAUUCAGAUAGCAUUGGUCCGUUAG .((((((((((((((....))))))))((((((((..((.......)).(((((.((((((....))).))).)))))..)))))))).(((((...........)))))...)))))). ( -47.00) >DroAna_CAF1 3325 120 - 1 GUGGUGCUUCAGCUCCGAGGCGCUGAAGAACCGCUUGCCGCACUCCGGAUGCGGGCAGGCGUAGUCCCGCUGGUUGUAAUGCCGCUUCAGGUGGGUGCUCAAGUAGCAUUGGUCGGUGAG (((((.(((((((.(....).))))))).)))))(..((((((.(((..((.((((.((((......))))(((......)))))))))..)))))))((((......))))..))..). ( -47.90) >DroPer_CAF1 4321 120 - 1 AUGAUGCUUCAGCUCGGAGGAGCUGAAGAAGCGCUUGCCGCAUUCCGGCUGCGGACAGGGAAAGUCACGCUGGUUGUAGUGCCGCUUCAUGUGCGUGUUCAGGUAGCACUGAUCUGUGAG ......(((((((((....)))))))))...(((..((((.....)))).))).(((((...(((...((((.(((.((..((((.....))).)..))))).)))))))..)))))... ( -44.80) >consensus AUGAUGCUUCAGCUCCGAGGAGCUGAAGAAGCGCUUGCCGCAGCCCGGCUGCGGACAGGCGAAGUCACGCUGAUUGUAGUGCCGCUUCAGGUGCGUAUUCAGAUAGCACUGAUCCGUUAG ..(((.(((((((((....)))))))))...(((((((((((.......))))).))))))..)))..((((.(((.((((.(((.......)))))))))).))))............. (-29.46 = -30.30 + 0.84)

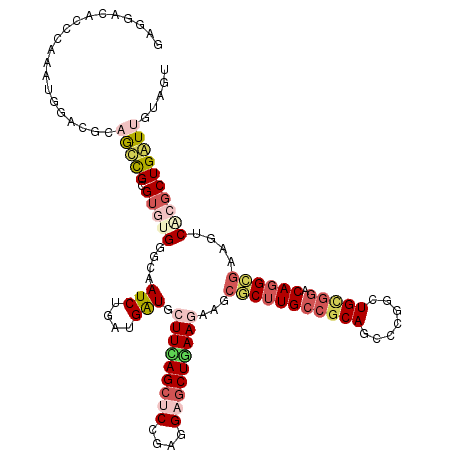
| Location | 7,981,938 – 7,982,058 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.72 |
| Mean single sequence MFE | -49.53 |
| Consensus MFE | -25.88 |
| Energy contribution | -27.47 |
| Covariance contribution | 1.59 |
| Combinations/Pair | 1.27 |
| Mean z-score | -1.85 |
| Structure conservation index | 0.52 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.624777 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
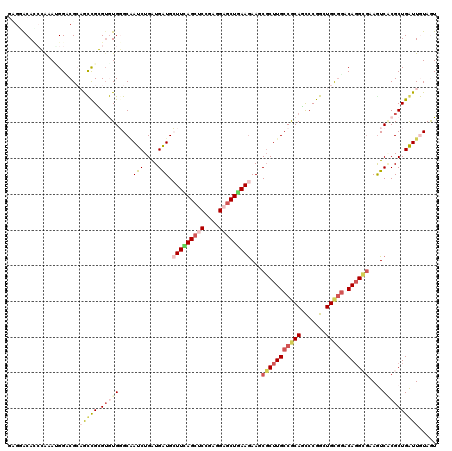
>X_DroMel_CAF1 7981938 120 - 22224390 GUGGACAGGCAAAUGGUCGCUGCUGGGUGUGGGCAAUCUGGUGGUGCUUCAGCUCCGAGGCACUAAAGAAUCUCUUGCCGCACUCCGGCUGUGGGCAUGGGAAGUCGCGCUGAUUGUAGU ((((((...(..(((.((((.(((((((((((.((((((..(((((((((......))))))))).))).....))))))))).))))).)))).)))..)..))).))).......... ( -50.60) >DroVir_CAF1 4922 120 - 1 GCGCACAGCGAAAGGGACGCAAUCGCGUAUGCGCUAUCUGAUGAUGGUUCAGCUCCGAGGAGCUGAAAAAGCGCUUGCCGCAGCCCGGUUGCGGACAGGCGAAGUCUCGCUGAUUAUAGU .....((((((.....((((....))))..((((((((....))))))(((((((....)))))))....))((((((((((((...))))))).)))))......))))))........ ( -52.40) >DroGri_CAF1 6327 120 - 1 AUGGACACCGAAAUGGACGGAGUCGCGUAUGGGCAAUCUGAUGAUGCUUUAGCUCCGAUGAGCUGAAGAAACGCUUGCCGCAGCCCGGCUGCGUACAGGCGAAAUCACGCUGAUUAUAGU ...(((.(((.......))).)))((((((.((....)).))(((.(((((((((....)))))))))...((((((.((((((...))))))..))))))..))))))).......... ( -41.40) >DroMoj_CAF1 5721 120 - 1 GCGAGCAUCGAAAUGGACGCAGCCGCGUGUGCGCAAUCUGAUGAUGGUUUAGCUCCGAGGAGCUAAAGAAGCGCUUGCCACAGCCCGGAUGCAGGCAGGCGAAAUCGCGCUGGUUGUAAU .((.....))........(((((((((((.((((.(((....)))..((((((((....))))))))...))))(((((...(((........))).)))))...))))).))))))... ( -44.30) >DroAna_CAF1 3365 120 - 1 CAGGACACCCGAAGGGCCGCAGCUGGGAGUGGGCGAUCUGGUGGUGCUUCAGCUCCGAGGCGCUGAAGAACCGCUUGCCGCACUCCGGAUGCGGGCAGGCGUAGUCCCGCUGGUUGUAAU ..((((((((....(.(((((..(.((((((((((....((((((.(((((((.(....).))))))).)))))))))).)))))).).))))).).)).)).))))............. ( -59.10) >DroPer_CAF1 4361 120 - 1 GAGUGCACGCGUACGGCCGAUACUGGGUGUGGGCGAUCUGAUGAUGCUUCAGCUCGGAGGAGCUGAAGAAGCGCUUGCCGCAUUCCGGCUGCGGACAGGGAAAGUCACGCUGGUUGUAGU ((((((.(((.((((.(((....))).)))).)))(((....))).(((((((((....)))))))))..))))))((..((..(((((....(((.......)))..))))).))..)) ( -49.40) >consensus GAGGACACCCAAAUGGACGCAGCCGCGUGUGGGCAAUCUGAUGAUGCUUCAGCUCCGAGGAGCUGAAGAAGCGCUUGCCGCAGCCCGGCUGCGGACAGGCGAAGUCACGCUGAUUGUAGU ....................(((((.(((((....(((....))).(((((((((....)))))))))...(((((((((((.......))))).))))))....))))))))))..... (-25.88 = -27.47 + 1.59)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:48:37 2006