
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 7,895,966 – 7,896,081 |
| Length | 115 |
| Max. P | 0.989491 |

| Location | 7,895,966 – 7,896,081 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 85.60 |
| Mean single sequence MFE | -27.28 |
| Consensus MFE | -16.23 |
| Energy contribution | -16.39 |
| Covariance contribution | 0.16 |
| Combinations/Pair | 1.04 |
| Mean z-score | -3.44 |
| Structure conservation index | 0.59 |
| SVM decision value | 1.18 |
| SVM RNA-class probability | 0.926749 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
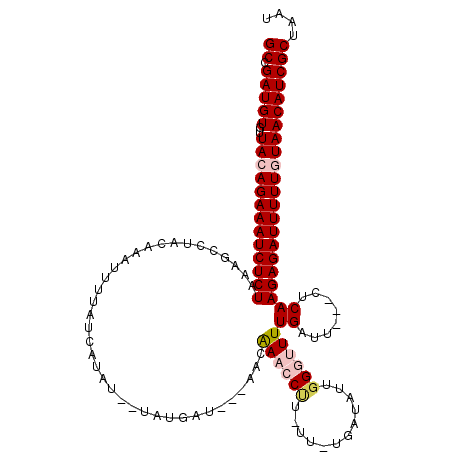
>X_DroMel_CAF1 7895966 115 + 22224390 AUUAGCGAUGUUACAAAAAUCUCUUGAG---AAUCAAAACCCAAUAUCA-AA-AAGGUUUUUUGCGAUCAUACCAUAUGAUAAAAUUUGUAGGCUUUAGAGAUUUCUGUAACACAUCGGC .....((((((((((.((((((((.(((---....((((((........-..-..))))))(((((((((((...)))))......)))))).))).)))))))).))))..)))))).. ( -27.10) >DroSec_CAF1 6661 111 + 1 AUUAGCGAUGUUACAAAAAUCUCUUGAG---UAACAAAACCCAAUAUCA-AA-AAGGUUCGUU---AUCAUA-CAUAUGAUAAAAUUUGUAGGCUUUAGAGAUUUCUGUAACACAUCGGC .....((((((((((.((((((((.(((---(.((((((((........-..-..)))...((---(((((.-...)))))))..)))))..)))).)))))))).))))..)))))).. ( -30.90) >DroSim_CAF1 6564 112 + 1 AUUAGCGAUGUUACAAAAAUCUCUUGAG---UAACAAAACCCAAUAUCAGAA-AAGGUUCGUU---AUCAUA-AAUAUGAUAAAAUUUGUAGGCUUUAGAGAUUUCUGUAACACAUCGGC .....((((((((((.((((((((.(((---(.(((((...........(((-....))).((---(((((.-...)))))))..)))))..)))).)))))))).))))..)))))).. ( -31.10) >DroEre_CAF1 6383 105 + 1 AUUAGCGAUGUUAUAAAAAUCUCUUGAG---AAUCAAAACCCAAUAUCA-AAAAUGUGUUGUU---AUCACA---UAA---GGAAUUUGUAA--UUUAGAGAUUUCUGUAACACAUCGGC .....((((((((((.((((((((.((.---...((((..((.......-...(((((.....---..))))---)..---))..))))...--)).)))))))).))))..)))))).. ( -21.90) >DroYak_CAF1 6564 107 + 1 AUUAGCGAUGUUAUAAAAAUCUCUUGAGUAUAAUCAAAAGCCAAUAUCA-AA-AGGUAUUGUU---AUCACA---UGU---GGAAUUUGUAA--UUUAGAGAUUUCUGUAACACAUCGGC .....((((((((((.((((((((....(((((........(((((((.-..-.)))))))..---.(((..---..)---))...))))).--...)))))))).))))..)))))).. ( -25.40) >consensus AUUAGCGAUGUUACAAAAAUCUCUUGAG___AAUCAAAACCCAAUAUCA_AA_AAGGUUUGUU___AUCAUA__AUAUGAUAAAAUUUGUAGGCUUUAGAGAUUUCUGUAACACAUCGGC .....((((((((((.((((((((.(((......((((...............................................))))....))).)))))))).))))..)))))).. (-16.23 = -16.39 + 0.16)


| Location | 7,895,966 – 7,896,081 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 85.60 |
| Mean single sequence MFE | -27.90 |
| Consensus MFE | -19.50 |
| Energy contribution | -20.54 |
| Covariance contribution | 1.04 |
| Combinations/Pair | 1.07 |
| Mean z-score | -3.44 |
| Structure conservation index | 0.70 |
| SVM decision value | 2.17 |
| SVM RNA-class probability | 0.989491 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7895966 115 - 22224390 GCCGAUGUGUUACAGAAAUCUCUAAAGCCUACAAAUUUUAUCAUAUGGUAUGAUCGCAAAAAACCUU-UU-UGAUAUUGGGUUUUGAUU---CUCAAGAGAUUUUUGUAACAUCGCUAAU ((.(((((..(((((((((((((((((((((.......(((((...(((.............)))..-..-))))).))))))))(...---..).)))))))))))))))))))).... ( -30.52) >DroSec_CAF1 6661 111 - 1 GCCGAUGUGUUACAGAAAUCUCUAAAGCCUACAAAUUUUAUCAUAUG-UAUGAU---AACGAACCUU-UU-UGAUAUUGGGUUUUGUUA---CUCAAGAGAUUUUUGUAACAUCGCUAAU ((.(((((..(((((((((((((..((...(((....(((((((...-.)))))---)).((((((.-.(-....)..)))))))))..---))..)))))))))))))))))))).... ( -32.40) >DroSim_CAF1 6564 112 - 1 GCCGAUGUGUUACAGAAAUCUCUAAAGCCUACAAAUUUUAUCAUAUU-UAUGAU---AACGAACCUU-UUCUGAUAUUGGGUUUUGUUA---CUCAAGAGAUUUUUGUAACAUCGCUAAU ((.(((((..(((((((((((((..((...(((....(((((((...-.)))))---)).((((((.-..........)))))))))..---))..)))))))))))))))))))).... ( -32.20) >DroEre_CAF1 6383 105 - 1 GCCGAUGUGUUACAGAAAUCUCUAAA--UUACAAAUUCC---UUA---UGUGAU---AACAACACAUUUU-UGAUAUUGGGUUUUGAUU---CUCAAGAGAUUUUUAUAACAUCGCUAAU ((.(((((.....((((((((((...--...((((....---..(---((((..---.....))))).))-))....((((........---))))))))))))))...))))))).... ( -22.70) >DroYak_CAF1 6564 107 - 1 GCCGAUGUGUUACAGAAAUCUCUAAA--UUACAAAUUCC---ACA---UGUGAU---AACAAUACCU-UU-UGAUAUUGGCUUUUGAUUAUACUCAAGAGAUUUUUAUAACAUCGCUAAU ((.(((((.....((((((((((..(--(((((......---...---))))))---..(((((.(.-..-.).))))).................))))))))))...))))))).... ( -21.70) >consensus GCCGAUGUGUUACAGAAAUCUCUAAAGCCUACAAAUUUUAUCAUAU__UAUGAU___AACAAACCUU_UU_UGAUAUUGGGUUUUGAUU___CUCAAGAGAUUUUUGUAACAUCGCUAAU ((.(((((..(((((((((((((.....................................((((((............))))))((........)))))))))))))))))))))).... (-19.50 = -20.54 + 1.04)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:47:31 2006