
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 7,830,066 – 7,830,226 |
| Length | 160 |
| Max. P | 0.842929 |

| Location | 7,830,066 – 7,830,186 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.33 |
| Mean single sequence MFE | -52.97 |
| Consensus MFE | -37.71 |
| Energy contribution | -36.93 |
| Covariance contribution | -0.77 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.22 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.598050 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7830066 120 + 22224390 UGGUGUCUGUCGGCGGGCGGUGGCAUCCAGGCCACCGGCAAUCGUCGCUGUGCCGACGCUCUCUUCGAGCUGAUCUGGCACGGCGAUGGCUCGCUCUCGUUCCGGGCUAACAACGGCAAG .......(((((((((((((((((......))))))......(((((((((((((((((((.....)))).).)).)))))))))))))))))).((((...)))).......))))).. ( -58.90) >DroVir_CAF1 3698 120 + 1 UGGUGCCUAUCUGCCGGCGGCGGCAUUCAGGCCACCGGCAAUCGUCGCUGUGCCGAUGCACUCUUCGAACUGAUCUGGCAUGGUGAUGGUUCGCUCUCGUUUCGCGCCAAUAAUGGCAAA .(((((((...(((((....)))))...))).))))(((((((((((((((((((((.((..........))))).))))))))))))))).)))..........((((....))))... ( -48.50) >DroGri_CAF1 3643 120 + 1 UGGUGCCUGUCAGCGGGCGGCGGCAUUCAGGCCACCGGCAAUCGUCGCUGUGCCGAUGCUUUAUUCGAGCUAAUCUGGCAUGGCGACGGCUCCUUGUCGUUCCGGGCCAACAAUGGCAAA .(.((((((....)))))).)(((......))).((((.(((.((((((((((((((((((.....))))..))).)))))))))))(((.....))))))))))((((....))))... ( -54.70) >DroWil_CAF1 19046 120 + 1 UGGUGUCUAUCGUCCGGCGGUGGCAUUCAGGCCACUGGCAAUAGACGCUGUGCCGAUGCUCUAUUCGAAUUAAUCUGGCACGGUGAUGGUUCCCUCUCAUUCCGUGCCAAUAAUGGUAAA .(((((((((.(((....((((((......))))))))).))))))))).((((((........)).........((((((((.(((((.......))))))))))))).....)))).. ( -45.00) >DroMoj_CAF1 3903 120 + 1 UGGUGCCUGUCGGCUGGCGGCGGCAUCCAGGCCACUGGCAAUCGUCGCUGUGCCGAUGCCCUCUUCGAGCUCAUCUGGCAUGGCGAUGGCUCGCUUUCUUUCCGCGCCAACAAUGGCAAG (((((((.((((.....)))))))).))).((((.((((..(((((((((((((((((..(((...)))..)))).)))))))))))))...((.........))))))....))))... ( -52.90) >DroAna_CAF1 2929 120 + 1 UGGUGCCUGUCGGCCGGCGGCGGUAUCCAGGCCACUGGGAAUCGGCGUUGUGCCGAUGCCCUUUUCGAGCUUAUCUGGCACGGGGACGGGUCCCUAUCCUUCCGUGCCAACAACGGCAAA .((.((..(((((((((((.((((.(((((....))))).)))).))))).)))))))))).......(((....(((((((((((..(....)..)))..)))))))).....)))... ( -57.80) >consensus UGGUGCCUGUCGGCCGGCGGCGGCAUCCAGGCCACCGGCAAUCGUCGCUGUGCCGAUGCUCUCUUCGAGCUAAUCUGGCACGGCGAUGGCUCCCUCUCGUUCCGCGCCAACAAUGGCAAA ...(((((((.(((((((((.(((......))).)))....(((((((((((((((........))((.....)).)))))))))))))............))).))).)))..)))).. (-37.71 = -36.93 + -0.77)


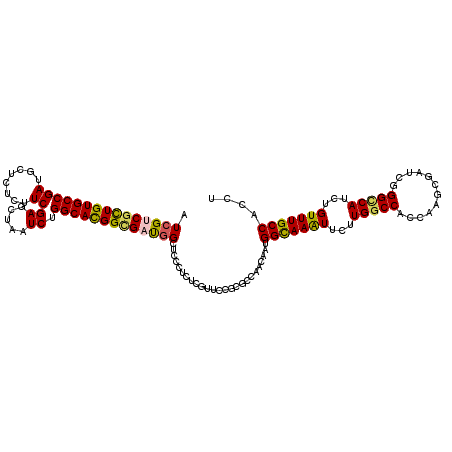
| Location | 7,830,106 – 7,830,226 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.39 |
| Mean single sequence MFE | -44.20 |
| Consensus MFE | -34.67 |
| Energy contribution | -34.20 |
| Covariance contribution | -0.47 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.57 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.76 |
| SVM RNA-class probability | 0.842929 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7830106 120 + 22224390 AUCGUCGCUGUGCCGACGCUCUCUUCGAGCUGAUCUGGCACGGCGAUGGCUCGCUCUCGUUCCGGGCUAACAACGGCAAGUUCUUGGCCACCAAGCGCUCUGGUCAUCUGUUUGCCACCU ..(((((((((((((((((((.....)))).).)).))))))))))))(((((.........))))).......((((((....((((((..........))))))....)))))).... ( -48.00) >DroVir_CAF1 3738 120 + 1 AUCGUCGCUGUGCCGAUGCACUCUUCGAACUGAUCUGGCAUGGUGAUGGUUCGCUCUCGUUUCGCGCCAAUAAUGGCAAAUUUUUGGCCACCAAGCGUUCCGGUCAUUUGUUUGCCACCU (((((((((((((((((.((..........))))).)))))))))))))).(((.........))).......((((((((...(((((............)))))...))))))))... ( -38.00) >DroGri_CAF1 3683 120 + 1 AUCGUCGCUGUGCCGAUGCUUUAUUCGAGCUAAUCUGGCAUGGCGACGGCUCCUUGUCGUUCCGGGCCAACAAUGGCAAAUUCUUGGCCACCAAGCGUUCGGGCCAUUUGUUUGCCACCU ...((((((((((((((((((.....))))..))).)))))))))))(((((...........))))).....((((((((...(((((............)))))...))))))))... ( -50.30) >DroWil_CAF1 19086 120 + 1 AUAGACGCUGUGCCGAUGCUCUAUUCGAAUUAAUCUGGCACGGUGAUGGUUCCCUCUCAUUCCGUGCCAAUAAUGGUAAAUUUUUGGCCACCAAAAGAUCCGGCCAUCUGUUUGCCACCU (((((.((.........)))))))...........((((((((.(((((.......)))))))))))))....((((((((...(((((............)))))...))))))))... ( -38.40) >DroMoj_CAF1 3943 120 + 1 AUCGUCGCUGUGCCGAUGCCCUCUUCGAGCUCAUCUGGCAUGGCGAUGGCUCGCUUUCUUUCCGCGCCAACAAUGGCAAGUUCCUGGCCACCAAGCGAUCGGGCCACUUGUUUGCCACUU ...(((((((((((((((..(((...)))..)))).)))))))))))(((.((.........)).))).....(((((((....(((((............)))))....)))))))... ( -45.00) >DroAna_CAF1 2969 120 + 1 AUCGGCGUUGUGCCGAUGCCCUUUUCGAGCUUAUCUGGCACGGGGACGGGUCCCUAUCCUUCCGUGCCAACAACGGCAAAUUCUUGGCCACCAAGCGCUCGGGACAUCUGUUUGCCACCU ...((((....((.(((((((.....((((.....(((((((((((..(....)..)))..)))))))).....(((.........))).......))))))).)))).)).)))).... ( -45.50) >consensus AUCGUCGCUGUGCCGAUGCUCUCUUCGAGCUAAUCUGGCACGGCGAUGGCUCCCUCUCGUUCCGCGCCAACAAUGGCAAAUUCUUGGCCACCAAGCGAUCGGGCCAUCUGUUUGCCACCU .(((((((((((((((........))((.....)).))))))))))))).........................(((((((...(((((............)))))...))))))).... (-34.67 = -34.20 + -0.47)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:46:43 2006