
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 7,767,036 – 7,767,203 |
| Length | 167 |
| Max. P | 0.999912 |

| Location | 7,767,036 – 7,767,153 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 89.75 |
| Mean single sequence MFE | -39.13 |
| Consensus MFE | -33.32 |
| Energy contribution | -33.00 |
| Covariance contribution | -0.32 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.29 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.01 |
| SVM RNA-class probability | 0.539769 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7767036 117 + 22224390 --GCUAUUGAUGUUGUGGGCCUUCUUAUCGCCUCCACGGCGGAUCUAUGUAUACCAAUCGGAUCGGAAGGCUGCACC-UAUGUAUACACAUGCAGUCGCACUUCCGAUCCGGGCAACUGG --.........(((((((.........(((((.....)))))...........))..((((((((((((((((((..-..(((....)))))))))....)))))))))))))))))... ( -39.75) >DroSec_CAF1 1134 113 + 1 --GCUAUUGAUGUUGUGGGCUUUCUUAUCGCCUCCCCGGCGGAUCUAUGUACACCAAUCGGAUCGGAUGGCUGCA-----UGUAUACACAUGCAGUCGCACUUCCGAUCCGGGCAACUGG --(((...(((...(((..(.......(((((.....)))))......)..)))..)))((((((((((((((((-----(((....)))))))))))....)))))))).)))...... ( -45.72) >DroSim_CAF1 1134 117 + 1 --GCUAUUGAUGUUGUGGGCUUUCUUAUCGCCUCCCCGGCGGAUCUAUGUACACCAAUCGGAUCGGAUGGCUGCACC-UAUGUAUACACAUGUAGUCGCACUUCCGAUCCGGGCAACUGG --(((...(((...(((..(.......(((((.....)))))......)..)))..)))((((((((.((.(((..(-(((((......))))))..))))))))))))).)))...... ( -38.22) >DroEre_CAF1 1171 113 + 1 --GCUAUUGAUGUUGUGGGCUUUCUUAUCGCCUCCCCGGCGGAUCUAUGUAUACCUAUUGGAUCGGGAGGCUGCAGC-----UAUACACAUACAGUCGCACUUCCGAUCUGGGCAACUGG --(((......((((..(.((..(....((((.....))))((((((...........)))))))..)).)..))))-----..........((((((......))).))))))...... ( -34.60) >DroYak_CAF1 1142 116 + 1 CAGCUAUUGAUGUUGUGGGCUUUCUUAUCGCCUCCCCGGCGGAUCUA----UAGCUAUCGGAUCGGGAGGCUGCACCCUACAUAUACACAUGCAGUCGCACUUCGGAUCCGGGCAACUGG ...........(((((.((((......(((((.....))))).....----.)))).(((((((.((((((((((...............))))))....)))).))))))))))))... ( -37.36) >consensus __GCUAUUGAUGUUGUGGGCUUUCUUAUCGCCUCCCCGGCGGAUCUAUGUAUACCAAUCGGAUCGGAAGGCUGCACC_UAUGUAUACACAUGCAGUCGCACUUCCGAUCCGGGCAACUGG ...........(((((...........(((((.....)))))...............((((((((((.(((((((...............))))))).....)))))))))))))))... (-33.32 = -33.00 + -0.32)


| Location | 7,767,074 – 7,767,183 |
|---|---|
| Length | 109 |
| Sequences | 5 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 90.38 |
| Mean single sequence MFE | -36.23 |
| Consensus MFE | -31.92 |
| Energy contribution | -31.60 |
| Covariance contribution | -0.32 |
| Combinations/Pair | 1.13 |
| Mean z-score | -1.39 |
| Structure conservation index | 0.88 |
| SVM decision value | 0.52 |
| SVM RNA-class probability | 0.768104 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7767074 109 + 22224390 GGAUCUAUGUAUACCAAUCGGAUCGGAAGGCUGCACC-UAUGUAUACACAUGCAGUCGCACUUCCGAUCCGGGCAACUGGCGGUCGGAAUGUGUGGGCGUUUCAAUAAAG .((((............((((((((((((((((((..-..(((....)))))))))....))))))))))))((.....))))))(((((((....)))))))....... ( -37.80) >DroSec_CAF1 1172 105 + 1 GGAUCUAUGUACACCAAUCGGAUCGGAUGGCUGCA-----UGUAUACACAUGCAGUCGCACUUCCGAUCCGGGCAACUGGCGGUCGGAAUGUGUGGGCGUUUCAAUAAAG .((((............((((((((((((((((((-----(((....)))))))))))....))))))))))((.....))))))(((((((....)))))))....... ( -42.90) >DroSim_CAF1 1172 109 + 1 GGAUCUAUGUACACCAAUCGGAUCGGAUGGCUGCACC-UAUGUAUACACAUGUAGUCGCACUUCCGAUCCGGGCAACUGGCGGUCGGAAUGUGUGGGCGUUUCAAUAAAG .((((............((((((((((.((.(((..(-(((((......))))))..)))))))))))))))((.....))))))(((((((....)))))))....... ( -35.40) >DroEre_CAF1 1209 105 + 1 GGAUCUAUGUAUACCUAUUGGAUCGGGAGGCUGCAGC-----UAUACACAUACAGUCGCACUUCCGAUCUGGGCAACUGGCGGUCGGAAUGUGUGGGCGUUUCAAUAAAG .((((((...........))))))(..(.(((...((-----(.((....)).)))(((((((((((((..(....)..)..))))))).)))))))).)..)....... ( -32.70) >DroYak_CAF1 1182 106 + 1 GGAUCUA----UAGCUAUCGGAUCGGGAGGCUGCACCCUACAUAUACACAUGCAGUCGCACUUCGGAUCCGGGCAACUGGCGGUCGGAAUGUGUGGGCGUUUCAAUAAAG .((((..----(((...(((((((.((((((((((...............))))))....)))).)))))))....)))..))))(((((((....)))))))....... ( -32.36) >consensus GGAUCUAUGUAUACCAAUCGGAUCGGAAGGCUGCACC_UAUGUAUACACAUGCAGUCGCACUUCCGAUCCGGGCAACUGGCGGUCGGAAUGUGUGGGCGUUUCAAUAAAG .((((............((((((((((.(((((((...............))))))).....))))))))))((.....))))))(((((((....)))))))....... (-31.92 = -31.60 + -0.32)


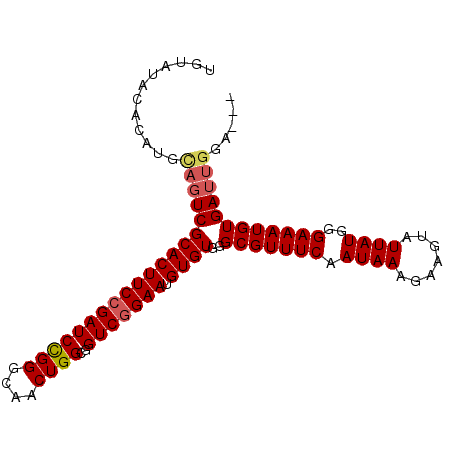
| Location | 7,767,113 – 7,767,203 |
|---|---|
| Length | 90 |
| Sequences | 5 |
| Columns | 98 |
| Reading direction | forward |
| Mean pairwise identity | 93.00 |
| Mean single sequence MFE | -31.18 |
| Consensus MFE | -29.08 |
| Energy contribution | -29.60 |
| Covariance contribution | 0.52 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.57 |
| Structure conservation index | 0.93 |
| SVM decision value | 4.51 |
| SVM RNA-class probability | 0.999912 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7767113 90 + 22224390 UGUAUACACAUGCAGUCGCACUUCCGAUCCGGGCAACUGGCGGUCGGAAUGUGUGGGCGUUUCAAUAAAGAAGUAUUAUGGGAAAUGUGA-------- (((((....))))).(((((((((((((((((....))))..))))))).))))))(((((((.((((.......))))..)))))))..-------- ( -32.40) >DroSec_CAF1 1207 98 + 1 UGUAUACACAUGCAGUCGCACUUCCGAUCCGGGCAACUGGCGGUCGGAAUGUGUGGGCGUUUCAAUAAAGAAGUAUUAUGGGAAAUGUGAUUGGAACA ..........(.((((((((((((((((((((....))))..))))))).))))..(((((((.((((.......))))..)))))))))))).)... ( -33.00) >DroSim_CAF1 1211 98 + 1 UGUAUACACAUGUAGUCGCACUUCCGAUCCGGGCAACUGGCGGUCGGAAUGUGUGGGCGUUUCAAUAAAGAAGUAUUAUGGGAAAUGUGAUUGGAACA ..........(.((((((((((((((((((((....))))..))))))).))))..(((((((.((((.......))))..)))))))))))).)... ( -31.30) >DroEre_CAF1 1246 93 + 1 --UAUACACAUACAGUCGCACUUCCGAUCUGGGCAACUGGCGGUCGGAAUGUGUGGGCGUUUCAAUAAAGAAGUAUUAUGGGAAAUGUGAUUGGA--- --..........(((((((((((((((((..(....)..)..))))))).))))..(((((((.((((.......))))..))))))))))))..--- ( -30.60) >DroYak_CAF1 1218 95 + 1 CAUAUACACAUGCAGUCGCACUUCGGAUCCGGGCAACUGGCGGUCGGAAUGUGUGGGCGUUUCAAUAAAGAAGUAUUAUGGGAAAUGUGAUUGGA--- ............((((((((((((.(((((((....))))..))).))).))))..(((((((.((((.......))))..))))))))))))..--- ( -28.60) >consensus UGUAUACACAUGCAGUCGCACUUCCGAUCCGGGCAACUGGCGGUCGGAAUGUGUGGGCGUUUCAAUAAAGAAGUAUUAUGGGAAAUGUGAUUGGA___ ............((((((((((((((((((((....))))..))))))).))))..(((((((.((((.......))))..))))))))))))..... (-29.08 = -29.60 + 0.52)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:46:09 2006