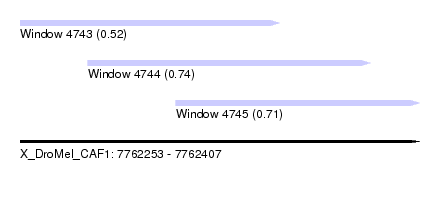
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 7,762,253 – 7,762,407 |
| Length | 154 |
| Max. P | 0.736310 |

| Location | 7,762,253 – 7,762,353 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | forward |
| Mean pairwise identity | 83.10 |
| Mean single sequence MFE | -41.43 |
| Consensus MFE | -30.11 |
| Energy contribution | -29.67 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.73 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.524889 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
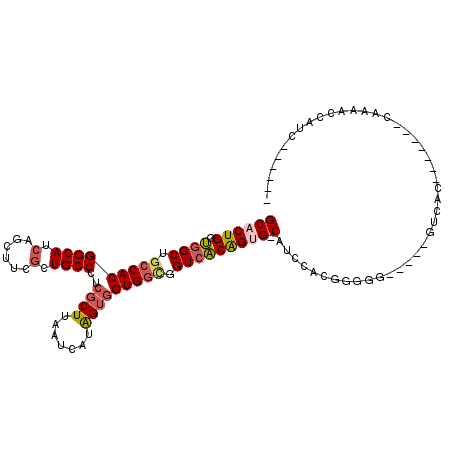
>X_DroMel_CAF1 7762253 100 + 22224390 --UCCAAUGGAACCAAGUUAAAUACGGCUACUGCUGGAGCGGCAGCUGCAUCCGCCGCUGCGGCACUGGUGGCUGCCACGGCAUCAGCUUCGCUGCCCUCGC --(((...))).............((((....))))(((.((((((.((....((((..(((((.(....))))))..))))....))...))))))))).. ( -38.60) >DroPse_CAF1 11471 99 + 1 CCACCAAUGGCACCAAGUUGAACACAGCUACCGCGGGGGCUGCAGCUGCCUCGGCUGCAGCAGCUCUGGCAGCCGCCACGGCAGCCGCCACCCUGCCC---C (((....))).....(((((....)))))...((((((((((((((((...)))))))))).....((((.((.((....)).)).))))))))))..---. ( -45.70) >DroSim_CAF1 10891 100 + 1 --UCCAAUGGAACCAAGUUAAAUACGGCCACUGCUGGAGCGGCAGCUGCGUCUGCCGCUGCGGCACUGGUGGCUGCCACGGCAUCAGCUUCGCUGCCCUCGC --.....((((((...))).....((((((((((((.((((((((......)))))))).))))...))))))))))).((((.(......).))))..... ( -40.90) >DroEre_CAF1 11703 100 + 1 --ACCAAUGGAACCAAGUUAAAUACGGCCACUGCUGGAGCGGCAGCUGCAUCCGCUGCAGCGGCACUGGUGGCGGCCACGGCAUCAGCUUCGCUGCCCUCGC --......................((((....))))(((.((((((.....((((....))))..((((((.((....)).))))))....))))))))).. ( -37.90) >DroYak_CAF1 11704 100 + 1 --GCCAAUGGAACCAAGUUGAAUACGGCCACUGCUGGAGCGGCGGCUGCAUCCGCUGCAGCGGCACUGGUGGCAGCCACGGCAUCAGCAUCGCUGCCCUCGC --......((.....(((.((...(((((.((((....)))).)))))..)).)))(((((((..((((((.(......).))))))..))))))))).... ( -39.80) >DroPer_CAF1 11540 99 + 1 CCACCAAUGGCACCAAGUUGAACACAGCUACCGCGGGGGCUGCAGCUGCCUCGGCUGCAGCAGCUCUGGCAGCCGCCACGGCAGCCGCCACCCUGCCC---C (((....))).....(((((....)))))...((((((((((((((((...)))))))))).....((((.((.((....)).)).))))))))))..---. ( -45.70) >consensus __ACCAAUGGAACCAAGUUAAAUACGGCCACUGCUGGAGCGGCAGCUGCAUCCGCUGCAGCGGCACUGGUGGCCGCCACGGCAUCAGCUUCGCUGCCCUCGC .......(((......((.....)).(((((((.((..((((((((.......))))))))..)).)))))))..))).((((..........))))..... (-30.11 = -29.67 + -0.44)

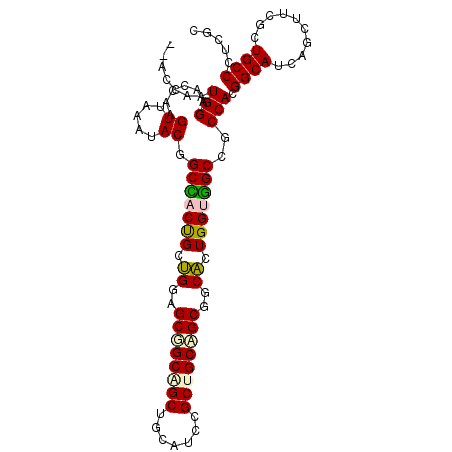
| Location | 7,762,279 – 7,762,388 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 80.93 |
| Mean single sequence MFE | -53.27 |
| Consensus MFE | -33.32 |
| Energy contribution | -32.55 |
| Covariance contribution | -0.77 |
| Combinations/Pair | 1.31 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.44 |
| SVM RNA-class probability | 0.736310 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7762279 109 + 22224390 UACUGCUGGAGCGGCAGCUGCAUCCGCCGCUGCGGCACUGGUGGCUGCCACGGCAUCAGCUUCGCUGCCCUCGCUUAAUCAUAGUGGUGGGGGUCACAGUG-CAUCCACG ......((((...(((((.((....)).))))).((((((.(((((.((((((((.(......).))))..((((.......)))))))).))))))))))-).)))).. ( -48.60) >DroPse_CAF1 11499 107 + 1 UACCGCGGGGGCUGCAGCUGCCUCGGCUGCAGCAGCUCUGGCAGCCGCCACGGCAGCCGCCACCCUGCCC---CUUAACCAUGGCAGUGCCAGUGGCAGUGGCAGUGGCG ....((.((((((((.((.((....)).)).)))))))).))...((((((.((.((.(((((((((((.---.........))))).)...))))).)).)).)))))) ( -55.90) >DroSec_CAF1 11382 109 + 1 CACUGCUGGAGCGGCAGCUGCGUCCGCCGCUGCGGCACUGGUGGCUGCCACGGCAUCAGCUUCGCUGCCCUCGCUUAAUCAUAGUGGUGGCGGUCACAGUG-CAUCCACG ......((((...(((((.((....)).))))).((((((.((((((((((((((.(......).))))..((((.......)))))))))))))))))))-).)))).. ( -52.90) >DroSim_CAF1 10917 109 + 1 CACUGCUGGAGCGGCAGCUGCGUCUGCCGCUGCGGCACUGGUGGCUGCCACGGCAUCAGCUUCGCUGCCCUCGCUUAAUCAUAGUGGUGGCGGUCACAGUG-CAUCCGCG ....((((.((((((((......)))))))).))((((((.((((((((((((((.(......).))))..((((.......)))))))))))))))))))-)....)). ( -55.60) >DroEre_CAF1 11729 109 + 1 CACUGCUGGAGCGGCAGCUGCAUCCGCUGCAGCGGCACUGGUGGCGGCCACGGCAUCAGCUUCGCUGCCCUCGCUUAAUCAUAGUGGUGGUGGUCACAGUG-CGUCCACG ...(((((.(((((.(......)))))).)))))((((((.((((.(((((((((.(......).))))..((((.......))))))))).)))))))))-)....... ( -49.40) >DroPer_CAF1 11568 107 + 1 UACCGCGGGGGCUGCAGCUGCCUCGGCUGCAGCAGCUCUGGCAGCCGCCACGGCAGCCGCCACCCUGCCC---CUGAACCAUGGCAGUGCCAGUGGCAGUGGCAGUGGCG ..((((((((((((((((((...)))))))))).....((((.((.((....)).)).))))))))(((.---(((..(((((((...)))).)))))).))).)))).. ( -57.20) >consensus CACUGCUGGAGCGGCAGCUGCAUCCGCCGCAGCGGCACUGGUGGCUGCCACGGCAUCAGCUUCGCUGCCCUCGCUUAAUCAUAGUGGUGGCGGUCACAGUG_CAUCCACG (((.((((..(((((.(((((....((((...))))....))))).)))...))..))))...((((((.(((((.......))))).))))).)...)))......... (-33.32 = -32.55 + -0.77)



| Location | 7,762,313 – 7,762,407 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 80.24 |
| Mean single sequence MFE | -40.17 |
| Consensus MFE | -23.58 |
| Energy contribution | -24.25 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.707694 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7762313 94 + 22224390 GCACUGGUGGCUGCCACGGCAUCAGCUUCGCUGCCCUCGCUUAAUCAUAGUGGUGGGGGUCACAGUG-CAUCCACGGGGG-----GUCAC-------CAAAACCAUC------ ((((((.(((((.((((((((.(......).))))..((((.......)))))))).))))))))))-)......((.((-----....)-------)....))...------ ( -37.40) >DroPse_CAF1 11533 109 + 1 GCUCUGGCAGCCGCCACGGCAGCCGCCACCCUGCCC---CUUAACCAUGGCAGUGCCAGUGGCAGUGGCAGUGGCGGCGGACUG-ACCACCGCCUCUCAAAACCAUCACCAGC ((..((((((((((....)).((((((((..((((.---((...(((((((...)))).))).)).)))))))))))))).)))-.)))..)).................... ( -46.10) >DroSec_CAF1 11416 94 + 1 GCACUGGUGGCUGCCACGGCAUCAGCUUCGCUGCCCUCGCUUAAUCAUAGUGGUGGCGGUCACAGUG-CAUCCACGGGUG-----GUCAC-------CAAAACCAUC------ ((((((.((((((((((((((.(......).))))..((((.......)))))))))))))))))))-).......((((-----((...-------....))))))------ ( -41.70) >DroSim_CAF1 10951 94 + 1 GCACUGGUGGCUGCCACGGCAUCAGCUUCGCUGCCCUCGCUUAAUCAUAGUGGUGGCGGUCACAGUG-CAUCCGCGGGGG-----GUCAC-------CAAAACCAUC------ ((((((.((((((((((((((.(......).))))..((((.......)))))))))))))))))))-)......((.((-----....)-------)....))...------ ( -41.20) >DroEre_CAF1 11763 97 + 1 GCACUGGUGGCGGCCACGGCAUCAGCUUCGCUGCCCUCGCUUAAUCAUAGUGGUGGUGGUCACAGUG-CGUCCACGGGAG--UGCAUCAC-------CAGAACCAUC------ ((((((.((((.(((((((((.(......).))))..((((.......))))))))).)))))))))-)...(((....)--))......-------..........------ ( -36.60) >DroYak_CAF1 11764 94 + 1 GCACUGGUGGCAGCCACGGCAUCAGCAUCGCUGCCCUCGCUUAAUCAUAGUGGUGGCGGUCACGGCG-CGUCCACGGGUG-----GUCAC-------CAGAACCAUC------ ...((((((((.(((.((......((...((((((.(((((.......))))).))))))....)).-......))))).-----)))))-------))).......------ ( -38.02) >consensus GCACUGGUGGCUGCCACGGCAUCAGCUUCGCUGCCCUCGCUUAAUCAUAGUGGUGGCGGUCACAGUG_CAUCCACGGGGG_____GUCAC_______CAAAACCAUC______ ((((((.((((.(((((((((.(......).))))..((((.......))))))))).))))))))).)............................................ (-23.58 = -24.25 + 0.67)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:46:05 2006