
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 7,535,043 – 7,535,269 |
| Length | 226 |
| Max. P | 0.765508 |

| Location | 7,535,043 – 7,535,157 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 90.56 |
| Mean single sequence MFE | -36.80 |
| Consensus MFE | -28.58 |
| Energy contribution | -29.22 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.51 |
| SVM RNA-class probability | 0.765508 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
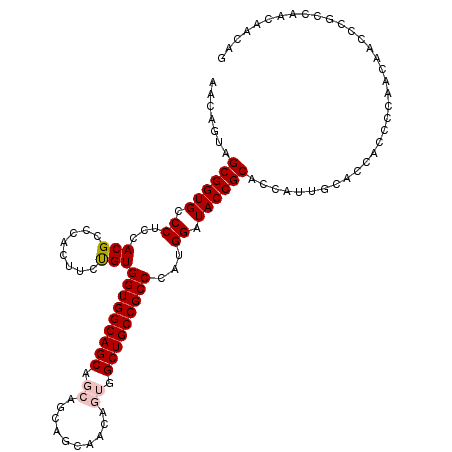
>X_DroMel_CAF1 7535043 114 - 22224390 AACAGUAGCGGUGCCCUCCACGACCACUUCUGUGGUGGCAGCAGCAGCAGCAACAGUGGCUGCCGCCCAUGGAUACCGCACCAUUGCACCACCCCAACAACCCGCCAACAACAG ....((((.(((((..((((.((((((....)))))(((.(((((.((.......)).))))).)))).))))....))))).))))........................... ( -37.60) >DroSec_CAF1 28275 114 - 1 AACAGUAGCGGUGCCCUCCACGCCCACUUCUGUGGUGGCAGCAGCAGCAGCAACAGUGGCUGCCGCCCAUGGAUACCGCACCAUUGCACCGACCCAACAACCCGCCAACAACAG ....((((.(((((..((((...((((....)))).(((.(((((.((.......)).))))).)))..))))....))))).))))........................... ( -35.70) >DroSim_CAF1 28576 114 - 1 AACAGUAGCGGUGCCCUCCACGCCCACUUCUGUGGUGGCAGCAGCAGCAGCAACAGUGGCUGCCGCCCAUGGAUACCGCACCAUUGCACCGACCCAACAACCCGCCAACAACAG ....((((.(((((..((((...((((....)))).(((.(((((.((.......)).))))).)))..))))....))))).))))........................... ( -35.70) >DroEre_CAF1 38782 108 - 1 CACGGUAGCGGUGGCCUCAACGUCCACUUCCGUGGUGGCAGC------AGGAACAGUGGCUGCCGCCCAUGGAUACCGCACCAUUGCACAACUCCAACAACCCGCCAACAGCAG ...(((.(((((((.(.....).))...((((((((((((((------..........))))))).))))))).)))))))).....................((.....)).. ( -37.60) >DroYak_CAF1 28969 108 - 1 CACAGUAGCGGUGGCCUCCACGCCCACUCCUGUGGUGGCAGC------AGCAACAGUGGCUGCCGCCCAUGGAUACCGCACCAUUGCACAACCCCAACAACCCGCCAAUAGCAG .......((((((((......)))...(((((.(((((((((------.((....)).))))))))))).))).)))))........................((.....)).. ( -37.40) >consensus AACAGUAGCGGUGCCCUCCACGCCCACUUCUGUGGUGGCAGCAGCAGCAGCAACAGUGGCUGCCGCCCAUGGAUACCGCACCAUUGCACCACCCCAACAACCCGCCAACAACAG .......((((((.((...(((........)))(((((((((.((..........)).)))))))))...)).))))))................................... (-28.58 = -29.22 + 0.64)

| Location | 7,535,157 – 7,535,269 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 83.97 |
| Mean single sequence MFE | -36.46 |
| Consensus MFE | -27.03 |
| Energy contribution | -28.53 |
| Covariance contribution | 1.50 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.23 |
| SVM RNA-class probability | 0.645327 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
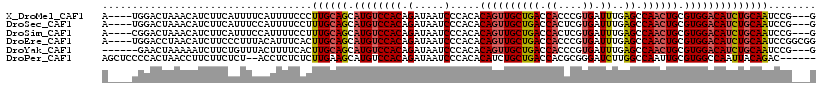
>X_DroMel_CAF1 7535157 112 + 22224390 C---CGGAUUGCAGAUGUCCACGCAGUUGGCUCAAAUCACGGGUGGUCAGCAACUGUGUGGGAUUAUCUGUGGACAUGCUGCAAGGGAAAAUGAAAAUGAAGAUGUUUAGUCCA----U .---.((((((((((((((((((((((((.((...((((....)))).))))))))))))))..)))))))((((((.((.((..............)).))))))))))))).----. ( -36.94) >DroSec_CAF1 28389 112 + 1 C---CGGAUUGCAGAUGUCCACGCAGUUGGCUCAAAUCACGAGUGGUCAGCAACUGUGUGGGAUUAUCUGUGGACAUGCUGCAAAGGAAAAUGGAAAUGAAGAUGUUUAGUCCA----U (---(...(((((((((((((((((((((.((...((((....)))).)))))))))...((.....)))))))))).)))))).))...(((((..((((....)))).))))----) ( -37.20) >DroSim_CAF1 28690 112 + 1 C---CGGAUUGCAGAUGUCCACGCAGUUGGCUCAAAUCACGAGUGGUCAGCAACUGUGUGGGAUUAUCUGUGGACAUGCUGCAAAGGAAAAUGGAAAUGAAGAUGUUUAGUCCG----U .---(((((((((((((((((((((((((.((...((((....)))).))))))))))))))..)))))))((((((.((.((..............)).))))))))))))))----. ( -38.24) >DroEre_CAF1 38890 115 + 1 CCGCCGGAUUGCAGAUGUCCACGCAGUUGGCUCAAAUCACGGGUGGUCAGCAACUGUGUGGGAUUAUCUGUGGACAUGCUGCAAGUGAAAUGUAAAGGGAAGAUGUUAGGUCCA----U .....((((((((((((((((((((((((.((...((((....)))).))))))))))))))..))))))).(((((.((.(...(........)...).))))))).))))).----. ( -36.40) >DroYak_CAF1 29077 110 + 1 C---CGGAUUGCAGAUGUCCACGCAGUUGGCUCAAAUCACGGGUGGUCAGCAACUGUGUGGGAUUAUCUGUGGACAUGCUGCAAGUGAAAAGUAAACAGAAGAUUUUUAGUUC------ .---....(((((((((((((((((((((.((...((((....)))).)))))))))...((.....)))))))))).)))))).............................------ ( -32.40) >DroPer_CAF1 93630 111 + 1 ------GUCUGUAAUUGGCCACGCAAUUGGCCAAGAUCCCGCGUGGUCAGCAGAUGUGUGGGAUUAUCUGUGGACAUGCUUCAAGAGAGAGGU--AGAGAAGAAGGUUAGUGGGGAGCU ------........(((((((......)))))))..((((((...(((.(((((((........))))))).))).((((((......)))))--).............)))))).... ( -37.60) >consensus C___CGGAUUGCAGAUGUCCACGCAGUUGGCUCAAAUCACGGGUGGUCAGCAACUGUGUGGGAUUAUCUGUGGACAUGCUGCAAGGGAAAAGGAAAAUGAAGAUGUUUAGUCCA____U .....((((((((((((((((((((((((.((...((((....)))).))))))))))))))..)))))))((((((.((....................)))))))))))))...... (-27.03 = -28.53 + 1.50)



| Location | 7,535,157 – 7,535,269 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 83.97 |
| Mean single sequence MFE | -27.22 |
| Consensus MFE | -21.95 |
| Energy contribution | -22.40 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.83 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.36 |
| SVM RNA-class probability | 0.704211 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
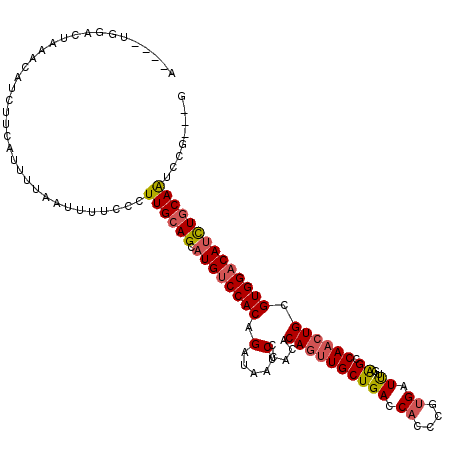
>X_DroMel_CAF1 7535157 112 - 22224390 A----UGGACUAAACAUCUUCAUUUUCAUUUUCCCUUGCAGCAUGUCCACAGAUAAUCCCACACAGUUGCUGACCACCCGUGAUUUGAGCCAACUGCGUGGACAUCUGCAAUCCG---G .----((((..........................((((((.((((((((.(.....).....((((((((.(.((....))...).)).)))))).))))))))))))))))))---. ( -27.26) >DroSec_CAF1 28389 112 - 1 A----UGGACUAAACAUCUUCAUUUCCAUUUUCCUUUGCAGCAUGUCCACAGAUAAUCCCACACAGUUGCUGACCACUCGUGAUUUGAGCCAACUGCGUGGACAUCUGCAAUCCG---G (----((((...............)))))...((.((((((.((((((((.(.....).....((((((((.(.((....))...).)).)))))).))))))))))))))...)---) ( -28.96) >DroSim_CAF1 28690 112 - 1 A----CGGACUAAACAUCUUCAUUUCCAUUUUCCUUUGCAGCAUGUCCACAGAUAAUCCCACACAGUUGCUGACCACUCGUGAUUUGAGCCAACUGCGUGGACAUCUGCAAUCCG---G .----((((..........................((((((.((((((((.(.....).....((((((((.(.((....))...).)).)))))).))))))))))))))))))---. ( -29.46) >DroEre_CAF1 38890 115 - 1 A----UGGACCUAACAUCUUCCCUUUACAUUUCACUUGCAGCAUGUCCACAGAUAAUCCCACACAGUUGCUGACCACCCGUGAUUUGAGCCAACUGCGUGGACAUCUGCAAUCCGGCGG .----((((..........................((((((.((((((((.(.....).....((((((((.(.((....))...).)).)))))).)))))))))))))))))).... ( -27.26) >DroYak_CAF1 29077 110 - 1 ------GAACUAAAAAUCUUCUGUUUACUUUUCACUUGCAGCAUGUCCACAGAUAAUCCCACACAGUUGCUGACCACCCGUGAUUUGAGCCAACUGCGUGGACAUCUGCAAUCCG---G ------.............................((((((.((((((((.(.....).....((((((((.(.((....))...).)).)))))).))))))))))))))....---. ( -26.70) >DroPer_CAF1 93630 111 - 1 AGCUCCCCACUAACCUUCUUCUCU--ACCUCUCUCUUGAAGCAUGUCCACAGAUAAUCCCACACAUCUGCUGACCACGCGGGAUCUUGGCCAAUUGCGUGGCCAAUUACAGAC------ .(.((((.................--...((......)).((.((..(((((((..........))))).))..)).)))))).)(((((((......)))))))........------ ( -23.70) >consensus A____UGGACUAAACAUCUUCAUUUUAAUUUUCCCUUGCAGCAUGUCCACAGAUAAUCCCACACAGUUGCUGACCACCCGUGAUUUGAGCCAACUGCGUGGACAUCUGCAAUCCG___G ...................................((((((.((((((((.(.....).....((((((((((.((....)).))..)).)))))).))))))))))))))........ (-21.95 = -22.40 + 0.45)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:44:12 2006