
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 7,491,226 – 7,491,432 |
| Length | 206 |
| Max. P | 0.982680 |

| Location | 7,491,226 – 7,491,333 |
|---|---|
| Length | 107 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.82 |
| Mean single sequence MFE | -40.58 |
| Consensus MFE | -26.86 |
| Energy contribution | -26.46 |
| Covariance contribution | -0.40 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.63 |
| Structure conservation index | 0.66 |
| SVM decision value | 1.92 |
| SVM RNA-class probability | 0.982680 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7491226 107 + 22224390 -----UUUUG--------GAAAGCGACAGCGACAAGCUGCUGAACAGCUCCAUUGUUGCAGCGGCCAUAACGCUGCAGCAGCAGAACGGCAGCAAUCUGCUGGCCAACACAAAUACGCCG -----..(((--------(..((((..(((.....)))((((....(.((..(((((((((((.......)))))))))))..)).)..))))....))))..))))............. ( -38.00) >DroVir_CAF1 77893 117 + 1 GAAGCCACUGGCAACAGCGAAAGCGACAGCGACAAGCUGCUGAAUAGCUCGAUUGUUGCAGCGGCCAUAACGCUGCA---ACAGAACGGCGGCAAUCUGCUGGCCAACACAAACACACCG ........((((....((....))..(((((....((((((((.....))..(((((((((((.......)))))))---))))...))))))....))))))))).............. ( -44.70) >DroGri_CAF1 72283 117 + 1 GAAGCCACUGGAAACAGCGAAAGUGAGAGCGACAAGCUGCUGAAUAGCUCGAUUGUUGCAGCGGCCAUAACGCUGCA---ACAGAACGGCGGCAAUCUGCUAGCCAACACAAAUACACCG .....(.(((....))).)...(((.((((..((......))....))))..(((((((((((.......)))))))---))))...((((((.....))).)))..))).......... ( -40.10) >DroMoj_CAF1 58053 117 + 1 GAAGCCACUGGAGGAAGCGAGGGCGAGAGCGACAAGCUGUUGAAUAGCUCGAUUGUUGCAGCGGCCAUAACGCUGCA---ACAGAACGGCGGCAAUCUGCUGGCCAACACAAAUACACCG ...(((.((.(......).)))))(((..((((.....)))).....)))..(((((((((((.......)))))))---))))...((((((.....))).)))............... ( -39.60) >DroPer_CAF1 42736 101 + 1 GA-GGAUCUG--------GAUC-------UGGCCAUCAGCUGAAUAGCUCCAUUGUUGCAGCGGCCAUCACGCUGCA---GCAGAACGGCGGCAAUCUGCUGGCCAACACCAAUACGCCG ..-((...((--------(...-------((((((.(((.((....(((...(((((((((((.......)))))))---))))...)))..))..))).))))))...))).....)). ( -40.50) >consensus GAAGCCACUGG____AGCGAAAGCGACAGCGACAAGCUGCUGAAUAGCUCGAUUGUUGCAGCGGCCAUAACGCUGCA___ACAGAACGGCGGCAAUCUGCUGGCCAACACAAAUACACCG .......(((..................(((((((...(((....)))....))))))).(((((......))))).....)))...((((((.....))).)))............... (-26.86 = -26.46 + -0.40)

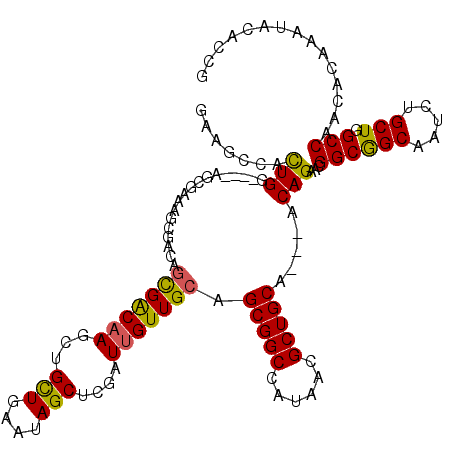

| Location | 7,491,253 – 7,491,373 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 85.75 |
| Mean single sequence MFE | -45.35 |
| Consensus MFE | -29.59 |
| Energy contribution | -29.84 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.08 |
| Mean z-score | -3.14 |
| Structure conservation index | 0.65 |
| SVM decision value | -0.06 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7491253 120 + 22224390 UGAACAGCUCCAUUGUUGCAGCGGCCAUAACGCUGCAGCAGCAGAACGGCAGCAAUCUGCUGGCCAACACAAAUACGCCGUCGCCCUCGCCGCCGUUGCUCAGCGCCGAGCAGCAGCAGC .((...((((..(((((((((((.......)))))))))))..))..((((((.....))).)))...........))..))......((.((.(((((((......))))))).)).)) ( -45.60) >DroVir_CAF1 77933 117 + 1 UGAAUAGCUCGAUUGUUGCAGCGGCCAUAACGCUGCA---ACAGAACGGCGGCAAUCUGCUGGCCAACACAAACACACCGUCGCCAUCGCCGCCGUUGCUGAGCGCUGAGCAGCAGCAAC ......(((((.(((((((((((.......)))))))---))))(((((((((.....(.((((..((...........)).)))).))))))))))..)))))((((.....))))... ( -50.40) >DroGri_CAF1 72323 117 + 1 UGAAUAGCUCGAUUGUUGCAGCGGCCAUAACGCUGCA---ACAGAACGGCGGCAAUCUGCUAGCCAACACAAAUACACCGUCGCCAUCGCCGCCGUUGCUGAGUGCCGAGCAGCAGCAAC ......(((((.(((((((((((.......)))))))---))))(((((((((.....((......................))....)))))))))((.....)))))))......... ( -44.05) >DroWil_CAF1 4 94 + 1 --------------------------AUAACGCUGCAACAACAGAACGGCGGCAAUCUGCUGGCCAAUACCAACACACCAUCGCCAUCGCCGCCGUUGCUGAGCGCCGAGCAGCAGCAAC --------------------------.....(((((.....((((((((((((.....(.((((..................)))).)))))))))).))).((.....)).)))))... ( -35.37) >DroMoj_CAF1 58093 117 + 1 UGAAUAGCUCGAUUGUUGCAGCGGCCAUAACGCUGCA---ACAGAACGGCGGCAAUCUGCUGGCCAACACAAAUACACCGUCGCCAUCGCCGCCGUUGCUCAGCGCCGAGCAGCAGCAAC ......(((...(((((((((((.......)))))))---))))...((((((.....(.((((..((...........)).)))).)))))))(((((((......))))))))))... ( -47.50) >DroPer_CAF1 42760 117 + 1 UGAAUAGCUCCAUUGUUGCAGCGGCCAUCACGCUGCA---GCAGAACGGCGGCAAUCUGCUGGCCAACACCAAUACGCCGUCGCCCUCGCCGCCGUUGCUCAGCGCCGAGCAGCAGCAAC ......(((...(((((((((((.......)))))))---))))...((((((.....((((((............))))..))....))))))(((((((......))))))))))... ( -49.20) >consensus UGAAUAGCUCGAUUGUUGCAGCGGCCAUAACGCUGCA___ACAGAACGGCGGCAAUCUGCUGGCCAACACAAAUACACCGUCGCCAUCGCCGCCGUUGCUCAGCGCCGAGCAGCAGCAAC ....((((......)))).............(((((........(((((((((.....(((((..............)))..))....)))))))))(((........))).)))))... (-29.59 = -29.84 + 0.25)

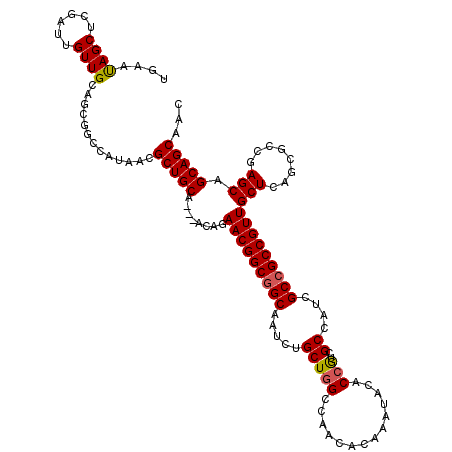
| Location | 7,491,253 – 7,491,373 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 85.75 |
| Mean single sequence MFE | -52.75 |
| Consensus MFE | -38.83 |
| Energy contribution | -39.75 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.75 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.663850 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7491253 120 - 22224390 GCUGCUGCUGCUCGGCGCUGAGCAACGGCGGCGAGGGCGACGGCGUAUUUGUGUUGGCCAGCAGAUUGCUGCCGUUCUGCUGCUGCAGCGUUAUGGCCGCUGCAACAAUGGAGCUGUUCA (((((((.((((((....)))))).)))))))..(((((.(((((.((((((........))))))))))).)(((((..((.(((((((.......))))))).))..))))).)))). ( -54.60) >DroVir_CAF1 77933 117 - 1 GUUGCUGCUGCUCAGCGCUCAGCAACGGCGGCGAUGGCGACGGUGUGUUUGUGUUGGCCAGCAGAUUGCCGCCGUUCUGU---UGCAGCGUUAUGGCCGCUGCAACAAUCGAGCUAUUCA (((((((..((.....)).)))))))(((((((((.((..(((..(....)..)))....))..)))))))))(((((((---(((((((.......))))))))))...))))...... ( -53.00) >DroGri_CAF1 72323 117 - 1 GUUGCUGCUGCUCGGCACUCAGCAACGGCGGCGAUGGCGACGGUGUAUUUGUGUUGGCUAGCAGAUUGCCGCCGUUCUGU---UGCAGCGUUAUGGCCGCUGCAACAAUCGAGCUAUUCA (((((((.(((...)))..)))))))(((((((((.((..(((..(....)..)))....))..)))))))))(((((((---(((((((.......))))))))))...))))...... ( -51.90) >DroWil_CAF1 4 94 - 1 GUUGCUGCUGCUCGGCGCUCAGCAACGGCGGCGAUGGCGAUGGUGUGUUGGUAUUGGCCAGCAGAUUGCCGCCGUUCUGUUGUUGCAGCGUUAU-------------------------- ...((....))..((((((..((((((((((.(((((((..(((.(((((((....))))))).)))..)))))))))))))))))))))))..-------------------------- ( -46.60) >DroMoj_CAF1 58093 117 - 1 GUUGCUGCUGCUCGGCGCUGAGCAACGGCGGCGAUGGCGACGGUGUAUUUGUGUUGGCCAGCAGAUUGCCGCCGUUCUGU---UGCAGCGUUAUGGCCGCUGCAACAAUCGAGCUAUUCA (((((((.((((((....)))))).)))))))(((((((((((((.((((((........))))))...))))))).(((---(((((((.......)))))))))).....)))))).. ( -54.20) >DroPer_CAF1 42760 117 - 1 GUUGCUGCUGCUCGGCGCUGAGCAACGGCGGCGAGGGCGACGGCGUAUUGGUGUUGGCCAGCAGAUUGCCGCCGUUCUGC---UGCAGCGUGAUGGCCGCUGCAACAAUGGAGCUAUUCA (((((.((.(((..((((((((((((((((((((((.(((((.(.....).))))).))......))))))))))..)))---).))))))...))).)).)))))..((((....)))) ( -56.20) >consensus GUUGCUGCUGCUCGGCGCUCAGCAACGGCGGCGAUGGCGACGGUGUAUUUGUGUUGGCCAGCAGAUUGCCGCCGUUCUGU___UGCAGCGUUAUGGCCGCUGCAACAAUCGAGCUAUUCA (((((.((.(((..(((((.((.(((((((((((..((...(((..((....))..))).))...)))))))))))))((....)))))))...))).)).))))).............. (-38.83 = -39.75 + 0.92)



| Location | 7,491,293 – 7,491,403 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 90.16 |
| Mean single sequence MFE | -41.71 |
| Consensus MFE | -31.18 |
| Energy contribution | -31.62 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.586084 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7491293 110 + 22224390 GCAGAACGGCAGCAAUCUGCUGGCCAACACAAAUACGCCGUCGCCCUCGCCGCCGUUGCUCAGCGCCGAGCAGCAGCAGCAGUUGCAGAGCAGUUUGCAACAGAGCGGCG ...((.(((((((.....)))(.....)........))))))((((((((.((.(((((((......))))))).)).)).((((((((....)))))))).))).))). ( -46.20) >DroVir_CAF1 77970 108 + 1 ACAGAACGGCGGCAAUCUGCUGGCCAACACAAACACACCGUCGCCAUCGCCGCCGUUGCUGAGCGCUGAGCAGCAGCAACAGUUGCAGAGCAGCCUGCAGAACGGCGU-- .......((((((.....))((((..((...........)).)))).))))((((((.(((((.((((..(.(((((....))))).)..)))))).)))))))))..-- ( -41.30) >DroGri_CAF1 72360 108 + 1 ACAGAACGGCGGCAAUCUGCUAGCCAACACAAAUACACCGUCGCCAUCGCCGCCGUUGCUGAGUGCCGAGCAGCAGCAACAGUUGCAGAGCAGCCUGCAGAACGGCGU-- .......((((((..........................))))))..(((((..(((((((..(((...))).)))))))..((((((......))))))..))))).-- ( -37.47) >DroMoj_CAF1 58130 110 + 1 ACAGAACGGCGGCAAUCUGCUGGCCAACACAAAUACACCGUCGCCAUCGCCGCCGUUGCUCAGCGCCGAGCAGCAGCAACAGUUGCAGAGCAGCCUGCAAAACGGCGUCG ....(((((((((.....(.((((..((...........)).)))).))))))))))((((.((.....)).(((((....))))).)))).(((........))).... ( -39.30) >DroPer_CAF1 42797 110 + 1 GCAGAACGGCGGCAAUCUGCUGGCCAACACCAAUACGCCGUCGCCCUCGCCGCCGUUGCUCAGCGCCGAGCAGCAGCAACAGUUGCAGAGCAGCCUGCAGCAGAACGGUG ((.((.(((((((.....))(((......)))...)))))))))...((((((.(((((((......))))))).))....(((((((......))))))).....)))) ( -44.30) >consensus ACAGAACGGCGGCAAUCUGCUGGCCAACACAAAUACACCGUCGCCAUCGCCGCCGUUGCUCAGCGCCGAGCAGCAGCAACAGUUGCAGAGCAGCCUGCAGAACGGCGU_G .......((((((.....))).)))......................((((((.(((((((......))))))).)).....((((((......))))))...))))... (-31.18 = -31.62 + 0.44)

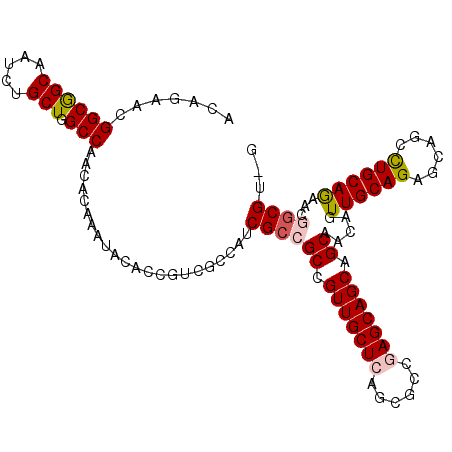

| Location | 7,491,293 – 7,491,403 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 90.16 |
| Mean single sequence MFE | -47.28 |
| Consensus MFE | -44.78 |
| Energy contribution | -43.66 |
| Covariance contribution | -1.12 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.27 |
| Structure conservation index | 0.95 |
| SVM decision value | 0.51 |
| SVM RNA-class probability | 0.765165 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7491293 110 - 22224390 CGCCGCUCUGUUGCAAACUGCUCUGCAACUGCUGCUGCUGCUCGGCGCUGAGCAACGGCGGCGAGGGCGACGGCGUAUUUGUGUUGGCCAGCAGAUUGCUGCCGUUCUGC ((((.(((.((((((........)))))).(((((((.((((((....)))))).))))))))))))))(((((((((((((........))))).))).)))))..... ( -51.60) >DroVir_CAF1 77970 108 - 1 --ACGCCGUUCUGCAGGCUGCUCUGCAACUGUUGCUGCUGCUCAGCGCUCAGCAACGGCGGCGAUGGCGACGGUGUGUUUGUGUUGGCCAGCAGAUUGCCGCCGUUCUGU --.(((((...((((((....)))))).(((((((((..((.....)).))))))))))))))((((((.((((.((((.((....)).)))).)))).))))))..... ( -46.90) >DroGri_CAF1 72360 108 - 1 --ACGCCGUUCUGCAGGCUGCUCUGCAACUGUUGCUGCUGCUCGGCACUCAGCAACGGCGGCGAUGGCGACGGUGUAUUUGUGUUGGCUAGCAGAUUGCCGCCGUUCUGU --..((((....((((.(.((...((....)).)).))))).))))...(((.((((((((((((.((..(((..(....)..)))....))..))))))))))))))). ( -44.60) >DroMoj_CAF1 58130 110 - 1 CGACGCCGUUUUGCAGGCUGCUCUGCAACUGUUGCUGCUGCUCGGCGCUGAGCAACGGCGGCGAUGGCGACGGUGUAUUUGUGUUGGCCAGCAGAUUGCCGCCGUUCUGU ...((((((((((((((....)))))))..(((((((.((((((....)))))).))))))))))))))((((((.((((((........))))))...))))))..... ( -47.70) >DroPer_CAF1 42797 110 - 1 CACCGUUCUGCUGCAGGCUGCUCUGCAACUGUUGCUGCUGCUCGGCGCUGAGCAACGGCGGCGAGGGCGACGGCGUAUUGGUGUUGGCCAGCAGAUUGCCGCCGUUCUGC ....((...((.((.(((...(((((...((((((((.((((((....)))))).)))))))).((.(((((.(.....).))))).)).)))))..))))).))...)) ( -45.60) >consensus C_ACGCCGUUCUGCAGGCUGCUCUGCAACUGUUGCUGCUGCUCGGCGCUGAGCAACGGCGGCGAUGGCGACGGUGUAUUUGUGUUGGCCAGCAGAUUGCCGCCGUUCUGU ...((((((..((((((....))))))..((((((((.((((((....)))))).))))))))))))))((((((.((((((........))))))...))))))..... (-44.78 = -43.66 + -1.12)

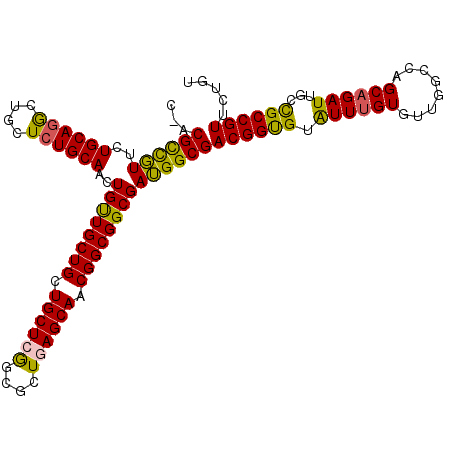
| Location | 7,491,333 – 7,491,432 |
|---|---|
| Length | 99 |
| Sequences | 5 |
| Columns | 102 |
| Reading direction | forward |
| Mean pairwise identity | 84.34 |
| Mean single sequence MFE | -44.66 |
| Consensus MFE | -34.88 |
| Energy contribution | -34.68 |
| Covariance contribution | -0.20 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.21 |
| Structure conservation index | 0.78 |
| SVM decision value | 1.29 |
| SVM RNA-class probability | 0.940592 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7491333 99 + 22224390 UCGCCCUCGCCGCCGUUGCUCAGCGCCGAGCAGCAGCAGCAGUUGCAGAGCAGUUUGCAACAGAGCGGCG---GUGUUGGCGGCGCCUGCCUCAAUCCCAAG ..((((((((.((.(((((((......))))))).)).)).((((((((....)))))))).))).)))(---(..((((.(((....)))))))..))... ( -47.10) >DroVir_CAF1 78010 93 + 1 UCGCCAUCGCCGCCGUUGCUGAGCGCUGAGCAGCAGCAACAGUUGCAGAGCAGCCUGCAGAACGGCGU---------CGGCGGCGCCUGUUUAAAUCCCAAG .((((..((.(((((((.(((((.((((..(.(((((....))))).)..)))))).)))))))))).---------))..))))................. ( -42.10) >DroGri_CAF1 72400 93 + 1 UCGCCAUCGCCGCCGUUGCUGAGUGCCGAGCAGCAGCAACAGUUGCAGAGCAGCCUGCAGAACGGCGU---------CGGUGGCGCUUGUUUAAAUCCCAAG .((((((((.(((((((((((..(((...))).)))))))..((((((......))))))...)))).---------))))))))................. ( -43.00) >DroMoj_CAF1 58170 99 + 1 UCGCCAUCGCCGCCGUUGCUCAGCGCCGAGCAGCAGCAACAGUUGCAGAGCAGCCUGCAAAACGGCGUCG---GCGUCGGCGGCGCUUGCUUGAAUCCCAAG ..((...((((((((.((((..((((((.((....)).....((((((......))))))..)))))).)---))).))))))))...))............ ( -45.00) >DroPer_CAF1 42837 102 + 1 UCGCCCUCGCCGCCGUUGCUCAGCGCCGAGCAGCAGCAACAGUUGCAGAGCAGCCUGCAGCAGAACGGUGGUGGUGUCGGCGGCGCCUGCCUCAAUCCGAAG (((((..((((((((((((((......))))))).......(((((((......)))))))........)))))))..)))(((....))).......)).. ( -46.10) >consensus UCGCCAUCGCCGCCGUUGCUCAGCGCCGAGCAGCAGCAACAGUUGCAGAGCAGCCUGCAGAACGGCGU_G___G_GUCGGCGGCGCCUGCUUAAAUCCCAAG ..((...((((((((((((((......))))))).((.....((((((......))))))....))............)))))))...))............ (-34.88 = -34.68 + -0.20)

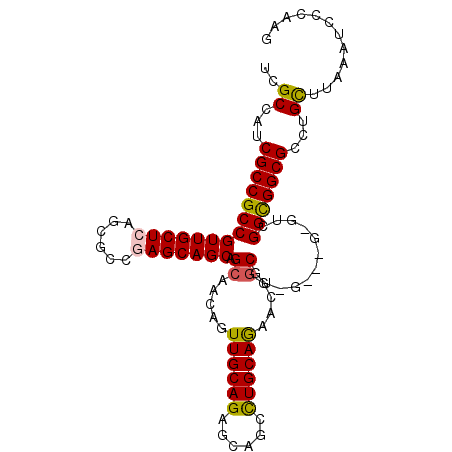

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:44:03 2006