
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 7,480,132 – 7,480,280 |
| Length | 148 |
| Max. P | 0.985652 |

| Location | 7,480,132 – 7,480,240 |
|---|---|
| Length | 108 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.25 |
| Mean single sequence MFE | -43.21 |
| Consensus MFE | -34.62 |
| Energy contribution | -34.44 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.98 |
| Structure conservation index | 0.80 |
| SVM decision value | 1.33 |
| SVM RNA-class probability | 0.942899 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7480132 108 + 22224390 GCACGUGGCCAGUUUAUCACUGUAUGGGUUAAUAUCUGUGUGCGUCUG------------UGUCUGUGUGAUGUGCAAAUGAUACACUCGCUCACACACAGCCAGAGACGCACAGUCACC ....(((((((((.....)))).((((((....))))))((((((((.------------((.(((((((.((.((.............)).))))))))).)).)))))))).))))). ( -41.22) >DroSec_CAF1 13275 108 + 1 GCACGUGGCCAGUUUGUCACUGUAUGGGUUAAUAUCUGUGUGCGUCUG------------UGUCUGUGUGAUGUGCAAAUGAUACACUCGCUCACACACAGUCAGAGACGCACAGUCACC ....(((((((((.....)))).((((((....))))))((((((((.------------((.(((((((.((.((.............)).))))))))).)).)))))))).))))). ( -42.32) >DroSim_CAF1 13752 108 + 1 GCACGUGGCCAGUUUGUCACUGUAUGGGUUAAUAUCUGUGUGCGUCUG------------UGUCUGUGUGAUGUGCAAAUGAUACACUCGCUCACACACAGCCAGAGACGCACAGUCACC ....(((((((((.....)))).((((((....))))))((((((((.------------((.(((((((.((.((.............)).))))))))).)).)))))))).))))). ( -41.22) >DroEre_CAF1 14168 120 + 1 GCACGUGGCCAGUUUGUCGCUGUAUGGGUUAGUAUCUGUGUGCGUCUGCGUCUGUGCGCGUGUCUGUGUGAUGUGCAAAUGAUACACGCGCUCACACACAGGCAGAGACGCACAGUCACC ....(((((((((.....)))).((((((....))))))((((((((((((....)))).((((((((((..((((...........))))...)))))))))).)))))))).))))). ( -48.10) >consensus GCACGUGGCCAGUUUGUCACUGUAUGGGUUAAUAUCUGUGUGCGUCUG____________UGUCUGUGUGAUGUGCAAAUGAUACACUCGCUCACACACAGCCAGAGACGCACAGUCACC ....(((((((((.....)))).((((((....))))))((((((((.............((.(((((((.((.((.............)).))))))))).)).)))))))).))))). (-34.62 = -34.44 + -0.19)

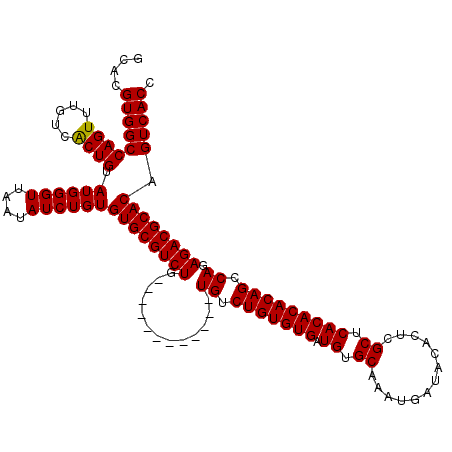
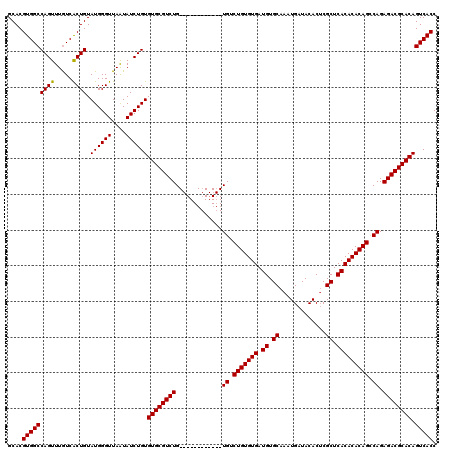
| Location | 7,480,132 – 7,480,240 |
|---|---|
| Length | 108 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.25 |
| Mean single sequence MFE | -42.93 |
| Consensus MFE | -36.89 |
| Energy contribution | -37.39 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.68 |
| Structure conservation index | 0.86 |
| SVM decision value | 2.01 |
| SVM RNA-class probability | 0.985652 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7480132 108 - 22224390 GGUGACUGUGCGUCUCUGGCUGUGUGUGAGCGAGUGUAUCAUUUGCACAUCACACAGACA------------CAGACGCACACAGAUAUUAACCCAUACAGUGAUAAACUGGCCACGUGC (((...(((((((((.((.(((((((((.(((((((...))))))).)).))))))).))------------.)))))))))................((((.....)))))))...... ( -43.90) >DroSec_CAF1 13275 108 - 1 GGUGACUGUGCGUCUCUGACUGUGUGUGAGCGAGUGUAUCAUUUGCACAUCACACAGACA------------CAGACGCACACAGAUAUUAACCCAUACAGUGACAAACUGGCCACGUGC (((...(((((((((.((.(((((((((.(((((((...))))))).)).))))))).))------------.)))))))))................((((.....)))))))...... ( -43.50) >DroSim_CAF1 13752 108 - 1 GGUGACUGUGCGUCUCUGGCUGUGUGUGAGCGAGUGUAUCAUUUGCACAUCACACAGACA------------CAGACGCACACAGAUAUUAACCCAUACAGUGACAAACUGGCCACGUGC (((...(((((((((.((.(((((((((.(((((((...))))))).)).))))))).))------------.)))))))))................((((.....)))))))...... ( -43.90) >DroEre_CAF1 14168 120 - 1 GGUGACUGUGCGUCUCUGCCUGUGUGUGAGCGCGUGUAUCAUUUGCACAUCACACAGACACGCGCACAGACGCAGACGCACACAGAUACUAACCCAUACAGCGACAAACUGGCCACGUGC (((..(((((.((.((((((((((((((.....(((((.....))))).((.....))..)))))))))..))))).)).))))).............(((.......))))))...... ( -40.40) >consensus GGUGACUGUGCGUCUCUGGCUGUGUGUGAGCGAGUGUAUCAUUUGCACAUCACACAGACA____________CAGACGCACACAGAUAUUAACCCAUACAGUGACAAACUGGCCACGUGC (((...(((((((((.((.(((((((((.(((((((...))))))).)).))))))).)).............)))))))))................((((.....)))))))...... (-36.89 = -37.39 + 0.50)

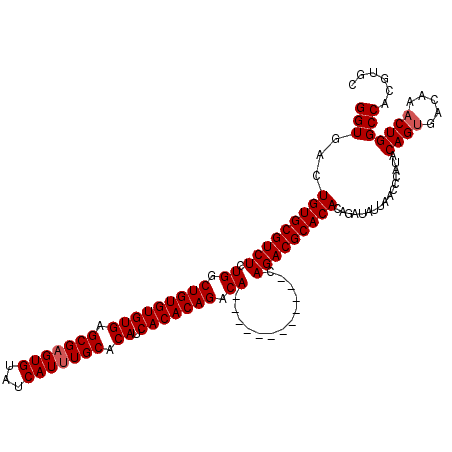

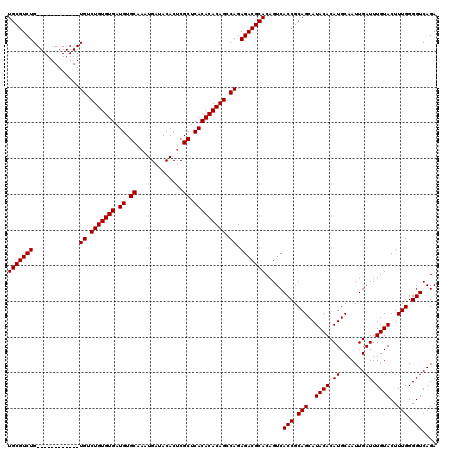
| Location | 7,480,172 – 7,480,280 |
|---|---|
| Length | 108 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.57 |
| Mean single sequence MFE | -38.52 |
| Consensus MFE | -28.96 |
| Energy contribution | -28.96 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.652842 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7480172 108 + 22224390 UGCGUCUG------------UGUCUGUGUGAUGUGCAAAUGAUACACUCGCUCACACACAGCCAGAGACGCACAGUCACCGCAGCAUACACAUGCAAUUGAUUUGUACUUUGGGGUCAGA (((((((.------------((.(((((((.((.((.............)).))))))))).)).))))))).....(((.(((..((((.((.......)).))))..))).))).... ( -35.82) >DroSec_CAF1 13315 108 + 1 UGCGUCUG------------UGUCUGUGUGAUGUGCAAAUGAUACACUCGCUCACACACAGUCAGAGACGCACAGUCACCGCAGCAUACACAUGCAAUUGAUUUGUACUUUGGGGUCAGA (((((((.------------((.(((((((.((.((.............)).))))))))).)).))))))).....(((.(((..((((.((.......)).))))..))).))).... ( -36.92) >DroSim_CAF1 13792 108 + 1 UGCGUCUG------------UGUCUGUGUGAUGUGCAAAUGAUACACUCGCUCACACACAGCCAGAGACGCACAGUCACCGCAGCAUACACAUGCAAUUGAUUUGUACUUUGGGGUCAGA (((((((.------------((.(((((((.((.((.............)).))))))))).)).))))))).....(((.(((..((((.((.......)).))))..))).))).... ( -35.82) >DroEre_CAF1 14208 120 + 1 UGCGUCUGCGUCUGUGCGCGUGUCUGUGUGAUGUGCAAAUGAUACACGCGCUCACACACAGGCAGAGACGCACAGUCACCGCAGCAUACACAUGCAAUUGAUUUGUACUUUGGGGUCAGA ((((((((((.((((((((.((((((((((..((((...........))))...))))))))))..).)))))))....))))).......))))).(((((((........))))))). ( -45.51) >consensus UGCGUCUG____________UGUCUGUGUGAUGUGCAAAUGAUACACUCGCUCACACACAGCCAGAGACGCACAGUCACCGCAGCAUACACAUGCAAUUGAUUUGUACUUUGGGGUCAGA (((((((.............((.(((((((.((.((.............)).))))))))).)).))))))).....(((.(((..((((.((.......)).))))..))).))).... (-28.96 = -28.96 + 0.00)


| Location | 7,480,172 – 7,480,280 |
|---|---|
| Length | 108 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.57 |
| Mean single sequence MFE | -39.25 |
| Consensus MFE | -33.14 |
| Energy contribution | -33.39 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.46 |
| SVM RNA-class probability | 0.955893 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7480172 108 - 22224390 UCUGACCCCAAAGUACAAAUCAAUUGCAUGUGUAUGCUGCGGUGACUGUGCGUCUCUGGCUGUGUGUGAGCGAGUGUAUCAUUUGCACAUCACACAGACA------------CAGACGCA ....(((.((..(((((.(((....).)).)))))..)).))).....(((((((.((.(((((((((.(((((((...))))))).)).))))))).))------------.))))))) ( -39.00) >DroSec_CAF1 13315 108 - 1 UCUGACCCCAAAGUACAAAUCAAUUGCAUGUGUAUGCUGCGGUGACUGUGCGUCUCUGACUGUGUGUGAGCGAGUGUAUCAUUUGCACAUCACACAGACA------------CAGACGCA ....(((.((..(((((.(((....).)).)))))..)).))).....(((((((.((.(((((((((.(((((((...))))))).)).))))))).))------------.))))))) ( -38.60) >DroSim_CAF1 13792 108 - 1 UCUGACCCCAAAGUACAAAUCAAUUGCAUGUGUAUGCUGCGGUGACUGUGCGUCUCUGGCUGUGUGUGAGCGAGUGUAUCAUUUGCACAUCACACAGACA------------CAGACGCA ....(((.((..(((((.(((....).)).)))))..)).))).....(((((((.((.(((((((((.(((((((...))))))).)).))))))).))------------.))))))) ( -39.00) >DroEre_CAF1 14208 120 - 1 UCUGACCCCAAAGUACAAAUCAAUUGCAUGUGUAUGCUGCGGUGACUGUGCGUCUCUGCCUGUGUGUGAGCGCGUGUAUCAUUUGCACAUCACACAGACACGCGCACAGACGCAGACGCA (((((((.((..(((((.(((....).)).)))))..)).)))..((((((((...((.(((((((((.(((.(((...))).))).)).))))))).)).))))))))...)))).... ( -40.40) >consensus UCUGACCCCAAAGUACAAAUCAAUUGCAUGUGUAUGCUGCGGUGACUGUGCGUCUCUGGCUGUGUGUGAGCGAGUGUAUCAUUUGCACAUCACACAGACA____________CAGACGCA ....(((.((..(((((.(((....).)).)))))..)).))).....(((((((.((.(((((((((.(((((((...))))))).)).))))))).)).............))))))) (-33.14 = -33.39 + 0.25)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:43:31 2006