
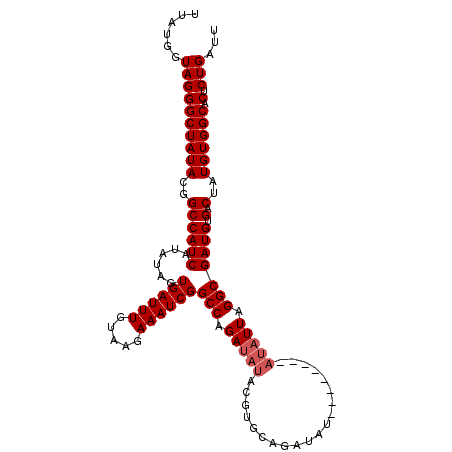
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 7,476,335 – 7,476,517 |
| Length | 182 |
| Max. P | 0.989774 |

| Location | 7,476,335 – 7,476,437 |
|---|---|
| Length | 102 |
| Sequences | 4 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 90.09 |
| Mean single sequence MFE | -27.83 |
| Consensus MFE | -20.15 |
| Energy contribution | -20.90 |
| Covariance contribution | 0.75 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.84 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.629110 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7476335 102 - 22224390 UUAUGGUAGGGCUAUACGGCCAUCAUAUAGUGAUUUGUAAGAAAUCGGCCAGAUAUACGUGCAGAUAU--------AUAUUAGGCGAUGUGACUAUGUGGCACUCUGAUU (((.(((..((((....)))).((((((((((((((.....))))).(((.((((((..(....)..)--------))))).)))......))))))))).))).))).. ( -25.60) >DroSec_CAF1 9561 90 - 1 UUAUGGUAGGGCUAUACGGCCAUCAUAUAGUGAUUUGUAAGAAAUCGGCCAGA------------UAU--------AUAUUAGGCGAUGUGACUAUGUGGCACUCUGAUU (((.(((..((((....)))).((((((((((((((.....))))).(((.((------------(..--------..))).)))......))))))))).))).))).. ( -21.80) >DroSim_CAF1 9099 102 - 1 UUAUGGUAGGGCUAUACGGCCAUCAUAUAGUGAUUUGUAAGAAAUCGGCCAGAUAUACGUGCAGAUAU--------AUAUUAGGCGAUGUGACUAUGUGGCACGCUGAUU .(((((.....)))))((((..((((((((((((((.....))))).(((.((((((..(....)..)--------))))).)))......)))))))))...))))... ( -27.00) >DroEre_CAF1 10272 110 - 1 UUAUGGUAGGGCUAUACGGCCAUCAUAUAGUGAUUUGUAAGAAAUCGGCCAGAUAUACGUGCAGAUAUAUGUAUGUAUAUUAGGCGAUGUGACUAUGUGGCACUCUGAUU (((.(((..((((....)))).((((((((((((((.....))))).(((.((((((((((((......)))))))))))).)))......))))))))).))).))).. ( -36.90) >consensus UUAUGGUAGGGCUAUACGGCCAUCAUAUAGUGAUUUGUAAGAAAUCGGCCAGAUAUACGUGCAGAUAU________AUAUUAGGCGAUGUGACUAUGUGGCACUCUGAUU ......((((((((((..((((((......((((((.....))))))(((.(((((....................))))).))))))).).)..)))))).).)))... (-20.15 = -20.90 + 0.75)

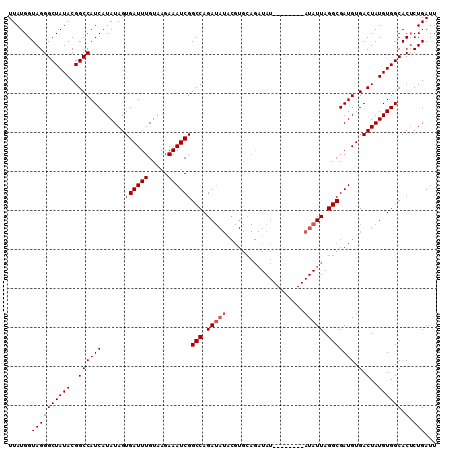
| Location | 7,476,369 – 7,476,477 |
|---|---|
| Length | 108 |
| Sequences | 4 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 93.58 |
| Mean single sequence MFE | -17.30 |
| Consensus MFE | -16.30 |
| Energy contribution | -16.30 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.94 |
| SVM decision value | 1.39 |
| SVM RNA-class probability | 0.949237 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7476369 108 + 22224390 --AUAUCUGCACGUAUAUCUGGCCGAUUUCUUACAAAUCACUAUAUGAUGGCCGUAUAGCCCUACCAUAAAUCUUUCAACAUGUUUAGCCAACAACUAAACAAUUUAUGA --.............(((.((((((((((.....))))).(.....)..))))).))).......(((((((.........(((((((.......)))))))))))))). ( -17.30) >DroSec_CAF1 9595 96 + 1 --AUA------------UCUGGCCGAUUUCUUACAAAUCACUAUAUGAUGGCCGUAUAGCCCUACCAUAAAUCUUUCAACAUGUUUAGCCAACAACUAAACAAUUUAUGA --.((------------(.((((((((((.....))))).(.....)..))))).))).......(((((((.........(((((((.......)))))))))))))). ( -17.30) >DroSim_CAF1 9133 108 + 1 --AUAUCUGCACGUAUAUCUGGCCGAUUUCUUACAAAUCACUAUAUGAUGGCCGUAUAGCCCUACCAUAAAUCUUUCAACAUGUUUAGCCAACAACUAAACAAUUUAUGA --.............(((.((((((((((.....))))).(.....)..))))).))).......(((((((.........(((((((.......)))))))))))))). ( -17.30) >DroEre_CAF1 10312 110 + 1 AUAUAUCUGCACGUAUAUCUGGCCGAUUUCUUACAAAUCACUAUAUGAUGGCCGUAUAGCCCUACCAUAAAUCUUUCAACAUGUUUAGCCAACAACUAAACAAUUUAUGA ...............(((.((((((((((.....))))).(.....)..))))).))).......(((((((.........(((((((.......)))))))))))))). ( -17.30) >consensus __AUAUCUGCACGUAUAUCUGGCCGAUUUCUUACAAAUCACUAUAUGAUGGCCGUAUAGCCCUACCAUAAAUCUUUCAACAUGUUUAGCCAACAACUAAACAAUUUAUGA ....................(((((((((.....))))).(.....)..))))............(((((((.........(((((((.......)))))))))))))). (-16.30 = -16.30 + 0.00)


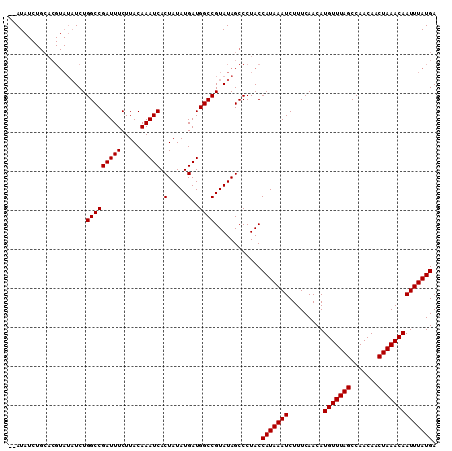
| Location | 7,476,397 – 7,476,517 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.33 |
| Mean single sequence MFE | -24.90 |
| Consensus MFE | -25.06 |
| Energy contribution | -24.74 |
| Covariance contribution | -0.32 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.71 |
| Structure conservation index | 1.01 |
| SVM decision value | 2.18 |
| SVM RNA-class probability | 0.989774 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7476397 120 + 22224390 UUACAAAUCACUAUAUGAUGGCCGUAUAGCCCUACCAUAAAUCUUUCAACAUGUUUAGCCAACAACUAAACAAUUUAUGACGAUUGUCUCCGCCAUGCGGGCGAAUCUUUUUGUAAGUAU (((((((((.(((((((.....)))))))......(((((((.........(((((((.......))))))))))))))..)))).....((((.....))))........))))).... ( -24.40) >DroSec_CAF1 9611 120 + 1 UUACAAAUCACUAUAUGAUGGCCGUAUAGCCCUACCAUAAAUCUUUCAACAUGUUUAGCCAACAACUAAACAAUUUAUGACGAUUGUCUCCGCCAUGCGGGCGAAUCUUUCUGUAAGUAU (((((((((.(((((((.....)))))))......(((((((.........(((((((.......))))))))))))))..)))).....((((.....))))........))))).... ( -24.30) >DroSim_CAF1 9161 120 + 1 UUACAAAUCACUAUAUGAUGGCCGUAUAGCCCUACCAUAAAUCUUUCAACAUGUUUAGCCAACAACUAAACAAUUUAUGACGAUUGUCUCCGCCAUGCGGGCGAAUCUUUCUGUAAGUAU (((((((((.(((((((.....)))))))......(((((((.........(((((((.......))))))))))))))..)))).....((((.....))))........))))).... ( -24.30) >DroEre_CAF1 10342 120 + 1 UUACAAAUCACUAUAUGAUGGCCGUAUAGCCCUACCAUAAAUCUUUCAACAUGUUUAGCCAACAACUAAACAAUUUAUGACGAUUGUCCCCGCCAUGCGGGCGAAUCUUUCUGUAAGUAU (((((((((.(((((((.....)))))))......(((((((.........(((((((.......))))))))))))))..)))).(((((((...))))).)).......))))).... ( -27.10) >DroYak_CAF1 13644 120 + 1 UUACAAAUCACUAUAUGAUGGCCGUAUAGCCCUACCAUAAAUCUUUCAACAUGUUUAGCCAACAACUAAACAAUUUAUGACGAUUGUCUCCGCCAUGCGGGCGAAUCUCUCUGUGAGUGU (((((((((.(((((((.....)))))))......(((((((.........(((((((.......))))))))))))))..)))).....((((.....))))........))))).... ( -24.40) >consensus UUACAAAUCACUAUAUGAUGGCCGUAUAGCCCUACCAUAAAUCUUUCAACAUGUUUAGCCAACAACUAAACAAUUUAUGACGAUUGUCUCCGCCAUGCGGGCGAAUCUUUCUGUAAGUAU (((((((((.(((((((.....)))))))......(((((((.........(((((((.......))))))))))))))..)))).(((((((...))))).)).......))))).... (-25.06 = -24.74 + -0.32)



| Location | 7,476,397 – 7,476,517 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.33 |
| Mean single sequence MFE | -30.16 |
| Consensus MFE | -29.20 |
| Energy contribution | -29.08 |
| Covariance contribution | -0.12 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.23 |
| Structure conservation index | 0.97 |
| SVM decision value | 0.29 |
| SVM RNA-class probability | 0.671979 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7476397 120 - 22224390 AUACUUACAAAAAGAUUCGCCCGCAUGGCGGAGACAAUCGUCAUAAAUUGUUUAGUUGUUGGCUAAACAUGUUGAAAGAUUUAUGGUAGGGCUAUACGGCCAUCAUAUAGUGAUUUGUAA .....((((((....((((((.....)))))).((((((.(((.....((((((((.....))))))))...)))..))))((((((..((((....))))))))))..))..)))))). ( -29.60) >DroSec_CAF1 9611 120 - 1 AUACUUACAGAAAGAUUCGCCCGCAUGGCGGAGACAAUCGUCAUAAAUUGUUUAGUUGUUGGCUAAACAUGUUGAAAGAUUUAUGGUAGGGCUAUACGGCCAUCAUAUAGUGAUUUGUAA .....((((((....((((((.....)))))).((((((.(((.....((((((((.....))))))))...)))..))))((((((..((((....))))))))))..))..)))))). ( -29.70) >DroSim_CAF1 9161 120 - 1 AUACUUACAGAAAGAUUCGCCCGCAUGGCGGAGACAAUCGUCAUAAAUUGUUUAGUUGUUGGCUAAACAUGUUGAAAGAUUUAUGGUAGGGCUAUACGGCCAUCAUAUAGUGAUUUGUAA .....((((((....((((((.....)))))).((((((.(((.....((((((((.....))))))))...)))..))))((((((..((((....))))))))))..))..)))))). ( -29.70) >DroEre_CAF1 10342 120 - 1 AUACUUACAGAAAGAUUCGCCCGCAUGGCGGGGACAAUCGUCAUAAAUUGUUUAGUUGUUGGCUAAACAUGUUGAAAGAUUUAUGGUAGGGCUAUACGGCCAUCAUAUAGUGAUUUGUAA .....((((((..(((((.(((((...)))))))..))).((((....((((((((.....))))))))............((((((..((((....))))))))))..)))))))))). ( -31.50) >DroYak_CAF1 13644 120 - 1 ACACUCACAGAGAGAUUCGCCCGCAUGGCGGAGACAAUCGUCAUAAAUUGUUUAGUUGUUGGCUAAACAUGUUGAAAGAUUUAUGGUAGGGCUAUACGGCCAUCAUAUAGUGAUUUGUAA (((.((((.((....((((((.....)))))).....)).(((.....((((((((.....))))))))...)))......((((((..((((....))))))))))..))))..))).. ( -30.30) >consensus AUACUUACAGAAAGAUUCGCCCGCAUGGCGGAGACAAUCGUCAUAAAUUGUUUAGUUGUUGGCUAAACAUGUUGAAAGAUUUAUGGUAGGGCUAUACGGCCAUCAUAUAGUGAUUUGUAA ....(((((((....((((((.....)))))).((((((.(((.....((((((((.....))))))))...)))..))))((((((..((((....))))))))))..))..))))))) (-29.20 = -29.08 + -0.12)

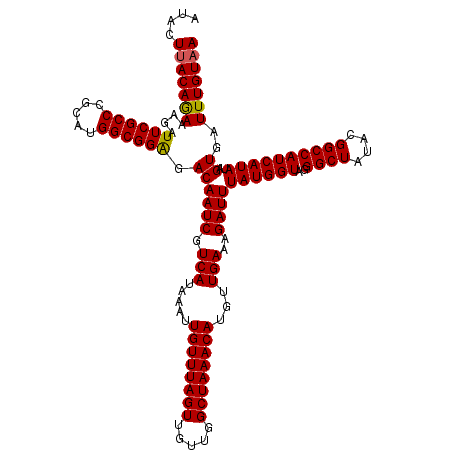

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:43:17 2006