
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 7,005,981 – 7,006,159 |
| Length | 178 |
| Max. P | 0.999655 |

| Location | 7,005,981 – 7,006,095 |
|---|---|
| Length | 114 |
| Sequences | 4 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 97.65 |
| Mean single sequence MFE | -34.53 |
| Consensus MFE | -32.70 |
| Energy contribution | -33.20 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.63 |
| Structure conservation index | 0.95 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.590248 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7005981 114 + 22224390 UUUGUGUGUGCCCGGAUGUGGUCGUUAGAUUAAUGCGCUUUGGCAACUUGCCAGUACAUAAAAUCUAUGUAUGAGCACCGAAAUACGAGUACGUGUAUGUGUAUGCGCGAGUGU (((((((((((.((.(((((.((((........(((....((((.....))))(((((((.....)))))))..))).......)))).)))))...)).)))))))))))... ( -34.66) >DroSec_CAF1 7233 114 + 1 UUUGUGUGUGCCCGGAUGUGGUCGUUAGAUUAAUGCGCUUUGGCAACUUGCCAGUACAUAAAAUCUAUGUAUGAGCACCGAAAUACGAGUACGUGUAUGUGUAUGCGCGAGUGU (((((((((((.((.(((((.((((........(((....((((.....))))(((((((.....)))))))..))).......)))).)))))...)).)))))))))))... ( -34.66) >DroEre_CAF1 13029 110 + 1 UUUGUGUGUGCCCGGAUGUGGUCGUUAGAUUAAUGCGCUUUGGCAACUUGCCAGUACAUAAAAUCUAUGUAUGAGCACCGAAAUACGAGUACGUG----UGUAUGCGCGAGUGU (((((((((((.(((...(((((....)))))....((((((((.....))))(((((((.....))))))))))).)))..(((((....))))----))))))))))))... ( -34.40) >DroYak_CAF1 21423 110 + 1 UUUGUGUGUGCCCGGAUGUGGUCGUUAGAUUAAUGCGCUUUGGCAACUUGCCAGUACAUAAAAUCUAUGUAUGAGCACCGAAAUACGAGUACGUG----UGUAUGCGCGAGUGU (((((((((((.(((...(((((....)))))....((((((((.....))))(((((((.....))))))))))).)))..(((((....))))----))))))))))))... ( -34.40) >consensus UUUGUGUGUGCCCGGAUGUGGUCGUUAGAUUAAUGCGCUUUGGCAACUUGCCAGUACAUAAAAUCUAUGUAUGAGCACCGAAAUACGAGUACGUG____UGUAUGCGCGAGUGU (((((((((((.(((...(((((....)))))....((((((((.....))))(((((((.....))))))))))).)))..(((((....)))))....)))))))))))... (-32.70 = -33.20 + 0.50)

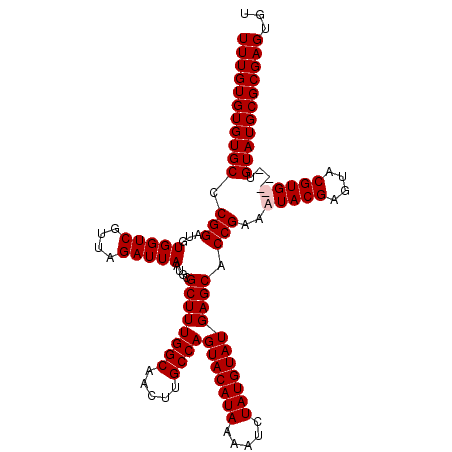

| Location | 7,006,021 – 7,006,135 |
|---|---|
| Length | 114 |
| Sequences | 4 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 95.81 |
| Mean single sequence MFE | -36.22 |
| Consensus MFE | -33.33 |
| Energy contribution | -34.82 |
| Covariance contribution | 1.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -4.15 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.57 |
| SVM RNA-class probability | 0.964814 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7006021 114 + 22224390 UGGCAACUUGCCAGUACAUAAAAUCUAUGUAUGAGCACCGAAAUACGAGUACGUGUAUGUGUAUGCGCGAGUGUCCUUACAUACACAUACACGUAUGUAUCCCUAUCGCCAUAC ((((....(((..(((((((.....)))))))..)))..(..((((..(((((((((((((((((...(((....))).)))))))))))))))))))))..)....))))... ( -41.50) >DroSec_CAF1 7273 110 + 1 UGGCAACUUGCCAGUACAUAAAAUCUAUGUAUGAGCACCGAAAUACGAGUACGUGUAUGUGUAUGCGCGAGUGUCCUUACAUACA----CACGUAUGUAUCCCUAUCGCCAUAC ((((....(((..(((((((.....)))))))..)))..(..((((..((((((((.((((((.((....)).....)))))).)----)))))))))))..)....))))... ( -33.80) >DroEre_CAF1 13069 106 + 1 UGGCAACUUGCCAGUACAUAAAAUCUAUGUAUGAGCACCGAAAUACGAGUACGUG----UGUAUGCGCGAGUGUCCUUACAUACA----CACGUAUGUAUCCCUAUCGCCAUAC ((((....(((..(((((((.....)))))))..)))..(..((((..(((((((----((((((...(((....))).))))))----)))))))))))..)....))))... ( -34.80) >DroYak_CAF1 21463 106 + 1 UGGCAACUUGCCAGUACAUAAAAUCUAUGUAUGAGCACCGAAAUACGAGUACGUG----UGUAUGCGCGAGUGUCCUUACAUACA----CACGUAUGUAUCCCUAUCGCCAUAC ((((....(((..(((((((.....)))))))..)))..(..((((..(((((((----((((((...(((....))).))))))----)))))))))))..)....))))... ( -34.80) >consensus UGGCAACUUGCCAGUACAUAAAAUCUAUGUAUGAGCACCGAAAUACGAGUACGUG____UGUAUGCGCGAGUGUCCUUACAUACA____CACGUAUGUAUCCCUAUCGCCAUAC ((((....(((..(((((((.....)))))))..)))..(..((((..(((((((((((((((((...(((....))).)))))))))))))))))))))..)....))))... (-33.33 = -34.82 + 1.50)


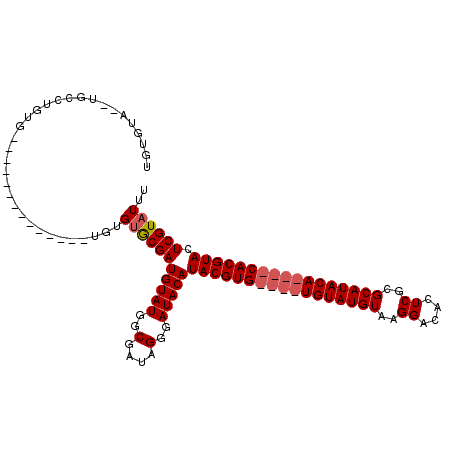
| Location | 7,006,021 – 7,006,135 |
|---|---|
| Length | 114 |
| Sequences | 4 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 95.81 |
| Mean single sequence MFE | -42.72 |
| Consensus MFE | -41.12 |
| Energy contribution | -42.62 |
| Covariance contribution | 1.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -4.86 |
| Structure conservation index | 0.96 |
| SVM decision value | 1.40 |
| SVM RNA-class probability | 0.949938 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7006021 114 - 22224390 GUAUGGCGAUAGGGAUACAUACGUGUAUGUGUAUGUAAGGACACUCGCGCAUACACAUACACGUACUCGUAUUUCGGUGCUCAUACAUAGAUUUUAUGUACUGGCAAGUUGCCA ...(((((((.(..((((.(((((((((((((((((..((....))..)))))))))))))))))...))))..)..((((..(((((((...)))))))..)))).))))))) ( -49.30) >DroSec_CAF1 7273 110 - 1 GUAUGGCGAUAGGGAUACAUACGUG----UGUAUGUAAGGACACUCGCGCAUACACAUACACGUACUCGUAUUUCGGUGCUCAUACAUAGAUUUUAUGUACUGGCAAGUUGCCA ...(((((((.(..((((.((((((----(((((((..((....))..))))))....)))))))...))))..)..((((..(((((((...)))))))..)))).))))))) ( -36.40) >DroEre_CAF1 13069 106 - 1 GUAUGGCGAUAGGGAUACAUACGUG----UGUAUGUAAGGACACUCGCGCAUACA----CACGUACUCGUAUUUCGGUGCUCAUACAUAGAUUUUAUGUACUGGCAAGUUGCCA ...(((((((.(..((((.((((((----(((((((..((....))..)))))))----))))))...))))..)..((((..(((((((...)))))))..)))).))))))) ( -42.60) >DroYak_CAF1 21463 106 - 1 GUAUGGCGAUAGGGAUACAUACGUG----UGUAUGUAAGGACACUCGCGCAUACA----CACGUACUCGUAUUUCGGUGCUCAUACAUAGAUUUUAUGUACUGGCAAGUUGCCA ...(((((((.(..((((.((((((----(((((((..((....))..)))))))----))))))...))))..)..((((..(((((((...)))))))..)))).))))))) ( -42.60) >consensus GUAUGGCGAUAGGGAUACAUACGUG____UGUAUGUAAGGACACUCGCGCAUACA____CACGUACUCGUAUUUCGGUGCUCAUACAUAGAUUUUAUGUACUGGCAAGUUGCCA ...(((((((.(..((((.(((((((((((((((((..((....))..)))))))))))))))))...))))..)..((((..(((((((...)))))))..)))).))))))) (-41.12 = -42.62 + 1.50)



| Location | 7,006,061 – 7,006,159 |
|---|---|
| Length | 98 |
| Sequences | 4 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 80.81 |
| Mean single sequence MFE | -28.44 |
| Consensus MFE | -23.17 |
| Energy contribution | -23.29 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.10 |
| Mean z-score | -4.46 |
| Structure conservation index | 0.81 |
| SVM decision value | 3.84 |
| SVM RNA-class probability | 0.999655 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7006061 98 + 22224390 AAAUACGAGUACGUGUAUGUGUAUGCGCGAGUGUCCUUACAUACACAUACACGUAUGUAUCCCUAUCGCCAUACAUCGCACACA--------------CACAGGCA--CACACA ..((((..(((((((((((((((((...(((....))).))))))))))))))))))))).......(((..............--------------....))).--...... ( -28.97) >DroSec_CAF1 7313 94 + 1 AAAUACGAGUACGUGUAUGUGUAUGCGCGAGUGUCCUUACAUACA----CACGUAUGUAUCCCUAUCGCCAUACAUCGCACACA--------------CACAGGCA--CACACA ............((((.(((((.((.((((((((...(((((((.----...)))))))...........)))).)))).)).)--------------)))).)))--)..... ( -27.14) >DroEre_CAF1 13109 82 + 1 AAAUACGAGUACGUG----UGUAUGCGCGAGUGUCCUUACAUACA----CACGUAUGUAUCCCUAUCGCCAUACAUCGCACAUA------------------------UACACA ............(((----((((((.((((((((...(((((((.----...)))))))...........)))).)))).))))------------------------))))). ( -26.44) >DroYak_CAF1 21503 106 + 1 AAAUACGAGUACGUG----UGUAUGCGCGAGUGUCCUUACAUACA----CACGUAUGUAUCCCUAUCGCCAUACAUCGUGCACAGACACUCACACACACACAUAAACUUACACA ......((((..(((----(((.((.(.(((((((..(((((((.----...)))))))........(((.......).))...))))))).).)).))))))..))))..... ( -31.20) >consensus AAAUACGAGUACGUG____UGUAUGCGCGAGUGUCCUUACAUACA____CACGUAUGUAUCCCUAUCGCCAUACAUCGCACACA______________CACAGGCA__CACACA ............((((...((((((.((((.......(((((((........)))))))......))))))))))..))))................................. (-23.17 = -23.29 + 0.12)


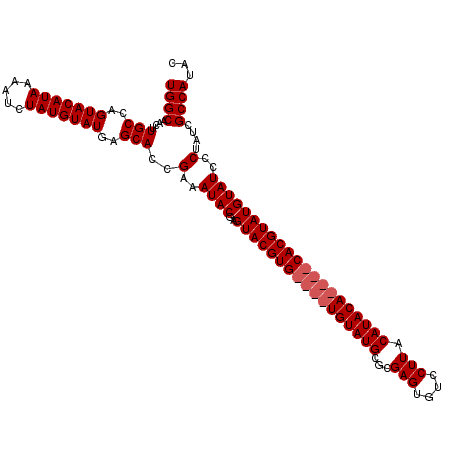
| Location | 7,006,061 – 7,006,159 |
|---|---|
| Length | 98 |
| Sequences | 4 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 80.81 |
| Mean single sequence MFE | -37.45 |
| Consensus MFE | -24.89 |
| Energy contribution | -26.45 |
| Covariance contribution | 1.56 |
| Combinations/Pair | 1.03 |
| Mean z-score | -4.36 |
| Structure conservation index | 0.66 |
| SVM decision value | 3.58 |
| SVM RNA-class probability | 0.999416 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 7006061 98 - 22224390 UGUGUG--UGCCUGUG--------------UGUGUGCGAUGUAUGGCGAUAGGGAUACAUACGUGUAUGUGUAUGUAAGGACACUCGCGCAUACACAUACACGUACUCGUAUUU .(((((--(.(((((.--------------(((((((...)))).)))))))).))))))((((((((((((((((..((....))..)))))))))))))))).......... ( -38.70) >DroSec_CAF1 7313 94 - 1 UGUGUG--UGCCUGUG--------------UGUGUGCGAUGUAUGGCGAUAGGGAUACAUACGUG----UGUAUGUAAGGACACUCGCGCAUACACAUACACGUACUCGUAUUU .(((((--((..((((--------------(((((((((.(((((............)))))(((----(.(......).)))))))))))))))))..)))))))........ ( -35.60) >DroEre_CAF1 13109 82 - 1 UGUGUA------------------------UAUGUGCGAUGUAUGGCGAUAGGGAUACAUACGUG----UGUAUGUAAGGACACUCGCGCAUACA----CACGUACUCGUAUUU ......------------------------...(((((((((((..(....)..)))))((((((----(((((((..((....))..)))))))----)))))).)))))).. ( -27.10) >DroYak_CAF1 21503 106 - 1 UGUGUAAGUUUAUGUGUGUGUGUGAGUGUCUGUGCACGAUGUAUGGCGAUAGGGAUACAUACGUG----UGUAUGUAAGGACACUCGCGCAUACA----CACGUACUCGUAUUU .(((..(((...(((((((((((((((((((((((((.(((((((............))))))))----)))))....)))))))))))))))))----))...)))..))).. ( -48.40) >consensus UGUGUA__UGCCUGUG______________UGUGUGCGAUGUAUGGCGAUAGGGAUACAUACGUG____UGUAUGUAAGGACACUCGCGCAUACA____CACGUACUCGUAUUU .................................(((((((((((..(....)..)))))(((((((((((((((((..((....))..))))))))))))))))).)))))).. (-24.89 = -26.45 + 1.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:39:27 2006