
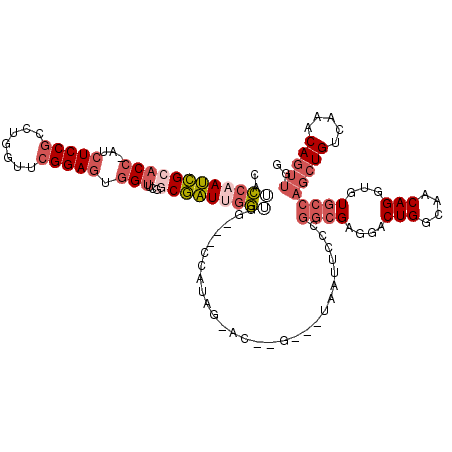
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 6,886,696 – 6,886,809 |
| Length | 113 |
| Max. P | 0.940287 |

| Location | 6,886,696 – 6,886,809 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 74.04 |
| Mean single sequence MFE | -41.45 |
| Consensus MFE | -27.79 |
| Energy contribution | -29.83 |
| Covariance contribution | 2.04 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.39 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.89 |
| SVM RNA-class probability | 0.875916 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6886696 113 + 22224390 CAACCAAUCGCACC-AUCUCCGCCUGGUUCGGAGUGGUUCGGCGAUUGGUGUAGCCCAUCG-AU--G---UAAUACCCGGCGAGGACUGGCAACAGGUGUGCCAGCUGUCAAACAGUUGG ..((((((((((((-(.(((((.......)))))))))...))))))))).....((.(((-.(--(---.......)).))))).(((....))).....(((((((.....))))))) ( -43.10) >DroVir_CAF1 31455 103 + 1 UCGGCAACUGGACUGGGCUCCGCUUGGUACGGAGUUGUC--GCAGUUGCCC----UCACAUCACGUGCGUCGACUGCUGGC---UACUGGCAACAGGU--------UGUCAGUCAGUCGA ..(((((((((((...((((((.......)))))).)))--.))))))))(----.(((.....))).)((((((((((((---..(((....)))..--------.))))).))))))) ( -51.70) >DroSec_CAF1 11020 113 + 1 CAUCCAAUCGCACC-AUCUCCCUCUGGUUCGGAGUGGUUCGGCGAUCGGUGGACCCCAUAG-AU--G---UAAUCCCCGGCGAGGACUGGCAACAGGUGUGCCAGCUGUCAAACAGUUGG ..(((..(((.(((-(.((((.........)))))))).)))((.((((.(((...((...-.)--)---...))))))))).)))(((....))).....(((((((.....))))))) ( -38.10) >DroSim_CAF1 11730 113 + 1 CAUCCAAUCGCACC-AUCUCCGCCUGGUUCGGAGUGGUUCGGCGAUCGGUGGACCCCAUAG-AC--G---UAAUCCCCGGCGAGGACUGGCAACAGGUGUGCCAGCUGUCAAACAGUUGG ........((((((-.((..((((.((..((((....))))(((.((.((((...)))).)-))--)---)....)).))))..)).((....))))))))(((((((.....))))))) ( -40.40) >DroEre_CAF1 10762 99 + 1 CAUCCAAUCGCUCC-AUCUCCGCCUGGUUCGGAAUGGUUUGCCGAUUCGUGG--------------------AAUCCCGGCGAGGACUGGCAACAGGUGUGCCAGCUGUCAAACAGUUGG ........(((.((-.((..((((.((..((((.(((....))).))))...--------------------...)).))))..)).((....)))).)))(((((((.....))))))) ( -34.50) >DroYak_CAF1 12817 108 + 1 CAUCCAAUCGCACCAAUCUCCGCCUGGUUCGGAGUGGUUUGGCGAUUGGACG------UAA-AC--C---UAAUUCCCGGCGAGGACUGGCAACAGGUGUGCCAGCUGUCAAACAGUUGG ..(((((((((((((..(((((.......)))))))))...)))))))))..------...-..--.---......((((((....(((....)))...))))(((((.....))))))) ( -40.90) >consensus CAUCCAAUCGCACC_AUCUCCGCCUGGUUCGGAGUGGUUCGGCGAUUGGUGG___CCAUAG_AC__G___UAAUUCCCGGCGAGGACUGGCAACAGGUGUGCCAGCUGUCAAACAGUUGG ..((((((((((((...(((((.......))))).)))...)))))))))............................((((....(((....)))...))))(((((.....))))).. (-27.79 = -29.83 + 2.04)


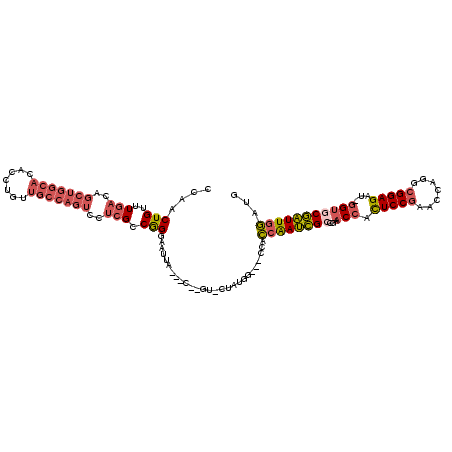
| Location | 6,886,696 – 6,886,809 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 74.04 |
| Mean single sequence MFE | -41.15 |
| Consensus MFE | -26.73 |
| Energy contribution | -27.90 |
| Covariance contribution | 1.17 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.68 |
| Structure conservation index | 0.65 |
| SVM decision value | 1.31 |
| SVM RNA-class probability | 0.940287 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6886696 113 - 22224390 CCAACUGUUUGACAGCUGGCACACCUGUUGCCAGUCCUCGCCGGGUAUUA---C--AU-CGAUGGGCUACACCAAUCGCCGAACCACUCCGAACCAGGCGGAGAU-GGUGCGAUUGGUUG ..........((..((((((((....).)))))))..))(((.(......---.--..-...).)))...(((((((((...(((((((((.......))))).)-)))))))))))).. ( -41.20) >DroVir_CAF1 31455 103 - 1 UCGACUGACUGACA--------ACCUGUUGCCAGUA---GCCAGCAGUCGACGCACGUGAUGUGA----GGGCAACUGC--GACAACUCCGUACCAAGCGGAGCCCAGUCCAGUUGCCGA (((((((.(((.(.--------..(((....)))..---).))))))))))((((.....)))).----.((((((((.--(((..((((((.....))))))....))))))))))).. ( -42.90) >DroSec_CAF1 11020 113 - 1 CCAACUGUUUGACAGCUGGCACACCUGUUGCCAGUCCUCGCCGGGGAUUA---C--AU-CUAUGGGGUCCACCGAUCGCCGAACCACUCCGAACCAGAGGGAGAU-GGUGCGAUUGGAUG ....(((..(((..((((((((....).)))))))..))).)))((((..---(--(.-...))..)))).((((((((...((((((((.........)))).)-)))))))))))... ( -42.90) >DroSim_CAF1 11730 113 - 1 CCAACUGUUUGACAGCUGGCACACCUGUUGCCAGUCCUCGCCGGGGAUUA---C--GU-CUAUGGGGUCCACCGAUCGCCGAACCACUCCGAACCAGGCGGAGAU-GGUGCGAUUGGAUG ....(((..(((..((((((((....).)))))))..))).)))((((..---(--(.-...))..)))).((((((((...(((((((((.......))))).)-)))))))))))... ( -44.80) >DroEre_CAF1 10762 99 - 1 CCAACUGUUUGACAGCUGGCACACCUGUUGCCAGUCCUCGCCGGGAUU--------------------CCACGAAUCGGCAAACCAUUCCGAACCAGGCGGAGAU-GGAGCGAUUGGAUG ((((.((((.((..((((((((....).)))))))..))(((..((((--------------------(...))))))))...((((((((.......)))).))-)))))).))))... ( -33.10) >DroYak_CAF1 12817 108 - 1 CCAACUGUUUGACAGCUGGCACACCUGUUGCCAGUCCUCGCCGGGAAUUA---G--GU-UUA------CGUCCAAUCGCCAAACCACUCCGAACCAGGCGGAGAUUGGUGCGAUUGGAUG ..........((..((((((((....).)))))))..))(((........---)--))-...------(((((((((((...(((((((((.......)))))..))))))))))))))) ( -42.00) >consensus CCAACUGUUUGACAGCUGGCACACCUGUUGCCAGUCCUCGCCGGGAAUUA___C__GU_CUAUGG___CCACCAAUCGCCGAACCACUCCGAACCAGGCGGAGAU_GGUGCGAUUGGAUG ....(((..(((..(((((((.......)))))))..))).)))...........................((((((((...(((.(((((.......)))))...)))))))))))... (-26.73 = -27.90 + 1.17)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:37:51 2006