
| Sequence ID | X_DroMel_CAF1 |
|---|---|
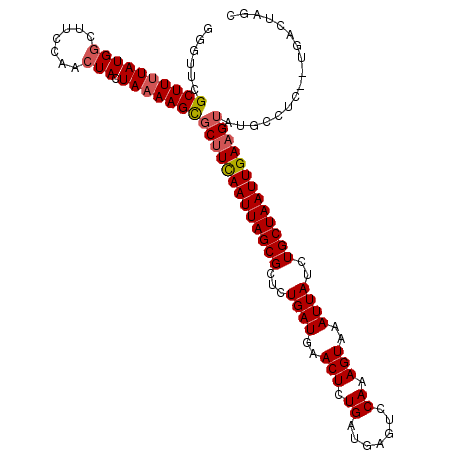
| Location | 6,735,281 – 6,735,392 |
| Length | 111 |
| Max. P | 0.946995 |

| Location | 6,735,281 – 6,735,392 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 88.39 |
| Mean single sequence MFE | -29.02 |
| Consensus MFE | -24.13 |
| Energy contribution | -24.52 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.16 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.60 |
| SVM RNA-class probability | 0.795433 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6735281 111 + 22224390 GGGUUCGCUUUUAUGGCUUCCAACUACUAAAAGCGCUUCAAUUAGCGCUCUGAUGAACUCUGAUGAGUCCAAAGUAAAUUAUCUGCUAAUUGAAGUAUGCCUC--UGACUAGC ((((..((((((((((.......))).)))))))(((((((((((((....(((((((((....))))..........))))))))))))))))))..)))).--........ ( -30.30) >DroSec_CAF1 21500 95 + 1 GGGUUCGCUUUUAUGGCUUCCAACUACUAAAAGCGCUUCAAUUAGCGCUCUGAUGAACUCUGAUGAGUCCAAAGUAAAUUAUCUGCUAAUUGAUG------------------ .....(((((((((((.......))).))))))))..((((((((((....(((((((((....))))..........)))))))))))))))..------------------ ( -22.80) >DroEre_CAF1 26022 111 + 1 GGGUUCGCUUUUAUGGCUUCCAACUACUAAAAGCGCUUCAAUUAGCGCUCUGAUGAACUCUGAUGAGUCCAAAGUAAAUUAUCUGCUAAUUGAAGUAUGCCUC--UGACUAGC ((((..((((((((((.......))).)))))))(((((((((((((....(((((((((....))))..........))))))))))))))))))..)))).--........ ( -30.30) >DroYak_CAF1 31349 113 + 1 GGGUUCGCUUUUAUGGCUUCCAACUACUAAAAGCGCUUCAAUUAGCGCUCUGAUGAACUCUGAUGAGUCCAAAGUAAAUUAUCUGCUAAUUGAAGUAUGCCUCUAUGACUAGC ((((..((((((((((.......))).)))))))(((((((((((((....(((((((((....))))..........))))))))))))))))))..))))........... ( -30.30) >DroAna_CAF1 24765 111 + 1 GGUUUCGCUUUUAUGGCCUC-GGCUACUAAAAGCGCUUUAAUUAGCGCUCUGAUGAACUCUGAUGAGCCCAAAGUAAAUUAUCUGCUAAUUGAAGUAUGCCUU-ACUCCUAAC (((...(((((((((((...-.)))).)))))))((((((((((((((((....(....)....))))....((........))))))))))))))..)))..-......... ( -33.00) >DroPer_CAF1 77866 110 + 1 GGCUUCGCUU-UAUGACUGCCAACUACUAAAAGUGCUUCAAUUAGCGCUCUGAUGAACUCUGAUGAGCCCAAAGUAAAUUAUCUGCUAAUUGAAGUAUGCCUC--CUGACAGC (((.(((...-..)))..)))...........((((((((((((((((((....(....)....))))....((........)))))))))))))))).....--........ ( -27.40) >consensus GGGUUCGCUUUUAUGGCUUCCAACUACUAAAAGCGCUUCAAUUAGCGCUCUGAUGAACUCUGAUGAGUCCAAAGUAAAUUAUCUGCUAAUUGAAGUAUGCCUC__UGACUAGC ......((((((((((.......))).)))))))(((((((((((((...((((..(((.((.......)).)))..))))..)))))))))))))................. (-24.13 = -24.52 + 0.39)


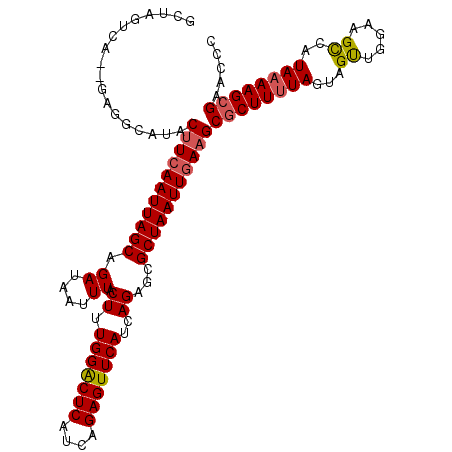
| Location | 6,735,281 – 6,735,392 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 88.39 |
| Mean single sequence MFE | -29.75 |
| Consensus MFE | -26.45 |
| Energy contribution | -26.62 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.37 |
| SVM RNA-class probability | 0.946995 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6735281 111 - 22224390 GCUAGUCA--GAGGCAUACUUCAAUUAGCAGAUAAUUUACUUUGGACUCAUCAGAGUUCAUCAGAGCGCUAAUUGAAGCGCUUUUAGUAGUUGGAAGCCAUAAAAGCGAACCC (((.....--..)))...(((((((((((.((....)).((.(((((((....)))))))..))...)))))))))))((((((((...((.....))..))))))))..... ( -31.40) >DroSec_CAF1 21500 95 - 1 ------------------CAUCAAUUAGCAGAUAAUUUACUUUGGACUCAUCAGAGUUCAUCAGAGCGCUAAUUGAAGCGCUUUUAGUAGUUGGAAGCCAUAAAAGCGAACCC ------------------..(((((((((.((....)).((.(((((((....)))))))..))...)))))))))..((((((((...((.....))..))))))))..... ( -26.30) >DroEre_CAF1 26022 111 - 1 GCUAGUCA--GAGGCAUACUUCAAUUAGCAGAUAAUUUACUUUGGACUCAUCAGAGUUCAUCAGAGCGCUAAUUGAAGCGCUUUUAGUAGUUGGAAGCCAUAAAAGCGAACCC (((.....--..)))...(((((((((((.((....)).((.(((((((....)))))))..))...)))))))))))((((((((...((.....))..))))))))..... ( -31.40) >DroYak_CAF1 31349 113 - 1 GCUAGUCAUAGAGGCAUACUUCAAUUAGCAGAUAAUUUACUUUGGACUCAUCAGAGUUCAUCAGAGCGCUAAUUGAAGCGCUUUUAGUAGUUGGAAGCCAUAAAAGCGAACCC ....(((.....)))...(((((((((((.((....)).((.(((((((....)))))))..))...)))))))))))((((((((...((.....))..))))))))..... ( -31.40) >DroAna_CAF1 24765 111 - 1 GUUAGGAGU-AAGGCAUACUUCAAUUAGCAGAUAAUUUACUUUGGGCUCAUCAGAGUUCAUCAGAGCGCUAAUUAAAGCGCUUUUAGUAGCC-GAGGCCAUAAAAGCGAAACC ....(((((-(.....))))))(((((((.((....)).((.(((((((....)))))))..))...)))))))....((((((((...((.-...))..))))))))..... ( -29.40) >DroPer_CAF1 77866 110 - 1 GCUGUCAG--GAGGCAUACUUCAAUUAGCAGAUAAUUUACUUUGGGCUCAUCAGAGUUCAUCAGAGCGCUAAUUGAAGCACUUUUAGUAGUUGGCAGUCAUA-AAGCGAAGCC ((((..((--(((.....(((((((((((.((....)).((.(((((((....)))))))..))...)))))))))))..)))))..)))).(((.((....-..))...))) ( -28.60) >consensus GCUAGUCA__GAGGCAUACUUCAAUUAGCAGAUAAUUUACUUUGGACUCAUCAGAGUUCAUCAGAGCGCUAAUUGAAGCGCUUUUAGUAGUUGGAAGCCAUAAAAGCGAACCC ..................(((((((((((.((....)).((.(((((((....)))))))..))...)))))))))))((((((((...((.....))..))))))))..... (-26.45 = -26.62 + 0.17)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:36:57 2006