
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 6,691,758 – 6,691,878 |
| Length | 120 |
| Max. P | 0.802982 |

| Location | 6,691,758 – 6,691,878 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.89 |
| Mean single sequence MFE | -50.33 |
| Consensus MFE | -25.64 |
| Energy contribution | -26.73 |
| Covariance contribution | 1.09 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.02 |
| Structure conservation index | 0.51 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.628874 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
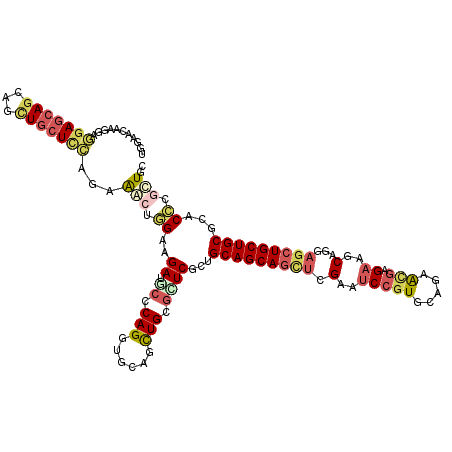
>X_DroMel_CAF1 6691758 120 + 22224390 UGGAACCGCGAGGAGCAGCAGCUCCUCCAGAAACUGGACGACUCACAGGUGCAACUGCGCUCGCUGCAGCAAUUGGAAUCCGUACAGGGCGAGAAGCAGGAGCUGCUGCGCACCCGUUGC .((..((....)).((((((((((((((((...)))))...........(((..((.(((((((....))....((...)).....)))))))..))))))))))))))....))..... ( -48.90) >DroPse_CAF1 2063 120 + 1 UGGAACAAGGAGGAGCAGCUGCUGCUGCAGAGGGCCGAAGAUGCCCAGCUGCAGCUGCGCUCGGUGCAGCAGCUCGAGUCCGUGCAGAACGAGAAGCAGGAGCUGCUGCGCACGCGGUGC .........(((..((((((((.(((.....((((.......))))))).)))))))).))).(((((((((((((..(((((.....))).))..)..))))))))))))......... ( -58.20) >DroGri_CAF1 2074 120 + 1 UGGGUCAAGGAAGAGCAACAGCUGCUCCAGAAACUGGAGGAUUCACAGGCCCAACUGCGUUCGCUGCAGCAGCUCGAAUCCGUGCAACAUGAGAAACAGGAGCUGCUGCGCACACGUUGU ((((((...(((.(((....))).((((((...))))))..)))...))))))((.((((..((.((((((((((...((((((...)))).)).....))))))))))))..)))).)) ( -49.00) >DroMoj_CAF1 2025 120 + 1 UGGAGCAAGGACGAGCAGCUGUUGCUCGAGAAGCUGGAGGAUGCGCAGGCUCAGUUGCGUUCGCUGCAGCAGCAAGAAACGGUGCACCAAGAGAAGCAGGAUCUGCUGCGCACCCGCUGC .(.(((.....(((((((...)))))))....((((.((((.((((((......)))))))).)).))))..........(((((..(....)..((((......))))))))).))).) ( -41.40) >DroAna_CAF1 2258 120 + 1 UGGAAUCGGGAGGAGCAGCAACUGCUCCAGAAACUGGACGAUACCCAGGUGCAGUUGCAUUCCCUGCAGCAGCUGGAGUCCGUUCAGGGCGAGAAACAGGAGCUGCUGCGCACCCGAUGC ....((((((..(.((((((...(((((.....(((((((..((((((.(((...((((.....))))))).)))).)).)))))))(........).))))))))))).).)))))).. ( -53.10) >DroPer_CAF1 2086 120 + 1 UGGAACAAGGAGGAGCAGCUGCUGAUUCAGAGGGCCGAAGAUGCCCAGCUGCAGCUGCGCUCGAUGCAGCAGCUCGAGUCCGUGCAGAACGAGAAGCAGGAGCUGCUGCGCACGCGCUGC ..............((((((.(((...))).((((.......)))))))))).((.((((..(.((((((((((((..(((((.....))).))..)..))))))))))))..)))).)) ( -51.40) >consensus UGGAACAAGGAGGAGCAGCAGCUGCUCCAGAAACUGGAAGAUGCCCAGGUGCAGCUGCGCUCGCUGCAGCAGCUCGAAUCCGUGCAGAACGAGAAGCAGGAGCUGCUGCGCACCCGCUGC ...........(((((((...)))))))...(((.((..((.((.(((......))).))))...(((((((((.(..(((((.....))).))..)...)))))))))...)).))).. (-25.64 = -26.73 + 1.09)

| Location | 6,691,758 – 6,691,878 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.89 |
| Mean single sequence MFE | -49.22 |
| Consensus MFE | -25.05 |
| Energy contribution | -26.38 |
| Covariance contribution | 1.34 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.26 |
| Structure conservation index | 0.51 |
| SVM decision value | 0.62 |
| SVM RNA-class probability | 0.802982 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
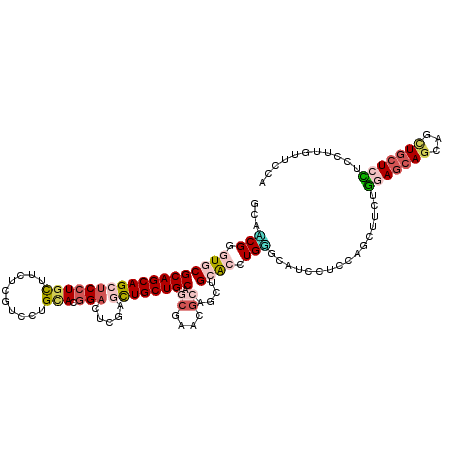
>X_DroMel_CAF1 6691758 120 - 22224390 GCAACGGGUGCGCAGCAGCUCCUGCUUCUCGCCCUGUACGGAUUCCAAUUGCUGCAGCGAGCGCAGUUGCACCUGUGAGUCGUCCAGUUUCUGGAGGAGCUGCUGCUCCUCGCGGUUCCA ((..((((.(.((((((((((((((..(((((((.....))....(((((((.((.....))))))))).....)))))..))((((...)))))))))))))))).)))))..)).... ( -51.90) >DroPse_CAF1 2063 120 - 1 GCACCGCGUGCGCAGCAGCUCCUGCUUCUCGUUCUGCACGGACUCGAGCUGCUGCACCGAGCGCAGCUGCAGCUGGGCAUCUUCGGCCCUCUGCAGCAGCAGCUGCUCCUCCUUGUUCCA ((((...))))((((((((((..(..((.(((.....))))))..))))))))))...(((.((((((((.(((((((.......)))).....))).))))))))..)))......... ( -53.60) >DroGri_CAF1 2074 120 - 1 ACAACGUGUGCGCAGCAGCUCCUGUUUCUCAUGUUGCACGGAUUCGAGCUGCUGCAGCGAACGCAGUUGGGCCUGUGAAUCCUCCAGUUUCUGGAGCAGCUGUUGCUCUUCCUUGACCCA ....(((.(((((((((((((((((..(....)..))).))).....)))))))).))).)))....(((((..(.(((..((((((...)))))).(((....))).))))..).)))) ( -40.70) >DroMoj_CAF1 2025 120 - 1 GCAGCGGGUGCGCAGCAGAUCCUGCUUCUCUUGGUGCACCGUUUCUUGCUGCUGCAGCGAACGCAACUGAGCCUGCGCAUCCUCCAGCUUCUCGAGCAACAGCUGCUCGUCCUUGCUCCA ((((((((((((((((((...))))....((..((....(((((.((((....)))).)))))..))..))..)))))))))....((((...))))....))))).............. ( -45.50) >DroAna_CAF1 2258 120 - 1 GCAUCGGGUGCGCAGCAGCUCCUGUUUCUCGCCCUGAACGGACUCCAGCUGCUGCAGGGAAUGCAACUGCACCUGGGUAUCGUCCAGUUUCUGGAGCAGUUGCUGCUCCUCCCGAUUCCA ..((((((...(((((((((.((((((........)))))).....))))))))).((((..((((((((((....))....(((((...)))))))))))))...)))))))))).... ( -52.40) >DroPer_CAF1 2086 120 - 1 GCAGCGCGUGCGCAGCAGCUCCUGCUUCUCGUUCUGCACGGACUCGAGCUGCUGCAUCGAGCGCAGCUGCAGCUGGGCAUCUUCGGCCCUCUGAAUCAGCAGCUGCUCCUCCUUGUUCCA (((((((((..((((((((((..(..((.(((.....))))))..)))))))))).....)))).(((((((..((((.......)))).)))...)))).))))).............. ( -51.20) >consensus GCAACGGGUGCGCAGCAGCUCCUGCUUCUCGUCCUGCACGGACUCGAGCUGCUGCAGCGAACGCAGCUGCACCUGGGCAUCCUCCAGCUUCUGGAGCAGCAGCUGCUCCUCCUUGUUCCA ...(((.((((((((((((((((((..........))).))).....)))))))).((....))....)))).)))................(((((((...)))))))........... (-25.05 = -26.38 + 1.34)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:36:45 2006