
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 6,566,808 – 6,567,208 |
| Length | 400 |
| Max. P | 0.999637 |

| Location | 6,566,808 – 6,566,928 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.67 |
| Mean single sequence MFE | -44.24 |
| Consensus MFE | -43.32 |
| Energy contribution | -42.84 |
| Covariance contribution | -0.48 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.20 |
| Structure conservation index | 0.98 |
| SVM decision value | 0.44 |
| SVM RNA-class probability | 0.736590 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6566808 120 + 22224390 CGUAUUGGGCAACAGGAGGCACAUGAGACCGCCGCCGAUGAGCAGACCCGACACGAUGAACGUGGGCGAUGGACAGUAGUUGUCCAGCAGCUGGGCAAUCACGAUGUUGCCAAUGAUGCC .(((((.(((((((((.(((..........))).))(.(((...(.(((..((((.....))))(((..(((((((...)))))))...)))))))..))))..)))))))...))))). ( -43.80) >DroSec_CAF1 11770 120 + 1 CGUAUUGGGCAACAGGAGGCACAUGAGACCGCCGCCGAUGAGCAGACCCGACACGAUGAACGUGGGCGACGGGCAGUAGUUGUCCAGCAGCUGGGCAAUCACGAUGUUGCCAAUGAUGCC .(((((.(((((((.........((...(((.((((.(((..((............))..))).)))).))).))((.(((((((((...))))))))).))..)))))))...))))). ( -44.10) >DroSim_CAF1 22296 120 + 1 CGUAUUGGGCAACAGGAGGCACAUGAGACCGCCGCCGAUGAGCAGACCCGACACGAUGAACGUGGGCGACGGGCAGUAGUUGUCCAGCAGCUGGGCAAUCACGAUGUUGCCAAUGAUGCC .(((((.(((((((.........((...(((.((((.(((..((............))..))).)))).))).))((.(((((((((...))))))))).))..)))))))...))))). ( -44.10) >DroEre_CAF1 11396 120 + 1 CGUAUUGGGCAACAGCAGGCACAUGAGACCACCGCCGAUGAGCAGACCCGACACGAUGAACGUGGGCGAUGGACAGUAGUUGUCCAGCAGCUGGGCAAUCACGAUGUUGCCAAUGAUGCC .(((((.(((((((.(............(((.((((.(((..((............))..))).)))).)))...((.(((((((((...))))))))).))).)))))))...))))). ( -42.80) >DroYak_CAF1 21214 120 + 1 CGUAUUGGGCAACAGCAGGCACAUGAGACCACCGCCGAUGAGCAGACCCGACACGAUGAACGUGGGCGAUGGACAGUAGUUGUCCAGCAGCUGGGCAAUUACGAUGUUGCCAAUGAUGCC .(((((.(((((((.(............(((.((((.(((..((............))..))).)))).)))...((((((((((((...))))))))))))).)))))))...))))). ( -46.40) >consensus CGUAUUGGGCAACAGGAGGCACAUGAGACCGCCGCCGAUGAGCAGACCCGACACGAUGAACGUGGGCGAUGGACAGUAGUUGUCCAGCAGCUGGGCAAUCACGAUGUUGCCAAUGAUGCC .(((((.(((((((..............(((.((((.(((..((............))..))).)))).)))...((.(((((((((...))))))))).))..)))))))...))))). (-43.32 = -42.84 + -0.48)



| Location | 6,566,808 – 6,566,928 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.67 |
| Mean single sequence MFE | -43.66 |
| Consensus MFE | -42.76 |
| Energy contribution | -42.48 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.51 |
| Structure conservation index | 0.98 |
| SVM decision value | 1.55 |
| SVM RNA-class probability | 0.963416 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6566808 120 - 22224390 GGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACAACUACUGUCCAUCGCCCACGUUCAUCGUGUCGGGUCUGCUCAUCGGCGGCGGUCUCAUGUGCCUCCUGUUGCCCAAUACG ((((.((..((((.(((.(.(((((((.....(.((((((.....)))))).)(((((((.....))))..(((....)))...)))))))))).))))))))...)).))))....... ( -44.80) >DroSec_CAF1 11770 120 - 1 GGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACAACUACUGCCCGUCGCCCACGUUCAUCGUGUCGGGUCUGCUCAUCGGCGGCGGUCUCAUGUGCCUCCUGUUGCCCAAUACG ((((.((..((((.(((.(((.(((.((((...)))).))).)))...((((((((((((.....))))..(((....)))...))))))))....)))))))...)).))))....... ( -43.40) >DroSim_CAF1 22296 120 - 1 GGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACAACUACUGCCCGUCGCCCACGUUCAUCGUGUCGGGUCUGCUCAUCGGCGGCGGUCUCAUGUGCCUCCUGUUGCCCAAUACG ((((.((..((((.(((.(((.(((.((((...)))).))).)))...((((((((((((.....))))..(((....)))...))))))))....)))))))...)).))))....... ( -43.40) >DroEre_CAF1 11396 120 - 1 GGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACAACUACUGUCCAUCGCCCACGUUCAUCGUGUCGGGUCUGCUCAUCGGCGGUGGUCUCAUGUGCCUGCUGUUGCCCAAUACG ((((.((..((((.(((.(.(((..((.....(.((((((.....)))))).)(((((((.....))))..(((....)))...)))))..))).))))))))...)).))))....... ( -43.40) >DroYak_CAF1 21214 120 - 1 GGCAUCAUUGGCAACAUCGUAAUUGCCCAGCUGCUGGACAACUACUGUCCAUCGCCCACGUUCAUCGUGUCGGGUCUGCUCAUCGGCGGUGGUCUCAUGUGCCUGCUGUUGCCCAAUACG ((((.((..((((.(((.(((.(((.((((...)))).))).))).(.((((((((((((.....))))..(((....)))...)))))))).)..)))))))...)).))))....... ( -43.30) >consensus GGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACAACUACUGUCCAUCGCCCACGUUCAUCGUGUCGGGUCUGCUCAUCGGCGGCGGUCUCAUGUGCCUCCUGUUGCCCAAUACG ((((.((..((((.(((.(((.(((.((((...)))).))).))).(.((((((((((((.....))))..(((....)))...)))))))).)..)))))))...)).))))....... (-42.76 = -42.48 + -0.28)



| Location | 6,566,848 – 6,566,968 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.00 |
| Mean single sequence MFE | -50.52 |
| Consensus MFE | -45.24 |
| Energy contribution | -44.36 |
| Covariance contribution | -0.88 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.21 |
| Structure conservation index | 0.90 |
| SVM decision value | 2.69 |
| SVM RNA-class probability | 0.996387 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6566848 120 - 22224390 CACCGGCGUGGCCAUUUCAAUGGUGGCCGCUCGCUUGGGCGGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACAACUACUGUCCAUCGCCCACGUUCAUCGUGUCGGGUCUGCU ..((((((((((((((.....)))))))))..((.(((((((((((((((....))..)))).)))).....(.((((((.....)))))).)))))).)).......)))))....... ( -52.30) >DroSec_CAF1 11810 120 - 1 CACCGGCGUGGCCAUUUCAAUGGUGGCCGCUCGCUUGGGCGGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACAACUACUGCCCGUCGCCCACGUUCAUCGUGUCGGGUCUGCU ..((((((((((((((.....)))))))))..(((.(((((((((...((....))..))).))))))))).))))).........(((((.....((((.....)))).)))))..... ( -51.90) >DroSim_CAF1 22336 120 - 1 CACCGGCGUGGCCAUUUCGAUGGUGGCCGCUCGCUUGGGCGGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACAACUACUGCCCGUCGCCCACGUUCAUCGUGUCGGGUCUGCU ..((((((((((((((.....)))))))))..(((.(((((((((...((....))..))).))))))))).))))).........(((((.....((((.....)))).)))))..... ( -51.90) >DroEre_CAF1 11436 120 - 1 CACCGGUGUGGCCAUUUCGAUGGUGGCCGCUCGCUUGGGCGGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACAACUACUGUCCAUCGCCCACGUUCAUCGUGUCGGGUCUGCU (((.(((((.((((.....((((((.((((((....)))))))))))))))).))))))))...((((.((.(.((((((.....)))))).))).((((.....))))..))))..... ( -48.60) >DroYak_CAF1 21254 120 - 1 CACCGGUGUGGCCAUUUCGAUGGUCGCCGCUCGAUUGGGCGGCAUCAUUGGCAACAUCGUAAUUGCCCAGCUGCUGGACAACUACUGUCCAUCGCCCACGUUCAUCGUGUCGGGUCUGCU ...((((((.((((.....(((((.(((((((....)))))))))))))))).)))))).....((((.((.(.((((((.....)))))).))).((((.....))))..))))..... ( -47.90) >consensus CACCGGCGUGGCCAUUUCGAUGGUGGCCGCUCGCUUGGGCGGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACAACUACUGUCCAUCGCCCACGUUCAUCGUGUCGGGUCUGCU ..(((((((((.((..((((((((.(((((((....))))))))))))))).......(((.(((.((((...)))).))).))))).))).))))((((.....))))..))....... (-45.24 = -44.36 + -0.88)


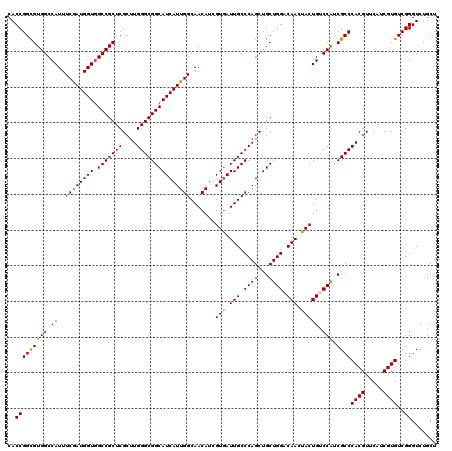
| Location | 6,566,888 – 6,567,008 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.00 |
| Mean single sequence MFE | -50.29 |
| Consensus MFE | -45.92 |
| Energy contribution | -45.20 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.91 |
| Structure conservation index | 0.91 |
| SVM decision value | 2.28 |
| SVM RNA-class probability | 0.991606 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6566888 120 + 22224390 UGUCCAGCAGCUGGGCAAUCACGAUGUUGCCAAUGAUGCCGCCCAAGCGAGCGGCCACCAUUGAAAUGGCCACGCCGGUGGCUCUCAGCUUGGUGGGGAAUACUUCGGUGAUGAGGCAGU (((((((...))))))).........(((((.......((.((((((((((.(((((((((....))(((...))))))))))))).)))))).).))..(((....)))....))))). ( -49.40) >DroSec_CAF1 11850 120 + 1 UGUCCAGCAGCUGGGCAAUCACGAUGUUGCCAAUGAUGCCGCCCAAGCGAGCGGCCACCAUUGAAAUGGCCACGCCGGUGGCCCUCAGCUUGGUGGGGAAUACUUCGGUGAUGAGGCAGU (((((((...))))))).........(((((.......((.((((((((((.(((((((((....))(((...))))))))))))).)))))).).))..(((....)))....))))). ( -52.40) >DroSim_CAF1 22376 120 + 1 UGUCCAGCAGCUGGGCAAUCACGAUGUUGCCAAUGAUGCCGCCCAAGCGAGCGGCCACCAUCGAAAUGGCCACGCCGGUGGCCCUCAGCUUGGUGGGGAAUACUUCGGUGAUGAGGCAGU (((((((...))))))).........(((((.......((.((((((((((.(((((((........(((...))))))))))))).)))))).).))..(((....)))....))))). ( -52.30) >DroEre_CAF1 11476 120 + 1 UGUCCAGCAGCUGGGCAAUCACGAUGUUGCCAAUGAUGCCGCCCAAGCGAGCGGCCACCAUCGAAAUGGCCACACCGGUGGCCCUCAGCUUGGUGGGGAAAACUUCGGUGAUGAGGCAGU (((((((...))))))).........(((((.......((.((((((((((.(((((((.................)))))))))).)))))).).))...((....)).....))))). ( -49.33) >DroYak_CAF1 21294 120 + 1 UGUCCAGCAGCUGGGCAAUUACGAUGUUGCCAAUGAUGCCGCCCAAUCGAGCGGCGACCAUCGAAAUGGCCACACCGGUGGCCCUCAGCUUGGUGGGGAAUACUUCGGUGAUCAGGCAGU (((((((...))))))).........(((((.....((((((.(....).)))))).(((((((...((((((....))))))......))))))).((.(((....))).)).))))). ( -48.00) >consensus UGUCCAGCAGCUGGGCAAUCACGAUGUUGCCAAUGAUGCCGCCCAAGCGAGCGGCCACCAUCGAAAUGGCCACGCCGGUGGCCCUCAGCUUGGUGGGGAAUACUUCGGUGAUGAGGCAGU (((((((...))))))).........(((((......(((((.(....).)))))((.(((((((..((((((....))))))((((......))))......))))))).)).))))). (-45.92 = -45.20 + -0.72)

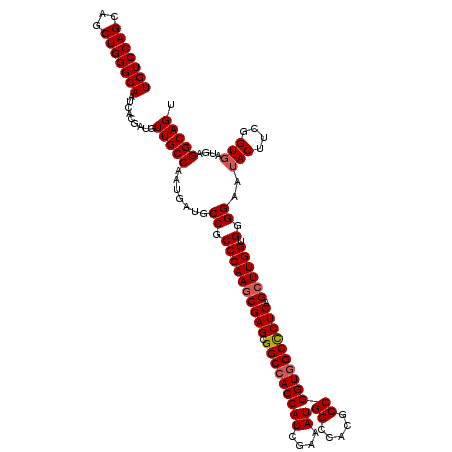
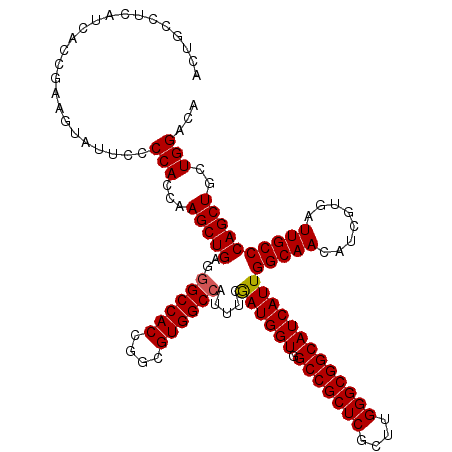
| Location | 6,566,888 – 6,567,008 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.00 |
| Mean single sequence MFE | -46.00 |
| Consensus MFE | -43.14 |
| Energy contribution | -43.10 |
| Covariance contribution | -0.04 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.94 |
| SVM decision value | 1.35 |
| SVM RNA-class probability | 0.944900 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6566888 120 - 22224390 ACUGCCUCAUCACCGAAGUAUUCCCCACCAAGCUGAGAGCCACCGGCGUGGCCAUUUCAAUGGUGGCCGCUCGCUUGGGCGGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACA ..(((.((......)).)))....(((...(((((...(((.(.((((((((((((.....)))))))))..))).))))((((((((((....))..)))).)))))))))..)))... ( -45.30) >DroSec_CAF1 11850 120 - 1 ACUGCCUCAUCACCGAAGUAUUCCCCACCAAGCUGAGGGCCACCGGCGUGGCCAUUUCAAUGGUGGCCGCUCGCUUGGGCGGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACA ............(((.(((........((((((.(((((((((((...(((.....))).)))))))).)))))))))(.((((((((((....))..)))).))))).)))..)))... ( -47.00) >DroSim_CAF1 22376 120 - 1 ACUGCCUCAUCACCGAAGUAUUCCCCACCAAGCUGAGGGCCACCGGCGUGGCCAUUUCGAUGGUGGCCGCUCGCUUGGGCGGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACA ............(((.(((........((((((.(((((((((((.((.........)).)))))))).)))))))))(.((((((((((....))..)))).))))).)))..)))... ( -47.10) >DroEre_CAF1 11476 120 - 1 ACUGCCUCAUCACCGAAGUUUUCCCCACCAAGCUGAGGGCCACCGGUGUGGCCAUUUCGAUGGUGGCCGCUCGCUUGGGCGGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACA ............(((.((((.......((((((.(((((((((((...(((.....))).)))))))).)))))))))(.((((((((((....))..)))).)))))))))..)))... ( -46.30) >DroYak_CAF1 21294 120 - 1 ACUGCCUGAUCACCGAAGUAUUCCCCACCAAGCUGAGGGCCACCGGUGUGGCCAUUUCGAUGGUCGCCGCUCGAUUGGGCGGCAUCAUUGGCAACAUCGUAAUUGCCCAGCUGCUGGACA ........................(((...(((((..((((((....)))))).....((((((.(((((((....)))))))))))))(((((........))))))))))..)))... ( -44.30) >consensus ACUGCCUCAUCACCGAAGUAUUCCCCACCAAGCUGAGGGCCACCGGCGUGGCCAUUUCGAUGGUGGCCGCUCGCUUGGGCGGCAUCAUUGGCAACAUCGUGAUUGCCCAGCUGCUGGACA ........................(((...(((((..((((((....)))))).....((((((.(((((((....)))))))))))))(((((........))))))))))..)))... (-43.14 = -43.10 + -0.04)

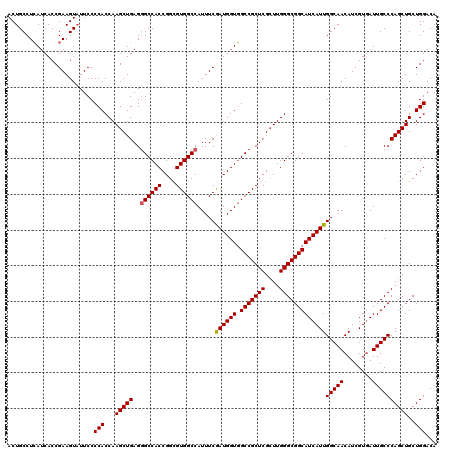
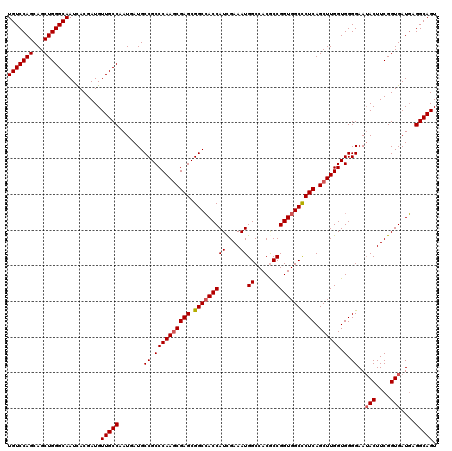
| Location | 6,566,928 – 6,567,048 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.50 |
| Mean single sequence MFE | -52.51 |
| Consensus MFE | -46.72 |
| Energy contribution | -47.40 |
| Covariance contribution | 0.68 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.89 |
| SVM RNA-class probability | 0.981422 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6566928 120 + 22224390 GCCCAAGCGAGCGGCCACCAUUGAAAUGGCCACGCCGGUGGCUCUCAGCUUGGUGGGGAAUACUUCGGUGAUGAGGCAGUCGAGCGCUGCAUUGGCCGCCGAAAUGGCACCACUGAAAAU (((....((.(((((((((((..(..(((((((....)))))...))..)..))))....(((....))).....(((((.....)))))..)))))))))....)))............ ( -47.50) >DroSec_CAF1 11890 120 + 1 GCCCAAGCGAGCGGCCACCAUUGAAAUGGCCACGCCGGUGGCCCUCAGCUUGGUGGGGAAUACUUCGGUGAUGAGGCAGUCGAGCGCCGCAUUGGCCGCCGAAAUGGCGCCACUGAAGAU .((((((((((.(((((((((....))(((...))))))))))))).)))))).).......(((((((((((.(((........))).))))(((.(((.....))))))))))))).. ( -54.80) >DroSim_CAF1 22416 120 + 1 GCCCAAGCGAGCGGCCACCAUCGAAAUGGCCACGCCGGUGGCCCUCAGCUUGGUGGGGAAUACUUCGGUGAUGAGGCAGUCGAGCGCCGCAUUGGCCGCCGAAAUGGCGCCACUGAAGAU .((((((((((.(((((((........(((...))))))))))))).)))))).).......(((((((((((.(((........))).))))(((.(((.....))))))))))))).. ( -54.70) >DroEre_CAF1 11516 120 + 1 GCCCAAGCGAGCGGCCACCAUCGAAAUGGCCACACCGGUGGCCCUCAGCUUGGUGGGGAAAACUUCGGUGAUGAGGCAGUCGAGCGCCGCAUUGGCCGCCGAGAUGGCGCCACUGAAGAU .((((((((((.(((((((.................)))))))))).)))))).).......(((((((((((.(((........))).))))(((.(((.....))))))))))))).. ( -52.83) >DroYak_CAF1 21334 120 + 1 GCCCAAUCGAGCGGCGACCAUCGAAAUGGCCACACCGGUGGCCCUCAGCUUGGUGGGGAAUACUUCGGUGAUCAGGCAGUCGAGCGCCGCAUUGGCUGCCGAGAUGGCGCCACUGAAGAU .(((.(((((((..((.....))....((((((....))))))....))))))).)))....(((((((((((.(((((((((((...)).)))))))))..))).....)))))))).. ( -52.70) >consensus GCCCAAGCGAGCGGCCACCAUCGAAAUGGCCACGCCGGUGGCCCUCAGCUUGGUGGGGAAUACUUCGGUGAUGAGGCAGUCGAGCGCCGCAUUGGCCGCCGAAAUGGCGCCACUGAAGAU .((((((((((.(((((((((....))((.....)))))))))))).)))))).).......(((((((((((.(((........))).))))(((.(((.....))))))))))))).. (-46.72 = -47.40 + 0.68)


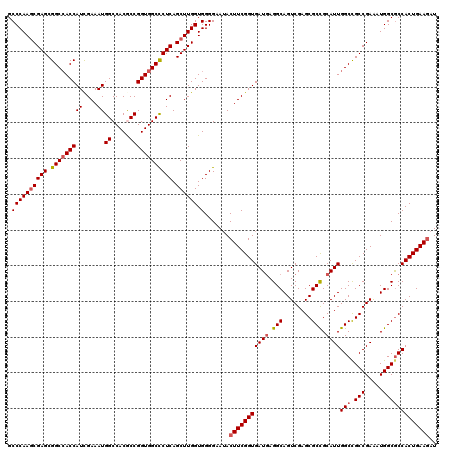
| Location | 6,566,928 – 6,567,048 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.50 |
| Mean single sequence MFE | -48.84 |
| Consensus MFE | -44.86 |
| Energy contribution | -45.30 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.31 |
| Structure conservation index | 0.92 |
| SVM decision value | 0.72 |
| SVM RNA-class probability | 0.833181 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6566928 120 - 22224390 AUUUUCAGUGGUGCCAUUUCGGCGGCCAAUGCAGCGCUCGACUGCCUCAUCACCGAAGUAUUCCCCACCAAGCUGAGAGCCACCGGCGUGGCCAUUUCAAUGGUGGCCGCUCGCUUGGGC ..(((((((((((..(((((((.((.....(((((....).))))....)).)))))))......))))..)))))))(((.(.((((((((((((.....)))))))))..))).)))) ( -44.80) >DroSec_CAF1 11890 120 - 1 AUCUUCAGUGGCGCCAUUUCGGCGGCCAAUGCGGCGCUCGACUGCCUCAUCACCGAAGUAUUCCCCACCAAGCUGAGGGCCACCGGCGUGGCCAUUUCAAUGGUGGCCGCUCGCUUGGGC ..((((..(((((((.....))).))))(((.((((......)))).)))....)))).........((((((.(((((((((((...(((.....))).)))))))).))))))))).. ( -51.50) >DroSim_CAF1 22416 120 - 1 AUCUUCAGUGGCGCCAUUUCGGCGGCCAAUGCGGCGCUCGACUGCCUCAUCACCGAAGUAUUCCCCACCAAGCUGAGGGCCACCGGCGUGGCCAUUUCGAUGGUGGCCGCUCGCUUGGGC ..((((..(((((((.....))).))))(((.((((......)))).)))....)))).........((((((.(((((((((((.((.........)).)))))))).))))))))).. ( -51.60) >DroEre_CAF1 11516 120 - 1 AUCUUCAGUGGCGCCAUCUCGGCGGCCAAUGCGGCGCUCGACUGCCUCAUCACCGAAGUUUUCCCCACCAAGCUGAGGGCCACCGGUGUGGCCAUUUCGAUGGUGGCCGCUCGCUUGGGC ..((((..(((((((.....))).))))(((.((((......)))).)))....)))).........((((((.(((((((((((...(((.....))).)))))))).))))))))).. ( -50.80) >DroYak_CAF1 21334 120 - 1 AUCUUCAGUGGCGCCAUCUCGGCAGCCAAUGCGGCGCUCGACUGCCUGAUCACCGAAGUAUUCCCCACCAAGCUGAGGGCCACCGGUGUGGCCAUUUCGAUGGUCGCCGCUCGAUUGGGC ....(((((((((((((((((((.(((.....))))).)))..(((....(((((..((...(((((......)).)))..))))))).)))......))))).))))))).))...... ( -45.50) >consensus AUCUUCAGUGGCGCCAUUUCGGCGGCCAAUGCGGCGCUCGACUGCCUCAUCACCGAAGUAUUCCCCACCAAGCUGAGGGCCACCGGCGUGGCCAUUUCGAUGGUGGCCGCUCGCUUGGGC ..((((..(((((((.....))).))))(((.((((......)))).)))....)))).........((((((.(((((((((((...(((.....))).)))))))).))))))))).. (-44.86 = -45.30 + 0.44)

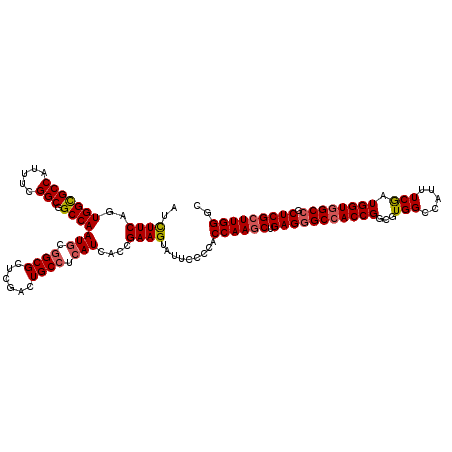
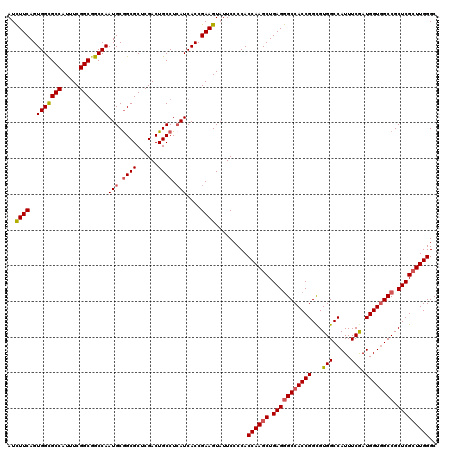
| Location | 6,567,008 – 6,567,128 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.33 |
| Mean single sequence MFE | -49.06 |
| Consensus MFE | -47.72 |
| Energy contribution | -47.80 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.20 |
| Structure conservation index | 0.97 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.740484 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6567008 120 - 22224390 CCGGUACCAUGACCGCCGGCAUCUGCUCGGCGUUGAUGUACUUUGUGCGCAGCUCCGUUCAGAACCUGGUCGUAUCGGCCAUUUUCAGUGGUGCCAUUUCGGCGGCCAAUGCAGCGCUCG ..(((((.((.(.(((((((....)).))))).).)))))))..(.((((.((........(((..((((((...))))))..)))..(((((((.....))).))))..)).)))).). ( -44.80) >DroSec_CAF1 11970 120 - 1 CCGGUACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUACUUUGUGCGCAGCUCCGUUCAGAACCUGGUCGUAUCGGCCAUCUUCAGUGGCGCCAUUUCGGCGGCCAAUGCGGCGCUCG ..........((.(((((.((((((..(((.((((.(((((...))))))))).)))..))))...(((((......(((((.....)))))(((.....)))))))).))))))).)). ( -50.60) >DroSim_CAF1 22496 120 - 1 CCGGUACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUACUUUGUGCGCAGCUCCGUUCAGAACCUGGUCGUAUCGGCCAUCUUCAGUGGCGCCAUUUCGGCGGCCAAUGCGGCGCUCG ..........((.(((((.((((((..(((.((((.(((((...))))))))).)))..))))...(((((......(((((.....)))))(((.....)))))))).))))))).)). ( -50.60) >DroEre_CAF1 11596 120 - 1 CCGGCACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUACUUUGUGCGCAGCUCCGUUCAGAACCUGGUCGUAUCGGCCAUCUUCAGUGGCGCCAUCUCGGCGGCCAAUGCGGCGCUCG ..........((.(((((.((((((..(((.((((.(((((...))))))))).)))..))))...(((((......(((((.....)))))(((.....)))))))).))))))).)). ( -50.60) >DroYak_CAF1 21414 120 - 1 CCGGCACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUACUUUGUGCGCAGCUCCGUUCAGAACCUGGUCGUAUCGGCCAUCUUCAGUGGCGCCAUCUCGGCAGCCAAUGCGGCGCUCG ..........((.(((((.((((((..(((.((((.(((((...))))))))).)))..))))...((((((...)))))).......(((((((.....))).)))).))))))).)). ( -48.70) >consensus CCGGUACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUACUUUGUGCGCAGCUCCGUUCAGAACCUGGUCGUAUCGGCCAUCUUCAGUGGCGCCAUUUCGGCGGCCAAUGCGGCGCUCG ..........((.(((((.((((((..(((.((((.(((((...))))))))).)))..))))...(((((......(((((.....)))))(((.....)))))))).))))))).)). (-47.72 = -47.80 + 0.08)



| Location | 6,567,048 – 6,567,168 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.83 |
| Mean single sequence MFE | -51.68 |
| Consensus MFE | -49.14 |
| Energy contribution | -48.78 |
| Covariance contribution | -0.36 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.27 |
| Structure conservation index | 0.95 |
| SVM decision value | 3.34 |
| SVM RNA-class probability | 0.999046 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6567048 120 - 22224390 CUGGGCAUGGACAUGCUGGGCAGGAAGUUCUUCCUAAUCGCCGGUACCAUGACCGCCGGCAUCUGCUCGGCGUUGAUGUACUUUGUGCGCAGCUCCGUUCAGAACCUGGUCGUAUCGGCC ((.(((((....))))).)).(((((....)))))....((((((((...(((((..((..((((..(((.((((.(((((...))))))))).)))..)))).))))))))))))))). ( -49.10) >DroSec_CAF1 12010 120 - 1 CUGGGCAUGGACAUGCUGGGCAGGAAGUUCUUCCUAAUCGCCGGUACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUACUUUGUGCGCAGCUCCGUUCAGAACCUGGUCGUAUCGGCC ((.(((((....))))).)).(((((....)))))....((((((((...(((((..((..((((..(((.((((.(((((...))))))))).)))..)))).))))))))))))))). ( -51.50) >DroSim_CAF1 22536 120 - 1 CUGGGCAUGGACAUGCUGGGCAGGAAGUUCUUCCUAAUCGCCGGUACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUACUUUGUGCGCAGCUCCGUUCAGAACCUGGUCGUAUCGGCC ((.(((((....))))).)).(((((....)))))....((((((((...(((((..((..((((..(((.((((.(((((...))))))))).)))..)))).))))))))))))))). ( -51.50) >DroEre_CAF1 11636 120 - 1 CUGGGCAUGGAUAUGCUGGGCAGGAAGUUCUUCCUCAUCGCCGGCACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUACUUUGUGCGCAGCUCCGUUCAGAACCUGGUCGUAUCGGCC ...(((...(((((((..((.(((((....)))))....((((((.........)))))).((((..(((.((((.(((((...))))))))).)))..)))).))..).)))))).))) ( -53.10) >DroYak_CAF1 21454 120 - 1 CUCGGCAUGGACAUGCUGGGUAGGAAGUUCUUCCUAAUCGCCGGCACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUACUUUGUGCGCAGCUCCGUUCAGAACCUGGUCGUAUCGGCC ((((((((....))))))))((((((....))))))...((((((.........)))))).((((..(((.((((.(((((...))))))))).)))..))))....(((((...))))) ( -53.20) >consensus CUGGGCAUGGACAUGCUGGGCAGGAAGUUCUUCCUAAUCGCCGGUACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUACUUUGUGCGCAGCUCCGUUCAGAACCUGGUCGUAUCGGCC ((((((..(((...((((((((((((....)))))....((((((.........))))))....)))))))((((.(((((...)))))))))))))))))).....(((((...))))) (-49.14 = -48.78 + -0.36)

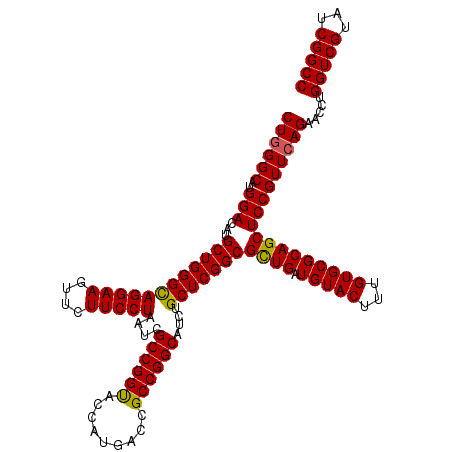
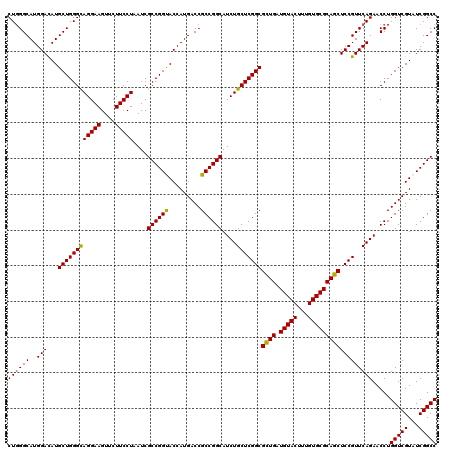
| Location | 6,567,088 – 6,567,208 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.17 |
| Mean single sequence MFE | -49.66 |
| Consensus MFE | -47.32 |
| Energy contribution | -47.36 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.95 |
| SVM decision value | 2.53 |
| SVM RNA-class probability | 0.994953 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6567088 120 + 22224390 UACAUCAACGCCGAGCAGAUGCCGGCGGUCAUGGUACCGGCGAUUAGGAAGAACUUCCUGCCCAGCAUGUCCAUGCCCAGAAUGGCCAGCAGAUUGGCCGGCAGCGCUGAGGCUAGUGAU ........(((((.((....)))))))((((((((..(((((...(((((....)))))(((..((((....)))).......((((((....)))))))))..)))))..)))).)))) ( -48.00) >DroSec_CAF1 12050 120 + 1 UACAUCAGCGCCGAGCAGAUGCCGGCGGUCAUGGUACCGGCGAUUAGGAAGAACUUCCUGCCCAGCAUGUCCAUGCCCAGAAUGGCCAGCAGAUUGGCCGGCAGCGCUGAGGCCAGUGAU ........(((((.((....)))))))((((((((..(((((...(((((....)))))(((..((((....)))).......((((((....)))))))))..)))))..)))).)))) ( -50.00) >DroSim_CAF1 22576 120 + 1 UACAUCAGCGCCGAGCAGAUGCCGGCGGUCAUGGUACCGGCGAUUAGGAAGAACUUCCUGCCCAGCAUGUCCAUGCCCAGAAUGGCCAGCAGAUUGGCCGGCAGCGCGGAGGCCAGUGAU ........(((((.((....)))))))((((((((.(((((....(((((....)))))(((..((((....)))).......((((((....))))))))).)).)))..)))).)))) ( -48.30) >DroEre_CAF1 11676 120 + 1 UACAUCAGCGCCGAGCAGAUGCCGGCGGUCAUGGUGCCGGCGAUGAGGAAGAACUUCCUGCCCAGCAUAUCCAUGCCCAGGAUGGCCAACAGAUUGGCCGGCAGCGCCGAGGCCAGUGAU ........(((((.((....)))))))((((((((..(((((...(((((....)))))(((......((((.......))))((((((....)))))))))..)))))..)))).)))) ( -51.90) >DroYak_CAF1 21494 120 + 1 UACAUCAGCGCCGAGCAGAUGCCGGCGGUCAUGGUGCCGGCGAUUAGGAAGAACUUCCUACCCAGCAUGUCCAUGCCGAGGAUGGCCAAAAGAUUGGCGGGCAGCGCCGAGGCCAGUGAU ........(((((.((....)))))))((((((((..(((((..((((((....))))))(((.((((....))))........(((((....))))))))...)))))..)))).)))) ( -50.10) >consensus UACAUCAGCGCCGAGCAGAUGCCGGCGGUCAUGGUACCGGCGAUUAGGAAGAACUUCCUGCCCAGCAUGUCCAUGCCCAGAAUGGCCAGCAGAUUGGCCGGCAGCGCCGAGGCCAGUGAU ........(((((.((....)))))))((((((((..(((((...(((((....)))))(((..((((....)))).......((((((....)))))))))..)))))..)))).)))) (-47.32 = -47.36 + 0.04)

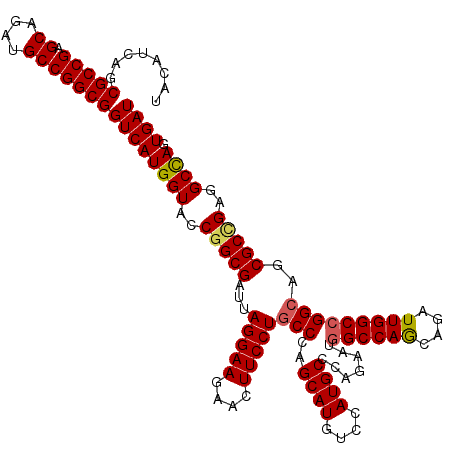
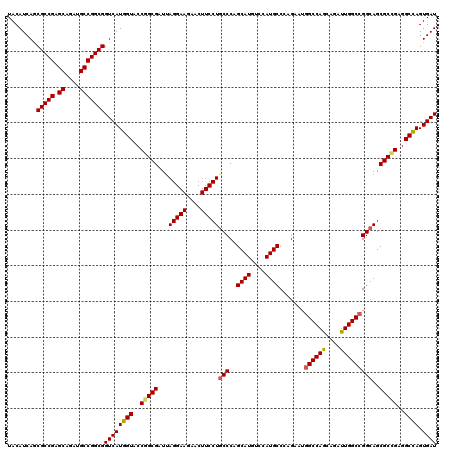
| Location | 6,567,088 – 6,567,208 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.17 |
| Mean single sequence MFE | -50.78 |
| Consensus MFE | -48.40 |
| Energy contribution | -48.08 |
| Covariance contribution | -0.32 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.95 |
| SVM decision value | 3.82 |
| SVM RNA-class probability | 0.999637 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6567088 120 - 22224390 AUCACUAGCCUCAGCGCUGCCGGCCAAUCUGCUGGCCAUUCUGGGCAUGGACAUGCUGGGCAGGAAGUUCUUCCUAAUCGCCGGUACCAUGACCGCCGGCAUCUGCUCGGCGUUGAUGUA ((((((((((.(((....((((((......))))))....)))))).)))..((((((((((((((....)))))....((((((.........))))))....)))))))))))))... ( -50.80) >DroSec_CAF1 12050 120 - 1 AUCACUGGCCUCAGCGCUGCCGGCCAAUCUGCUGGCCAUUCUGGGCAUGGACAUGCUGGGCAGGAAGUUCUUCCUAAUCGCCGGUACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUA .......((.((((((((((((((......)))))).......(((((....)))))(((((((((....)))))....((((((.........))))))....)))))))))))).)). ( -51.50) >DroSim_CAF1 22576 120 - 1 AUCACUGGCCUCCGCGCUGCCGGCCAAUCUGCUGGCCAUUCUGGGCAUGGACAUGCUGGGCAGGAAGUUCUUCCUAAUCGCCGGUACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUA ...((((((.........((((((((......)))))......(((((....))))).)))(((((....)))))....))))))..(((.(.(((((((....)).))))).).))).. ( -47.80) >DroEre_CAF1 11676 120 - 1 AUCACUGGCCUCGGCGCUGCCGGCCAAUCUGUUGGCCAUCCUGGGCAUGGAUAUGCUGGGCAGGAAGUUCUUCCUCAUCGCCGGCACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUA .......((.((((((((((.((((((....))))))((((.......))))..)).(((((((((....)))).....((((((.........))))))...))))))))))))).)). ( -52.40) >DroYak_CAF1 21494 120 - 1 AUCACUGGCCUCGGCGCUGCCCGCCAAUCUUUUGGCCAUCCUCGGCAUGGACAUGCUGGGUAGGAAGUUCUUCCUAAUCGCCGGCACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUA .......((.(((((((((..((((((....)))))....((((((((....))))))))((((((....))))))...((((((.........))))))....)..))))))))).)). ( -51.40) >consensus AUCACUGGCCUCAGCGCUGCCGGCCAAUCUGCUGGCCAUUCUGGGCAUGGACAUGCUGGGCAGGAAGUUCUUCCUAAUCGCCGGUACCAUGACCGCCGGCAUCUGCUCGGCGCUGAUGUA .......((.((((((((...(((((......)))))......(((((....)))))(((((((((....)))))....((((((.........))))))....)))))))))))).)). (-48.40 = -48.08 + -0.32)

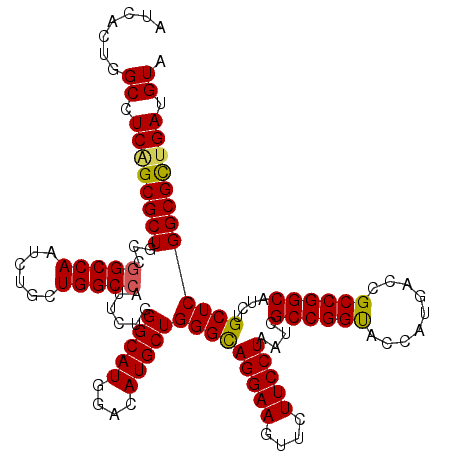
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:34:55 2006