
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 6,566,423 – 6,566,580 |
| Length | 157 |
| Max. P | 0.999950 |

| Location | 6,566,423 – 6,566,543 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.50 |
| Mean single sequence MFE | -27.18 |
| Consensus MFE | -27.34 |
| Energy contribution | -27.18 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.01 |
| Structure conservation index | 1.01 |
| SVM decision value | 2.96 |
| SVM RNA-class probability | 0.997915 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
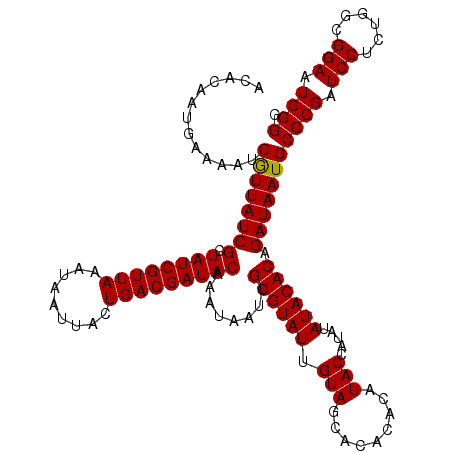
>X_DroMel_CAF1 6566423 120 + 22224390 ACACAAUGAAAAUCGUUAUCGCUAUCGUUAAAUAAUUACUGACGAUACAAAUAAUCGUGUAUUGUAGCACACACAUACCAUAUAUACACAGAUAAUGCCCGAUCCUCUGGCGGAAUGGGG .............((((((((.((((((((.........)))))))))........((((((.(((.........))).....)))))).)))))))((((.(((......))).)))). ( -27.10) >DroSec_CAF1 11387 120 + 1 ACACAGUGAAAAUCGUUAUCGCUAUCGUUAAAUAAUUACUGACGAUACAAAUAAUCGUGUAUUGUAGCACACACAUACCAUAUAUACACAGAUAAUGCCCGAUCCUCUGGCGGAAUGGGG .............((((((((.((((((((.........)))))))))........((((((.(((.........))).....)))))).)))))))((((.(((......))).)))). ( -27.10) >DroSim_CAF1 21913 120 + 1 ACACAGUGAAAAUCGUUAUCGCUAUCGUUAAAUAAUUACUGACGAUACAAAUAAUCGUGUAUUGUAGCACACACAUACCAUAUAUACACAGAUAAUGCCCGAUCCUCUGACGGAAUGGGG .............((((((((.((((((((.........)))))))))........((((((.(((.........))).....)))))).)))))))((((.(((......))).)))). ( -27.10) >DroEre_CAF1 11017 120 + 1 ACUCAAUGAAAAUCGUUAUCGCUAUCGUUAAAUAAUUACUGACGAUACAAAUAAUCGUGUAUUGUAGCACACACAUACCAUAUAUACACAGAUAAUGCCCGAUCCUCUGGCGGAAUGGGG .............((((((((.((((((((.........)))))))))........((((((.(((.........))).....)))))).)))))))((((.(((......))).)))). ( -27.10) >DroYak_CAF1 20849 120 + 1 ACACAAUGAAAAUCAUUAUCGCUAUCGUUAAAUAAUUACUGACGAUACAAAUAAUCGUGUAUUGUAGCACACACAUACCAUAUAUACACAGAUAAUGCCCGAUCCUCUGGCGGAAUGGGG .............((((((((.((((((((.........)))))))))........((((((.(((.........))).....)))))).)))))))((((.(((......))).)))). ( -27.50) >consensus ACACAAUGAAAAUCGUUAUCGCUAUCGUUAAAUAAUUACUGACGAUACAAAUAAUCGUGUAUUGUAGCACACACAUACCAUAUAUACACAGAUAAUGCCCGAUCCUCUGGCGGAAUGGGG .............((((((((.((((((((.........)))))))))........((((((.(((.........))).....)))))).)))))))((((.(((......))).)))). (-27.34 = -27.18 + -0.16)

| Location | 6,566,463 – 6,566,580 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.19 |
| Mean single sequence MFE | -32.10 |
| Consensus MFE | -31.82 |
| Energy contribution | -32.22 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.99 |
| SVM decision value | 4.79 |
| SVM RNA-class probability | 0.999950 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6566463 117 - 22224390 ---AAAAAACCACCCGCGACCCACAAGUAAUUUCCAUUUGCCCCAUUCCGCCAGAGGAUCGGGCAUUAUCUGUGUAUAUAUGGUAUGUGUGUGCUACAAUACACGAUUAUUUGUAUCGUC ---............((((...((((((((((......(((((...(((......)))..)))))......((((((...((((((....))))))..)))))))))))))))).)))). ( -32.80) >DroSec_CAF1 11427 116 - 1 ---AA-AACCCACCCGCGACCCACAUUUAAUUUCCAUUUGCCCCAUUCCGCCAGAGGAUCGGGCAUUAUCUGUGUAUAUAUGGUAUGUGUGUGCUACAAUACACGAUUAUUUGUAUCGUC ---..-.........((((...(((..(((((......(((((...(((......)))..)))))......((((((...((((((....))))))..)))))))))))..))).)))). ( -29.30) >DroSim_CAF1 21953 116 - 1 ---AA-AACCCACCCGCGACCCACAAGUAAUUUCCAUUUGCCCCAUUCCGUCAGAGGAUCGGGCAUUAUCUGUGUAUAUAUGGUAUGUGUGUGCUACAAUACACGAUUAUUUGUAUCGUC ---..-.........((((...((((((((((......(((((...(((......)))..)))))......((((((...((((((....))))))..)))))))))))))))).)))). ( -32.80) >DroEre_CAF1 11057 119 - 1 AUCCC-ACCCCACCCGCGACCCACAAGUAAUUUCCAUUUGCCCCAUUCCGCCAGAGGAUCGGGCAUUAUCUGUGUAUAUAUGGUAUGUGUGUGCUACAAUACACGAUUAUUUGUAUCGUC .....-.........((((...((((((((((......(((((...(((......)))..)))))......((((((...((((((....))))))..)))))))))))))))).)))). ( -32.80) >DroYak_CAF1 20889 119 - 1 AGCCC-ACCCCACCCGCGACCCACAAGUAAUUUCCAUUUGCCCCAUUCCGCCAGAGGAUCGGGCAUUAUCUGUGUAUAUAUGGUAUGUGUGUGCUACAAUACACGAUUAUUUGUAUCGUC .....-.........((((...((((((((((......(((((...(((......)))..)))))......((((((...((((((....))))))..)))))))))))))))).)))). ( -32.80) >consensus ___AA_AACCCACCCGCGACCCACAAGUAAUUUCCAUUUGCCCCAUUCCGCCAGAGGAUCGGGCAUUAUCUGUGUAUAUAUGGUAUGUGUGUGCUACAAUACACGAUUAUUUGUAUCGUC ...............((((...((((((((((......(((((...(((......)))..)))))......((((((...((((((....))))))..)))))))))))))))).)))). (-31.82 = -32.22 + 0.40)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:34:44 2006