
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 6,561,966 – 6,562,106 |
| Length | 140 |
| Max. P | 0.956870 |

| Location | 6,561,966 – 6,562,080 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 76.38 |
| Mean single sequence MFE | -45.43 |
| Consensus MFE | -24.01 |
| Energy contribution | -25.52 |
| Covariance contribution | 1.50 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.95 |
| SVM RNA-class probability | 0.888937 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
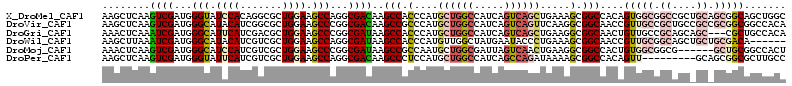
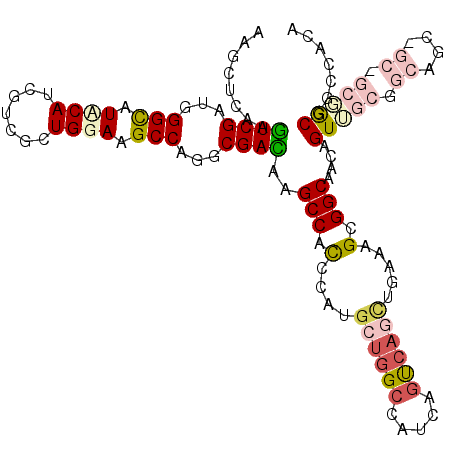
>X_DroMel_CAF1 6561966 114 + 22224390 AAGCUCAAGUCGAUGGGUAUCCACAGGCGCUGGAAGCCAGGCGACAAGCCACCCAUGCUGGCCAUCAGUCAGCUGAAAGCGGCCACAGUGGCGGCCGCUGCAGCGGCAGCUGGC .((((...((((...(((.((((.......)))).)))...))))..(((((...((.(((((.((((....))))....)))))))))))).(((((....)))))))))... ( -49.60) >DroVir_CAF1 946 114 + 1 AAGCUCAAGUCGAUGGGCAUACAUCGGCGCUGGAAGCCCGGCGACAAGCCGCCCAUGCUGGCCAUCAGUCAGUUCAAGGCGGCAACCGUUGCCGCUGCCGCCGCGGCGGCCACA ..(.((..(((((((......)))))))(((((....))))))))..((((((...((((((.....))))))....))))))....((.((((((((....)))))))).)). ( -58.00) >DroGri_CAF1 9640 111 + 1 AAACUCAAAUCGAUGGGCAUUCAUCGACGCUGGAAGCCCGGCGAUAAGCCACCCAUGCUGGCCAUCAGUCAGCUGAAGGCGGCAACUGUUGCCGCAGCAGC---CGCUGCCACA .........(((((((....)))))))((((((....))))))....(((...((.((((((.....))))))))..)))((((.....))))(((((...---.))))).... ( -43.90) >DroWil_CAF1 9561 108 + 1 AAGCUUAAAUCGAUGGGCAUACAUCGUCGCUGGAAGCCAGGCGAUAAGCCACCCAUGUUGGCUAUGAAUACCCUGAAAGCGGCAACCGUUGCGGCAGCUGCUGCGACA------ ..((((...(((..(((.((.(((.((((((((...))).))))).(((((.......)))))))).)).))))))))))(....).((((((((....)))))))).------ ( -36.10) >DroMoj_CAF1 9651 108 + 1 AAACUCAAGUCGAUGGGCAUCCAUCGUCGCUGGAAGCCCGGCGAUAAGCCGCCAAUGCUGGCGAUUAGUCAACUGAAGGCGGCCACUGUGGCGGCG------GCUGCGGCCACU ...........(((((....)))))((((((((....))))))))..(((((((..((((.(..((((....)))).).)))).....)))))))(------((....)))... ( -48.60) >DroPer_CAF1 14037 105 + 1 AAGCUCAAGUCGAUGGGUAUUCAUCGUCGCUGGAAGCCAGGCGACAAGCCCUCCAUGCUGGCCAUCAGCCAGAUAAAAGCGGCCACAGUU---------GCAGCGGCGCUUGCC ((((.......(((((....)))))(((((((.(((((.(((.....))).......(((((.....)))))........))).....))---------.)))))))))))... ( -36.40) >consensus AAGCUCAAGUCGAUGGGCAUACAUCGUCGCUGGAAGCCAGGCGACAAGCCACCCAUGCUGGCCAUCAGUCAGCUGAAAGCGGCAACAGUUGCGGCAGC_GC_GCGGCGGCCACA ........((((...(((.((((.......)))).)))...))))..(((.(....((((((.....)))))).....).)))....(((((.((....)).)))))....... (-24.01 = -25.52 + 1.50)
| Location | 6,562,006 – 6,562,106 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 83.18 |
| Mean single sequence MFE | -59.38 |
| Consensus MFE | -46.38 |
| Energy contribution | -47.58 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.23 |
| Mean z-score | -3.81 |
| Structure conservation index | 0.78 |
| SVM decision value | 1.47 |
| SVM RNA-class probability | 0.956870 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6562006 100 + 22224390 GCGACAAGCCACCCAUGCUGGCCAUCAGUCAGCUGAAAGCGGCCACAGUGGCGGCCGCUGCAGCGGCAGCUGGCUUGGCCGGCGUCGGCGGUGUUGUUGG-------------- .(((((((((.((.((((((((((..(((((((((..(((((((.(....).))))))).......))))))))))))))))))).)).))).)))))).-------------- ( -61.20) >DroVir_CAF1 986 108 + 1 GCGACAAGCCGCCCAUGCUGGCCAUCAGUCAGUUCAAGGCGGCAACCGUUGCCGCUGCCGCCGCGGCGGCCACAUUGGCGGCGAAUGGCG------CCAGCUUUGGUCUGCCGA (((((..((((((...((((((.....))))))....))))))....((.((((((((....)))))))).)).((((((.(....).))------)))).....))).))... ( -52.70) >DroSec_CAF1 7077 100 + 1 GUGACAAGCCACCGAUGCUGGCCAUCAGUCAGCUGAAAGCGGCCACAGUGGCGGCCGCCGCAGCGGCAGCUGGCUUGGCCGGCGUCGGCGGUGUUGUUGG-------------- ..((((((((.(((((((((((((..(((((((((...(((....).(((((....))))).))..)))))))))))))))))))))).))).)))))..-------------- ( -62.20) >DroSim_CAF1 17297 100 + 1 GGGACAAGCCACCGAUGCUGGCCAUCAGUCAGCUGAAAGCGGCCACAGUGGCGGCCGCCGCAGCGGCAGCUGGCUUGGCCGGCGUCGGCGGUGUUGUUGG-------------- ..((((((((.(((((((((((((..(((((((((...(((....).(((((....))))).))..)))))))))))))))))))))).))).)))))..-------------- ( -62.40) >DroEre_CAF1 6742 100 + 1 GCGACAAGCCACCCAUGCUGGCCAUCAGCCAGCUGAAAGCGGCCACAGUGGCGGCCGCCGCAGCGGCAGCUGGCUUGGCCGGCGUCGGCGGUGUCGUUGG-------------- (((((..(((.((.((((((((((..(((((((((...(((....).(((((....))))).))..))))))))))))))))))).)).))))))))...-------------- ( -61.10) >DroYak_CAF1 16553 100 + 1 GCGACAAGCCACCCAUGCUGGCCAUCAGCCAGCUGAAAGCGGCCACAGUGGCGGCUGCCGCAGCGGCAGCUGGCUUAGCCGGCGUCGGCGGUGUCGUUGG-------------- (((((..(((.((.((((((((....((((((((...(((.(((.....))).)))((((...))))))))))))..)))))))).)).))))))))...-------------- ( -56.70) >consensus GCGACAAGCCACCCAUGCUGGCCAUCAGUCAGCUGAAAGCGGCCACAGUGGCGGCCGCCGCAGCGGCAGCUGGCUUGGCCGGCGUCGGCGGUGUUGUUGG______________ .((((..(((.((.((((((((((..((((((((.......(((.....))).(((((....))))))))))))))))))))))).)).)))...))))............... (-46.38 = -47.58 + 1.20)



| Location | 6,562,006 – 6,562,106 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 83.18 |
| Mean single sequence MFE | -53.57 |
| Consensus MFE | -39.76 |
| Energy contribution | -40.32 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.23 |
| Mean z-score | -3.09 |
| Structure conservation index | 0.74 |
| SVM decision value | 1.01 |
| SVM RNA-class probability | 0.899594 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6562006 100 - 22224390 --------------CCAACAACACCGCCGACGCCGGCCAAGCCAGCUGCCGCUGCAGCGGCCGCCACUGUGGCCGCUUUCAGCUGACUGAUGGCCAGCAUGGGUGGCUUGUCGC --------------...((((..(((((.(.((.(((((((.((((((.......(((((((((....)))))))))..)))))).))..))))).)).).))))).))))... ( -48.70) >DroVir_CAF1 986 108 - 1 UCGGCAGACCAAAGCUGG------CGCCAUUCGCCGCCAAUGUGGCCGCCGCGGCGGCAGCGGCAACGGUUGCCGCCUUGAACUGACUGAUGGCCAGCAUGGGCGGCUUGUCGC .((((........))))(------((.((...((((((.(((((((((.((.((((((((((....).))))))))).))..(.....).)))))).))).)))))).)).))) ( -53.10) >DroSec_CAF1 7077 100 - 1 --------------CCAACAACACCGCCGACGCCGGCCAAGCCAGCUGCCGCUGCGGCGGCCGCCACUGUGGCCGCUUUCAGCUGACUGAUGGCCAGCAUCGGUGGCUUGUCAC --------------...((((..(((((((.((.(((((((.((((((.......(((((((((....)))))))))..)))))).))..))))).)).))))))).))))... ( -52.50) >DroSim_CAF1 17297 100 - 1 --------------CCAACAACACCGCCGACGCCGGCCAAGCCAGCUGCCGCUGCGGCGGCCGCCACUGUGGCCGCUUUCAGCUGACUGAUGGCCAGCAUCGGUGGCUUGUCCC --------------...((((..(((((((.((.(((((((.((((((.......(((((((((....)))))))))..)))))).))..))))).)).))))))).))))... ( -52.50) >DroEre_CAF1 6742 100 - 1 --------------CCAACGACACCGCCGACGCCGGCCAAGCCAGCUGCCGCUGCGGCGGCCGCCACUGUGGCCGCUUUCAGCUGGCUGAUGGCCAGCAUGGGUGGCUUGUCGC --------------....((((((((((.(.((.((((((((((((((.......(((((((((....)))))))))..)))))))))..))))).)).).)))))..))))). ( -60.40) >DroYak_CAF1 16553 100 - 1 --------------CCAACGACACCGCCGACGCCGGCUAAGCCAGCUGCCGCUGCGGCAGCCGCCACUGUGGCCGCUUUCAGCUGGCUGAUGGCCAGCAUGGGUGGCUUGUCGC --------------....((((((((((.(.((.((((((((((((((..((.((.((((......)))).)).))...)))))))))..))))).)).).)))))..))))). ( -54.20) >consensus ______________CCAACAACACCGCCGACGCCGGCCAAGCCAGCUGCCGCUGCGGCGGCCGCCACUGUGGCCGCUUUCAGCUGACUGAUGGCCAGCAUGGGUGGCUUGUCGC .......................(((((.(.((.(((((((.(((((((....))(((((((((....)))))))))...))))).))..))))).)).).)))))........ (-39.76 = -40.32 + 0.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:34:40 2006