
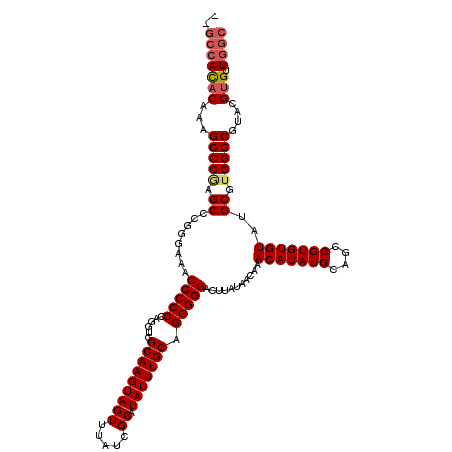
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 6,268,863 – 6,269,155 |
| Length | 292 |
| Max. P | 0.996938 |

| Location | 6,268,863 – 6,268,981 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.71 |
| Mean single sequence MFE | -41.16 |
| Consensus MFE | -37.36 |
| Energy contribution | -38.08 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.27 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.47 |
| SVM RNA-class probability | 0.750585 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6268863 118 + 22224390 --GCCACACAAAGCCCGAGCCCAGGAAACCCCCCAGGUGGCAAGUAGUUUAUCGCAUAUUUGCAGGGGGACUUAUAACAAACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGC --(((((((...(((((.((...(....)(((((.....(((((((((.....)).))))))).)))))...........(((((((....)))))))..)).)))))....))).)))) ( -42.00) >DroSec_CAF1 11035 118 + 1 --GCCACACAAAGCCCGAGCCCGGGAAACCCCCCAGGUGGCAAGUAGUUUAUCGCAUAUUUGCAGGGGGACUUAUAACAAACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGC --(((((((...(((((.((..((....))((((.....(((((((((.....)).)))))))..))))...........(((((((....)))))))..)).)))))....))).)))) ( -43.50) >DroSim_CAF1 12381 118 + 1 --GCCACACAAAGCCCGAGCCCGGGAAACCCCCCAGGUGGCAAGUAGUUUAUCGCAUAUUUGCAGGGGGACUUAUAACAAACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGC --(((((((...(((((.((..((....))((((.....(((((((((.....)).)))))))..))))...........(((((((....)))))))..)).)))))....))).)))) ( -43.50) >DroEre_CAF1 128 115 + 1 -----AUCCCAAGCCCAAGCCCGGGAAACCCCCCAGGUGGCAAGUAGUUUAUCGCAUAUUUGCAGGGGGACUUAUAACAAACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGC -----.((((..((....))..))))...(((((.....(((((((((.....)).))))))).)))))....................((((..(((((((....)))))))..).))) ( -37.30) >DroYak_CAF1 8162 120 + 1 CGGCCACACAAAGCCCCAGCCCGGAAAACCCCCAAGGUGGCAAGUAGUUUAUCGCAUAUUUGCAGGGGGACUUAUAACAAACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGC ..(((((((...((((..((........(((((......(((((((((.....)).))))))).)))))...........(((((((....)))))))..))..))))....))).)))) ( -39.50) >consensus __GCCACACAAAGCCCGAGCCCGGGAAACCCCCCAGGUGGCAAGUAGUUUAUCGCAUAUUUGCAGGGGGACUUAUAACAAACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGC ..(((((((...(((((.((........(((((......(((((((((.....)).))))))).)))))...........(((((((....)))))))..)).)))))....))).)))) (-37.36 = -38.08 + 0.72)


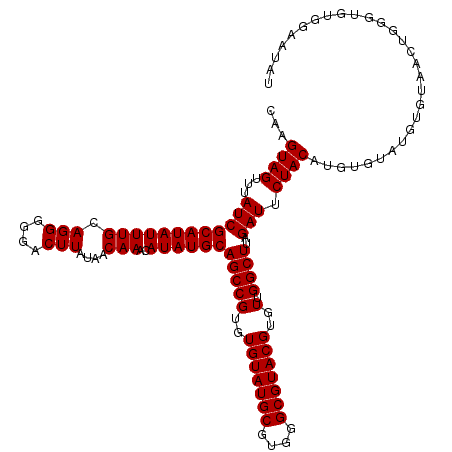
| Location | 6,268,901 – 6,269,021 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.25 |
| Mean single sequence MFE | -34.68 |
| Consensus MFE | -30.50 |
| Energy contribution | -30.50 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.30 |
| Structure conservation index | 0.88 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.544764 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6268901 120 + 22224390 CAAGUAGUUUAUCGCAUAUUUGCAGGGGGACUUAUAACAAACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGCUUAGAUUCUACAUGUGUUUGUGUAACUGGGUGUGGAAUAU ...........((((((..(((((((....)))...(((((((((((.(((((..(((((((....)))))))..).))))(((...))))))))))))))))))....))))))..... ( -36.90) >DroSec_CAF1 11073 120 + 1 CAAGUAGUUUAUCGCAUAUUUGCAGGGGGACUUAUAACAAACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGCUUAGAUUCUACAUGUGCAUGUGUAACUGGGUGUGGAAUAU ((..(((((....(((....)))(((....))).......(((((((((((((..(((((((....)))))))..).))).(((...)))....))))))))))))))..))........ ( -33.50) >DroSim_CAF1 12419 120 + 1 CAAGUAGUUUAUCGCAUAUUUGCAGGGGGACUUAUAACAAACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGCUUAGAUUCUACAUGUGUAUGUGUAACUGGGUGUGGAAUAU ...........((((((..(((((((....)))...(((.(((((((.(((((..(((((((....)))))))..).))))(((...)))))))))).)))))))....))))))..... ( -33.40) >DroEre_CAF1 163 120 + 1 CAAGUAGUUUAUCGCAUAUUUGCAGGGGGACUUAUAACAAACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGCUUAGAUUCUACAUGUGUGUGGGUAGCUGGGCGAGGAAUAU ......((((.((((.((((..((((....))).......(((((((.(((((..(((((((....)))))))..).))))(((...)))))))))))..)))).....)))).)))).. ( -35.40) >DroYak_CAF1 8202 120 + 1 CAAGUAGUUUAUCGCAUAUUUGCAGGGGGACUUAUAACAAACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGCUUAGAUUCUACAUGUGUAUGGGUAACUGGGUGUGCAAUAU ...((((...((((((((((((.(((....)))....)))..))))))(((((..(((((((....)))))))..).))))..))).)))).((..(((((....))..)))..)).... ( -34.20) >consensus CAAGUAGUUUAUCGCAUAUUUGCAGGGGGACUUAUAACAAACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGCUUAGAUUCUACAUGUGUAUGUGUAACUGGGUGUGGAAUAU ...((((...((((((((((((.(((....)))....)))..))))))(((((..(((((((....)))))))..).))))..))).))))............................. (-30.50 = -30.50 + 0.00)

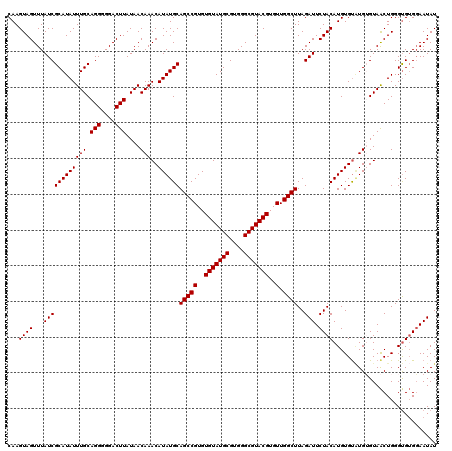
| Location | 6,268,941 – 6,269,052 |
|---|---|
| Length | 111 |
| Sequences | 3 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 90.62 |
| Mean single sequence MFE | -35.33 |
| Consensus MFE | -31.40 |
| Energy contribution | -31.40 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.75 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.44 |
| SVM RNA-class probability | 0.953888 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6268941 111 + 22224390 ACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGCUUAGAUUCUACAUGUGUUUGUGUAACUGGGUGUGGAAUAUGUGGGAUAUGUG----GAACAUGUGUAGGUAACGG (((((((.(((((..(((((((....)))))))..).))))....((((((((((.((..((((.((......))..))))..)))))))))----))))))))))......... ( -33.50) >DroSec_CAF1 11113 111 + 1 ACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGCUUAGAUUCUACAUGUGCAUGUGUAACUGGGUGUGGAAUAUGUGGGAUAUGUG----GAACAUGUGUAGGCGAAGG (((((((.(((((..(((((((....)))))))..).))))....((((((((((((((((...((.....))...))))))...)))))))----))))))))))......... ( -37.70) >DroYak_CAF1 8242 109 + 1 ACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGCUUAGAUUCUACAUGUGUAUGGGUAACUGGGUGUGCAAUAUGUGGGAUAUGUGUGGU-AACAUGUGUAGGU----- (((((((.(((((..(((((((....)))))))..).))))...(((((((((((((((((....))....)))).))))))))))).........-..)))))))....----- ( -34.80) >consensus ACAUAUGCAGCCGUGUGUAUGCGUGGGCGUACGUGUUGGCUUAGAUUCUACAUGUGUAUGUGUAACUGGGUGUGGAAUAUGUGGGAUAUGUG____GAACAUGUGUAGGU_A_GG (((((((.(((((..(((((((....)))))))..).))))...(((((((((((.....................)))))))))))............)))))))......... (-31.40 = -31.40 + 0.00)



| Location | 6,269,021 – 6,269,116 |
|---|---|
| Length | 95 |
| Sequences | 3 |
| Columns | 95 |
| Reading direction | forward |
| Mean pairwise identity | 87.99 |
| Mean single sequence MFE | -38.17 |
| Consensus MFE | -30.81 |
| Energy contribution | -30.60 |
| Covariance contribution | -0.21 |
| Combinations/Pair | 1.13 |
| Mean z-score | -4.58 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.38 |
| SVM RNA-class probability | 0.948768 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6269021 95 + 22224390 GUGGGAUAUGUGGAACAUGUGUAGGUAACGGUGUGUGUGUGCGUGAGUGUGUGUCUGAGAAUGGGUACACAUGUACACAUGUACCGCAGCCAUCU ...(.(((((.....))))).).(((..(((((..(((((((....((((((..((......))..)))))))))))))..)))))..))).... ( -37.30) >DroSec_CAF1 11193 93 + 1 GUGGGAUAUGUGGAACAUGUGUAGGCGAAGGUGUGUGUGCGAGUGUGGGUGUGGAU--ACACAGAUACACAUGUACACAUGUACCGCAGCCAUCU ...((...(((((.((((((.((.((....)).)).(((((.(((((..((((...--.))))..))))).))))))))))).))))).)).... ( -37.90) >DroSim_CAF1 12539 93 + 1 GUGGGAUAUGUGGAACAUGUGUAGGCGAAGGUGUGUGUGCGAGUGUGGGUGUGGAC--ACACAGAUACACAUGUACACAUGUACCGCAGCCAUCU ...((...(((((.(((((((((.((....))((((((((..(((((........)--)))).).))))))).))))))))).))))).)).... ( -39.30) >consensus GUGGGAUAUGUGGAACAUGUGUAGGCGAAGGUGUGUGUGCGAGUGUGGGUGUGGAU__ACACAGAUACACAUGUACACAUGUACCGCAGCCAUCU ...(.(((((.....))))).).(((...((((((((((((.(((((..((((......))))..))))).)))))))))..)))...))).... (-30.81 = -30.60 + -0.21)

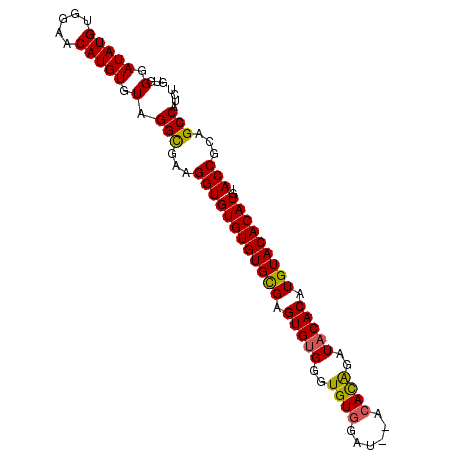
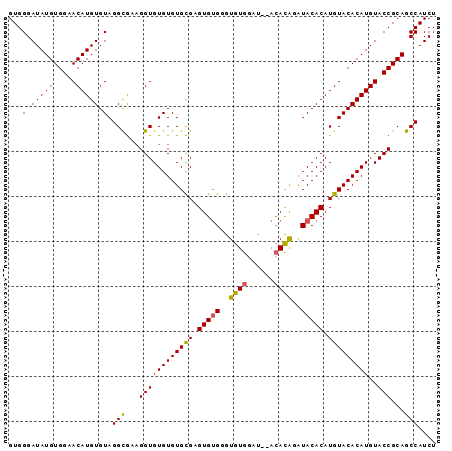
| Location | 6,269,021 – 6,269,116 |
|---|---|
| Length | 95 |
| Sequences | 3 |
| Columns | 95 |
| Reading direction | reverse |
| Mean pairwise identity | 87.99 |
| Mean single sequence MFE | -22.57 |
| Consensus MFE | -17.00 |
| Energy contribution | -18.00 |
| Covariance contribution | 1.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.09 |
| Structure conservation index | 0.75 |
| SVM decision value | 1.02 |
| SVM RNA-class probability | 0.900973 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6269021 95 - 22224390 AGAUGGCUGCGGUACAUGUGUACAUGUGUACCCAUUCUCAGACACACACUCACGCACACACACACCGUUACCUACACAUGUUCCACAUAUCCCAC .((((..((.((.(((((((((..(((((............))))).....(((...........)))....))))))))).)).)))))).... ( -18.60) >DroSec_CAF1 11193 93 - 1 AGAUGGCUGCGGUACAUGUGUACAUGUGUAUCUGUGU--AUCCACACCCACACUCGCACACACACCUUCGCCUACACAUGUUCCACAUAUCCCAC .((((..((.((.(((((((((...((((...((((.--...))))...))))..((............)).))))))))).)).)))))).... ( -22.60) >DroSim_CAF1 12539 93 - 1 AGAUGGCUGCGGUACAUGUGUACAUGUGUAUCUGUGU--GUCCACACCCACACUCGCACACACACCUUCGCCUACACAUGUUCCACAUAUCCCAC .((((..((.((.(((((((((...(((....(((((--((.(............).)))))))....))).))))))))).)).)))))).... ( -26.50) >consensus AGAUGGCUGCGGUACAUGUGUACAUGUGUAUCUGUGU__AUCCACACCCACACUCGCACACACACCUUCGCCUACACAUGUUCCACAUAUCCCAC .((((..((.((.(((((((((..(((((....((((............))))....)))))..........))))))))).)).)))))).... (-17.00 = -18.00 + 1.00)


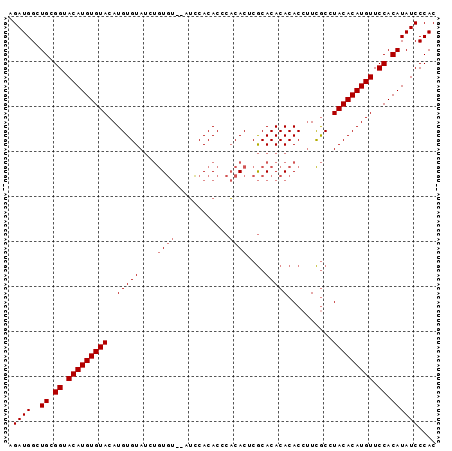
| Location | 6,269,052 – 6,269,155 |
|---|---|
| Length | 103 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.54 |
| Mean single sequence MFE | -42.25 |
| Consensus MFE | -29.81 |
| Energy contribution | -31.25 |
| Covariance contribution | 1.44 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.89 |
| Structure conservation index | 0.71 |
| SVM decision value | 2.77 |
| SVM RNA-class probability | 0.996938 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 6269052 103 - 22224390 -ACGAUGGGCAUAACCCGUCGUGGGGCUGCGGACACGGUCAGAUGGCUGCGGUACAUGU----------------GUACAUGUGUACCCAUUCUCAGACACACACUCACGCACACACACA -..((((((.....))))))(((.(..((((((....(((.((.(..((.(((((((((----------------....)))))))))))..))).)))......)).))))..).))). ( -36.00) >DroSec_CAF1 11224 101 - 1 -ACGAUGGGCAUAACCCGUCGUGGGGCUGCGGACACGGUCAGAUGGCUGCGGUACAUGU----------------GUACAUGUGUAUCUGUGU--AUCCACACCCACACUCGCACACACA -..((((((.....))))))((((((.((.((((((((((....))))..(((((((((----------------....))))))))).))).--.))).))))).)))........... ( -36.80) >DroSim_CAF1 12570 101 - 1 -ACGAUGGGCAUAACCCGUCGUGGGGCUGCGGACACGGUCAGAUGGCUGCGGUACAUGU----------------GUACAUGUGUAUCUGUGU--GUCCACACCCACACUCGCACACACA -..((((((.....))))))((((((.((.((((((((((....))))(((((((((((----------------....))))))).))))))--)))).))))).)))........... ( -42.70) >DroYak_CAF1 8351 107 - 1 GGCGAUGGGCAUAACCCGUCGUGGGGCUGCGGACACGGUCAGAUGGCUGCGGUACAUGUGUACAUGUGUACAUGUGUACAUGUGUAUCUGUGU--AUGCACACCCACAC----------- ...((((((.....))))))(((((..((((.((((((((....))))..(((((((((((((((((....))))))))))))))))).))))--.))))..)))))..----------- ( -53.50) >consensus _ACGAUGGGCAUAACCCGUCGUGGGGCUGCGGACACGGUCAGAUGGCUGCGGUACAUGU________________GUACAUGUGUAUCUGUGU__AUCCACACCCACACUCGCACACACA ...((((((.....))))))((((((((((((.(((.....).)).)))))))((((((..(((((....)))))..))))))...................)))))............. (-29.81 = -31.25 + 1.44)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:32:12 2006