| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 777,130 – 777,237 |
| Length | 107 |
| Max. P | 0.892748 |

| Location | 777,130 – 777,237 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.12 |
| Mean single sequence MFE | -29.30 |
| Consensus MFE | -23.79 |
| Energy contribution | -23.93 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.80 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.97 |
| SVM RNA-class probability | 0.892748 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
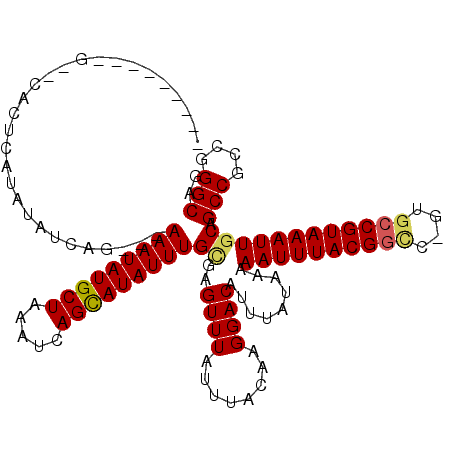
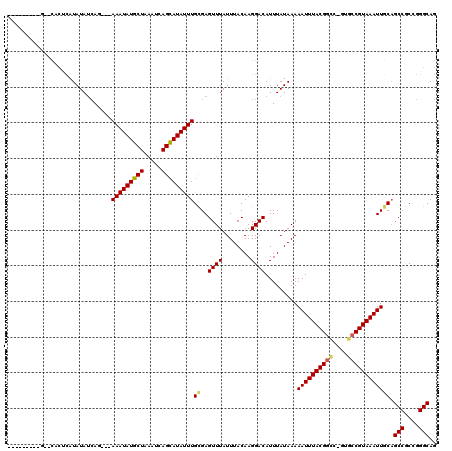
>X_DroMel_CAF1 777130 107 + 22224390 ---------GGACACACAUAUAUCAG---AAAUAUGCUAAAUCAGCAUAUUUGCGAGUUUAUUUACAAGGACAUUUAUAAAAAUUUACGGUC-GUGCCGUAAAUUGCAGCCGCCGGGCAG ---------.................---(((((((((.....)))))))))((..((((........)))).........(((((((((..-...))))))))))).(((....))).. ( -25.90) >DroVir_CAF1 142986 105 + 1 ---------G--CAUUCAUAUAUCAC---AAAUAUGCUAAAUCAGCAUAUUUGCGAGUUUAUUUACAAGGACAUUUAUAAAAAUUUACGGCC-GUGCCGUAAAUUGCAGCCGCCGGGCAG ---------(--(.....((((((.(---(((((((((.....)))))))))).))((((........))))...))))..((((((((((.-..)))))))))))).(((....))).. ( -31.90) >DroGri_CAF1 164418 105 + 1 ---------G--CAUUCAUAUAUCAU---AAAUAUGCUAAAUCAGUAUAUUUGUGAGUUUAUUUACAAGGACAUUUAUAAAAAUUUACGGCC-CUGCCGUAAAUUUCCGCCGCCGGGCAG ---------.--..........((((---(((((((((.....)))))))))))))............(((..........((((((((((.-..)))))))))))))(((....))).. ( -30.20) >DroEre_CAF1 105689 104 + 1 ------------CACUCGUAUAUCAG---AAAUAUGCUAAAUCAGCAUAUUUGCGAGUUUAUUUACAAGGACAUUUAUAAAAAUUUACGGGC-GUGCCGUAAAUUGCAGCCGCCGGGCAG ------------.(((((((......---.((((((((.....))))))))))))))).......................(((((((((..-...)))))))))...(((....))).. ( -27.51) >DroWil_CAF1 134049 120 + 1 UUACUCCCCGUAUUCGUAUAUAGAAGAGAAAAUAUGCUAAAUCAGCAUAUUUGUGAGUUUAUUUACAAGGACAUUUAUAAAAAUUUACGACCCAGGCCGUAAAUUGCAGCCGCCGGGCAG ...((.((((.....(((.(((((.....(((((((((.....))))))))).....))))).)))..((...........((((((((.(....).))))))))....))..)))).)) ( -28.36) >DroMoj_CAF1 168277 105 + 1 ---------G--CAUUCAUAUAUCAC---AAAUAUGCUAAAUCAGCAUAUUUGCGAGUUUAUUUACAAGGACAUUUAUAAAAAUUUACGGCC-GUGCCGUAAAUUGCAGCCGCCGGGCAG ---------(--(.....((((((.(---(((((((((.....)))))))))).))((((........))))...))))..((((((((((.-..)))))))))))).(((....))).. ( -31.90) >consensus _________G__CACUCAUAUAUCAG___AAAUAUGCUAAAUCAGCAUAUUUGCGAGUUUAUUUACAAGGACAUUUAUAAAAAUUUACGGCC_GUGCCGUAAAUUGCAGCCGCCGGGCAG .............................(((((((((.....)))))))))((..((((........)))).........((((((((((....)))))))))))).(((....))).. (-23.79 = -23.93 + 0.14)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:39:18 2006