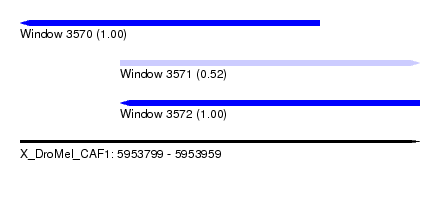
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 5,953,799 – 5,953,959 |
| Length | 160 |
| Max. P | 0.999990 |

| Location | 5,953,799 – 5,953,919 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.17 |
| Mean single sequence MFE | -40.93 |
| Consensus MFE | -37.31 |
| Energy contribution | -36.70 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.91 |
| SVM decision value | 2.61 |
| SVM RNA-class probability | 0.995703 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 5953799 120 - 22224390 AAUUGCGAUGACGCGGCCGAUUGCGUGCGCAAUUGCUGCGUCAGCUCGGUUAUUGCUCCCAUAUUCUUUGUGAUAUUCGUGCUAAUGGCGCAAUUCGUUUUAGUGAACGUCGUCGUGGCU ..(..((((((((((((.((((((....)))))))))))((((((.((..(((..(.............)..)))..)).))...)))).......((((....)))))))))))..).. ( -42.02) >DroVir_CAF1 12159 120 - 1 AACUGCGAUGACGCGGCCGAUUGCGUGCGCAAUUGCUGCGUCAGCUCGUUUAUUGCUCCCAUAUUCUUUGUGAUAUUCGUUCUUAUGGCGCAAUUCGUUUUAGUGAACGUCGUCGUGGCU ..(..((((((((((((.((((((....)))))))))))((..(((((...(((((..(((((.....((.......))....))))).))))).))....)))..)))))))))..).. ( -41.30) >DroPse_CAF1 1498 120 - 1 AAUUGCGAUGACGCGGCCGAUUGUGUACGCAAUUGCUGCGUCAGCUCGGUUAUUGCUCCCAUAUUCUUUGUGAUAUUCGUGCUAAUGGCGCAAUUCGUUUUGGUGAAUGUCGUCGUGGCU (((((((..((((((((.((((((....))))))))))))))(((.((..(((..(.............)..)))..)).))).....))))))).((..((..(.....)..))..)). ( -40.92) >DroGri_CAF1 11551 120 - 1 AACUGCGAUGACGCGGCCGAUUGCGUGCGCAAUUGUUGCGUCAGCUCGGUUAUUGCUCCCAUAUUCUUUGUGAUAUUCGUUCUUAUGGCGCAAUUCGUGUUAGUGAACGUCGUCGUGGCU ..(..(((((((((((((((..((..(((((.....)))))..)))))))...)))...((((.....))))......((((...((((((.....))))))..)))))))))))..).. ( -42.20) >DroWil_CAF1 1431 120 - 1 AACUGCGAUGACGCGGCUGAUUGCGUACGCAAUUGCUGCGUCAGCUCGGUCAUUGCUCCCAUAUUCUUUGUGAUAUUCGUGCUAAUGGCGCAAUUCGUUUUAGUGAACGUCGUCGUGGCU ..(..((((((((((((.((((((....)))))))))))((((((.(((..((..(.............)..))..))).))...)))).......((((....)))))))))))..).. ( -41.72) >DroMoj_CAF1 13272 120 - 1 AACUGCGAUGACGCCGCCGAUUGCGUGCGCAAUUGCUGCGUCAGCUCAUUUAUUGCUCCCAUAUUCUUUGUGAUAUUCGUUCUUAUGGCGCAAUUCGUUUUAGUGAACGUCGUCGUGGCU ..(..((((((((.((((((((((....))))))).(((((((((.........))...((((.....)))).............)))))))..........)))..))))))))..).. ( -37.40) >consensus AACUGCGAUGACGCGGCCGAUUGCGUGCGCAAUUGCUGCGUCAGCUCGGUUAUUGCUCCCAUAUUCUUUGUGAUAUUCGUGCUAAUGGCGCAAUUCGUUUUAGUGAACGUCGUCGUGGCU ..(..((((((((((((.((((((....)))))))))))((..(((.....(((((.((((((.....))))......((....)))).))))).......)))..)))))))))..).. (-37.31 = -36.70 + -0.61)

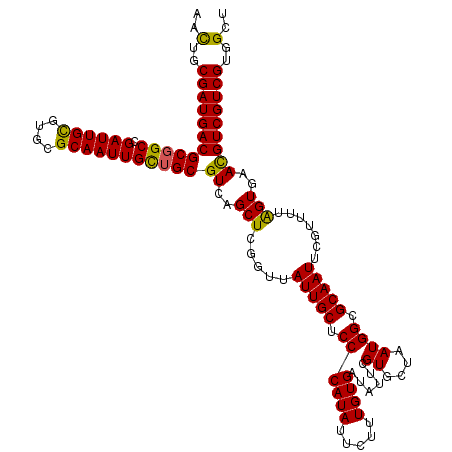
| Location | 5,953,839 – 5,953,959 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.78 |
| Mean single sequence MFE | -33.30 |
| Consensus MFE | -31.42 |
| Energy contribution | -31.33 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.08 |
| Mean z-score | -0.81 |
| Structure conservation index | 0.94 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.515396 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 5953839 120 + 22224390 ACGAAUAUCACAAAGAAUAUGGGAGCAAUAACCGAGCUGACGCAGCAAUUGCGCACGCAAUCGGCCGCGUCAUCGCAAUUAUCCCUCAGCGUAUCCUUCAUGAUGCCGUUCCAAUUGUCG (((..(((((..(((.(((((.(((..((((.(((..((((((.((.(((((....)))))..)).)))))))))...))))..)))..))))).)))..))))).)))........... ( -32.90) >DroVir_CAF1 12199 120 + 1 ACGAAUAUCACAAAGAAUAUGGGAGCAAUAAACGAGCUGACGCAGCAAUUGCGCACGCAAUCGGCCGCGUCAUCGCAGUUAUCCCUCAGCGUGUCCUUCAUGAUGCCGUUCCAGUUGUCG (((..(((((..(((.(((((.(((..((((.(((..((((((.((.(((((....)))))..)).)))))))))...))))..)))..))))).)))..))))).)))........... ( -34.00) >DroPse_CAF1 1538 120 + 1 ACGAAUAUCACAAAGAAUAUGGGAGCAAUAACCGAGCUGACGCAGCAAUUGCGUACACAAUCGGCCGCGUCAUCGCAAUUAUCCCUCAGCGUGUCCUUCAUGAUGCCGUUCCAAUUAUCG (((..(((((..(((.(((((.(((..((((((((..((((((((...)))))).))...))))..(((....)))..))))..)))..))))).)))..))))).)))........... ( -29.30) >DroGri_CAF1 11591 120 + 1 ACGAAUAUCACAAAGAAUAUGGGAGCAAUAACCGAGCUGACGCAACAAUUGCGCACGCAAUCGGCCGCGUCAUCGCAGUUAUCCCUCAGCGUGUCCUUCAUGAUGCCGUUCCAGUUGUCG (((..(((((..(((.(((((.(((..(((((.(.(.((((((..(.(((((....))))).)...)))))).).).)))))..)))..))))).)))..))))).)))........... ( -33.20) >DroWil_CAF1 1471 120 + 1 ACGAAUAUCACAAAGAAUAUGGGAGCAAUGACCGAGCUGACGCAGCAAUUGCGUACGCAAUCAGCCGCGUCAUCGCAGUUAUCCCUCAGCGUGUCCUUCAUGAUGCCGUUCCAAUUAUCG (((..(((((..(((.(((((.(((..(((((.(.(.((((((.((.(((((....)))))..)).)))))).).).)))))..)))..))))).)))..))))).)))........... ( -35.70) >DroMoj_CAF1 13312 120 + 1 ACGAAUAUCACAAAGAAUAUGGGAGCAAUAAAUGAGCUGACGCAGCAAUUGCGCACGCAAUCGGCGGCGUCAUCGCAGUUAUCCCUCAGCGUGUCCUUCAUGAUGCCGUUCCAGUUGUCG (((..(((((..(((.(((((.(((..((((.((.(.((((((.((.(((((....)))))..)).)))))).).)).))))..)))..))))).)))..))))).)))........... ( -34.70) >consensus ACGAAUAUCACAAAGAAUAUGGGAGCAAUAACCGAGCUGACGCAGCAAUUGCGCACGCAAUCGGCCGCGUCAUCGCAGUUAUCCCUCAGCGUGUCCUUCAUGAUGCCGUUCCAAUUGUCG (((..(((((..(((.(((((.(((..((((.(((..((((((.((.(((((....)))))..)).)))))))))...))))..)))..))))).)))..))))).)))........... (-31.42 = -31.33 + -0.08)


| Location | 5,953,839 – 5,953,959 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.78 |
| Mean single sequence MFE | -45.18 |
| Consensus MFE | -45.29 |
| Energy contribution | -45.43 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.72 |
| Structure conservation index | 1.00 |
| SVM decision value | 5.60 |
| SVM RNA-class probability | 0.999990 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 5953839 120 - 22224390 CGACAAUUGGAACGGCAUCAUGAAGGAUACGCUGAGGGAUAAUUGCGAUGACGCGGCCGAUUGCGUGCGCAAUUGCUGCGUCAGCUCGGUUAUUGCUCCCAUAUUCUUUGUGAUAUUCGU ...........((((.((((..(((((((.(..(((.((((((((.(.(((((((((.((((((....))))))))))))))).).)))))))).))).).)))))))..))))..)))) ( -49.70) >DroVir_CAF1 12199 120 - 1 CGACAACUGGAACGGCAUCAUGAAGGACACGCUGAGGGAUAACUGCGAUGACGCGGCCGAUUGCGUGCGCAAUUGCUGCGUCAGCUCGUUUAUUGCUCCCAUAUUCUUUGUGAUAUUCGU ...........((((.((((..(((((......(((.(((((..(((((((((((((.((((((....)))))))))))))))..))))))))).))).....)))))..))))..)))) ( -44.30) >DroPse_CAF1 1538 120 - 1 CGAUAAUUGGAACGGCAUCAUGAAGGACACGCUGAGGGAUAAUUGCGAUGACGCGGCCGAUUGUGUACGCAAUUGCUGCGUCAGCUCGGUUAUUGCUCCCAUAUUCUUUGUGAUAUUCGU ...........((((.((((..(((((......(((.((((((((.(.(((((((((.((((((....))))))))))))))).).)))))))).))).....)))))..))))..)))) ( -45.10) >DroGri_CAF1 11591 120 - 1 CGACAACUGGAACGGCAUCAUGAAGGACACGCUGAGGGAUAACUGCGAUGACGCGGCCGAUUGCGUGCGCAAUUGUUGCGUCAGCUCGGUUAUUGCUCCCAUAUUCUUUGUGAUAUUCGU ...........((((.((((..(((((......(((.((((((((.(.(((((((((.((((((....))))))))))))))).).)))))))).))).....)))))..))))..)))) ( -46.70) >DroWil_CAF1 1471 120 - 1 CGAUAAUUGGAACGGCAUCAUGAAGGACACGCUGAGGGAUAACUGCGAUGACGCGGCUGAUUGCGUACGCAAUUGCUGCGUCAGCUCGGUCAUUGCUCCCAUAUUCUUUGUGAUAUUCGU ...........((((.((((..(((((......(((.(((.((((.(.(((((((((.((((((....))))))))))))))).).)))).))).))).....)))))..))))..)))) ( -45.30) >DroMoj_CAF1 13312 120 - 1 CGACAACUGGAACGGCAUCAUGAAGGACACGCUGAGGGAUAACUGCGAUGACGCCGCCGAUUGCGUGCGCAAUUGCUGCGUCAGCUCAUUUAUUGCUCCCAUAUUCUUUGUGAUAUUCGU ...........((((.((((..(((((......(((.(((((.((.(.((((((.((.((((((....)))))))).)))))).).)).))))).))).....)))))..))))..)))) ( -40.00) >consensus CGACAACUGGAACGGCAUCAUGAAGGACACGCUGAGGGAUAACUGCGAUGACGCGGCCGAUUGCGUGCGCAAUUGCUGCGUCAGCUCGGUUAUUGCUCCCAUAUUCUUUGUGAUAUUCGU ...........((((.((((..(((((......(((.((((((((.(.(((((((((.((((((....))))))))))))))).).)))))))).))).....)))))..))))..)))) (-45.29 = -45.43 + 0.14)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:28:38 2006