
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 5,933,635 – 5,933,821 |
| Length | 186 |
| Max. P | 0.956170 |

| Location | 5,933,635 – 5,933,746 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 74.79 |
| Mean single sequence MFE | -41.48 |
| Consensus MFE | -17.22 |
| Energy contribution | -17.28 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.32 |
| Mean z-score | -1.91 |
| Structure conservation index | 0.42 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.512546 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 5933635 111 + 22224390 UGUGCAUUCAUCGGAAGAAUCUCGUUUGCAGCACAGGCAGCCAGGUGUAUACAGUUCUUGUAACUGACCAUCUGGUUGGUUAACUGCACCUGGUUGGCGGCUACGUUGGCG (((((.....((....)).....(....).)))))(.((((((((((....((((.....((((..(((....)))..)))))))))))))))))).).(((.....))). ( -42.40) >DroPse_CAF1 23403 110 + 1 UGGGCAUUCAUCGGCAGUAUCUCGUUGGCCGCACACGCGGCCAAAUGUAUGCAGUUCUUGUAGCUGACCAACUGCGUCGGCAGCUGUACAGAGGA-GCCGCCGGAUCCAGA ((((...((..((((.((.((((.((((((((....)))))))).....(((((((......(((((((....).)))))))))))))..)))).-)).)))))))))).. ( -44.00) >DroEre_CAF1 20915 111 + 1 UGCGCAUUCAUGGGCAGAAUUUCAUUGGCAGCACAGGCGGCCAGGUGUAUACAGUUCUUGUAACUGACCAUUUGGUUGGGCAACUGCACCUGGUUGGCGGCUACAUUGCCG (((.((....)).)))..........((((((...(.(((((((((((((((((...))))).(..(((....)))..).....)))))))))))).).)).....)))). ( -41.50) >DroYak_CAF1 23745 111 + 1 UGCGCAUUCAUGGGCAGAAUCUCAUUGGCAGCACAGGCGGCCAGGUGUAUACAGUUCUGGUAACUGACCAUCUGGUUGGGCAACUGCACCUGGUUGGAGGCUACAUUGACG (((.((....)).))).....(((.((..(((.....(((((((((((...(((...((((.....)))).)))...((....)))))))))))))...))).)).))).. ( -37.20) >DroMoj_CAF1 36137 89 + 1 UGGGCAUUCAUCGGCAGAAUCUCAUUGGCUGCACAGGCGGCCAAAUGGAUGCAGUUCUUGUAGCUGACCAGCUGGGAGGCCGACUGCGU---------------------- ...(((....(((((.(.((((..((((((((....))))))))..)))).)....(((.(((((....))))).)))))))).)))..---------------------- ( -36.20) >DroPer_CAF1 22872 110 + 1 UGGGCAUUCAUCGGCAGUAUCUCGUUGGCCGCACACGCGGCCAAAUGUAUGCAGUUCUUGUAGCUGACCAGCUGCGUCGGCAGCUGUACAGAGGA-GCCGCCGGAUCCAGA ((((...((..((((.((.((((.((((((((....)))))))).((((..(((((.....)))))..(((((((....))))))))))))))).-)).)))))))))).. ( -47.60) >consensus UGGGCAUUCAUCGGCAGAAUCUCAUUGGCAGCACAGGCGGCCAAAUGUAUACAGUUCUUGUAACUGACCAUCUGGGUGGGCAACUGCACCUGGUU_GCGGCUACAUUGACG ..(((........((((.......(((((.((....)).))))).(((...(((((.....))))).(((......)))))).)))).........)))............ (-17.22 = -17.28 + 0.06)
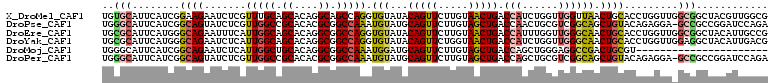


| Location | 5,933,675 – 5,933,782 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 80.25 |
| Mean single sequence MFE | -43.30 |
| Consensus MFE | -29.62 |
| Energy contribution | -29.37 |
| Covariance contribution | -0.26 |
| Combinations/Pair | 1.36 |
| Mean z-score | -2.86 |
| Structure conservation index | 0.68 |
| SVM decision value | 1.46 |
| SVM RNA-class probability | 0.956170 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 5933675 107 + 22224390 CCAGGUGUAUACAGUUCUUGUAACUGACCAUCUGGUUGGUUAACUGCACCUGGUUGGCGGCUACGUUGGCGGCACCA-CCAGAGCCGCCCACUAACUGUUGGACACCA ...(((((..(((((.....((((..(((....)))..))))........((((.(((((((...((((.(....).-))))))))))).)))))))))...))))). ( -45.80) >DroSec_CAF1 21359 107 + 1 CCAGGUGUAUACAGUUCUUGUAACUGACCAUCUGGUUGGUUAACUGCACCUGGUUGGCGGCUACAUUGGCGGCACCA-CCAGAGCCGCCCACUAACUGUUGGACACCA ...(((((..(((((.....((((..(((....)))..))))........((((.(((((((...((((.(....).-))))))))))).)))))))))...))))). ( -45.80) >DroSim_CAF1 27484 107 + 1 CCAGGUGUAUACAGUUCUUGUAACUGACCAUCUGGUUGGUUAACUGCACCUGGUUGGCGGCUACAUUGGCGGCACCA-CCAGAGCCGCCCACUAACUGUUGGACACCA ...(((((..(((((.....((((..(((....)))..))))........((((.(((((((...((((.(....).-))))))))))).)))))))))...))))). ( -45.80) >DroEre_CAF1 20955 107 + 1 CCAGGUGUAUACAGUUCUUGUAACUGACCAUUUGGUUGGGCAACUGCACCUGGUUGGCGGCUACAUUGCCGGCACCA-CCUGAGCCGCCCACCAUCUGCUGGACUCCG ((((.......(((((.....)))))..((..((((.((((..((.((..((((.(.((((......)))).)))))-..))))..))))))))..))))))...... ( -38.90) >DroYak_CAF1 23785 107 + 1 CCAGGUGUAUACAGUUCUGGUAACUGACCAUCUGGUUGGGCAACUGCACCUGGUUGGAGGCUACAUUGACGCCACCA-CCUGAGCCGCCCACCAGCUGCUGGACUCCG ...((.((...((((.(((((....((((....))))((((....((.(..(((.((.(((.........))).)))-)).).)).)))))))))..)))).)).)). ( -38.60) >DroPer_CAF1 22912 104 + 1 CCAAAUGUAUGCAGUUCUUGUAGCUGACCAGCUGCGUCGGCAGCUGUACAGAGGA-GCCGCCGGAUCCAGAGCCCGGUAUAGAGCC---GGGCUGCAGAUGGGCCAGG ((........((.((((((....(((..(((((((....)))))))..)))))))-)).)).((.((((.((((((((.....)))---))))).....)))))).)) ( -44.90) >consensus CCAGGUGUAUACAGUUCUUGUAACUGACCAUCUGGUUGGGCAACUGCACCUGGUUGGCGGCUACAUUGGCGGCACCA_CCAGAGCCGCCCACCAACUGUUGGACACCA (((((((....(((((.....)))))..))))))).((((...(.(((..((((.(((((((....((.......)).....))))))).))))..))).)...)))) (-29.62 = -29.37 + -0.26)


| Location | 5,933,675 – 5,933,782 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 80.25 |
| Mean single sequence MFE | -41.55 |
| Consensus MFE | -24.81 |
| Energy contribution | -25.55 |
| Covariance contribution | 0.74 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.11 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.62 |
| SVM RNA-class probability | 0.802316 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 5933675 107 - 22224390 UGGUGUCCAACAGUUAGUGGGCGGCUCUGG-UGGUGCCGCCAACGUAGCCGCCAACCAGGUGCAGUUAACCAACCAGAUGGUCAGUUACAAGAACUGUAUACACCUGG .(((((...((((((..(((((((((((((-((....)))))..).))))))).............((((..(((....)))..))))))..))))))..)))))... ( -41.90) >DroSec_CAF1 21359 107 - 1 UGGUGUCCAACAGUUAGUGGGCGGCUCUGG-UGGUGCCGCCAAUGUAGCCGCCAACCAGGUGCAGUUAACCAACCAGAUGGUCAGUUACAAGAACUGUAUACACCUGG .(((((...((((((..(((((((((((((-((....)))))..).))))))).............((((..(((....)))..))))))..))))))..)))))... ( -41.90) >DroSim_CAF1 27484 107 - 1 UGGUGUCCAACAGUUAGUGGGCGGCUCUGG-UGGUGCCGCCAAUGUAGCCGCCAACCAGGUGCAGUUAACCAACCAGAUGGUCAGUUACAAGAACUGUAUACACCUGG .(((((...((((((..(((((((((((((-((....)))))..).))))))).............((((..(((....)))..))))))..))))))..)))))... ( -41.90) >DroEre_CAF1 20955 107 - 1 CGGAGUCCAGCAGAUGGUGGGCGGCUCAGG-UGGUGCCGGCAAUGUAGCCGCCAACCAGGUGCAGUUGCCCAACCAAAUGGUCAGUUACAAGAACUGUAUACACCUGG ......((((((..((((((((((((((..-((((..((((......))))...))))..)).)))))))).))))..))..(((((.....))))).......)))) ( -41.40) >DroYak_CAF1 23785 107 - 1 CGGAGUCCAGCAGCUGGUGGGCGGCUCAGG-UGGUGGCGUCAAUGUAGCCUCCAACCAGGUGCAGUUGCCCAACCAGAUGGUCAGUUACCAGAACUGUAUACACCUGG .((.((...(((((((((((((((((((((-(((.(((.........))).)).)))...)).)))))))).))))).((((.....))))...))))..)).))... ( -41.20) >DroPer_CAF1 22912 104 - 1 CCUGGCCCAUCUGCAGCCC---GGCUCUAUACCGGGCUCUGGAUCCGGCGGC-UCCUCUGUACAGCUGCCGACGCAGCUGGUCAGCUACAAGAACUGCAUACAUUUGG .((((((...(((((((((---((.......))))))).......(((((((-(.........))))))))..))))..))))))....................... ( -41.00) >consensus CGGUGUCCAACAGUUAGUGGGCGGCUCUGG_UGGUGCCGCCAAUGUAGCCGCCAACCAGGUGCAGUUAACCAACCAGAUGGUCAGUUACAAGAACUGUAUACACCUGG ...................(((((((.....(((.....)))....)))))))..(((((((..........(((....)))(((((.....)))))....))))))) (-24.81 = -25.55 + 0.74)

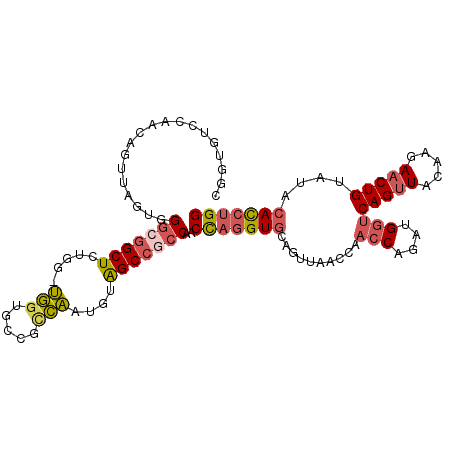

| Location | 5,933,715 – 5,933,821 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 80.12 |
| Mean single sequence MFE | -47.45 |
| Consensus MFE | -28.43 |
| Energy contribution | -28.43 |
| Covariance contribution | 0.01 |
| Combinations/Pair | 1.44 |
| Mean z-score | -1.70 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.15 |
| SVM RNA-class probability | 0.608650 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 5933715 106 - 22224390 AGAACUAGGGAAGGCGCUGGCCGCCGCCCAGCAGAGGGGUGGUGUCCAACAGUUAGUGGGCGGCUCUGGUGGUGCCGCCAACGUAGCCGC---------CAACCAGGUGCAGUUA .........((..(((((((.((((((((.......)))))))).))........((.(((((((((((((....)))))..).))))))---------).))..)))))..)). ( -47.10) >DroSec_CAF1 21399 106 - 1 AGAACUAGAGAAGGCGCUGGCCGCCGCCCAGCAGAGGGGUGGUGUCCAACAGUUAGUGGGCGGCUCUGGUGGUGCCGCCAAUGUAGCCGC---------CAACCAGGUGCAGUUA .........((..(((((((.((((((((.......)))))))).))........((.(((((((((((((....)))))..).))))))---------).))..)))))..)). ( -47.00) >DroSim_CAF1 27524 106 - 1 AGAACUAGAGAAGGCGCUGGCCGCCGCCCAGCAGAGGGGUGGUGUCCAACAGUUAGUGGGCGGCUCUGGUGGUGCCGCCAAUGUAGCCGC---------CAACCAGGUGCAGUUA .........((..(((((((.((((((((.......)))))))).))........((.(((((((((((((....)))))..).))))))---------).))..)))))..)). ( -47.00) >DroEre_CAF1 20995 106 - 1 AGAACUGGAGAAGGCGCUGGCCGCCGCCCAGCAGAGGGGCGGAGUCCAGCAGAUGGUGGGCGGCUCAGGUGGUGCCGGCAAUGUAGCCGC---------CAACCAGGUGCAGUUG ..(((((.(......(((((.(.((((((.......)))))).).)))))...((((.((((((((((.((....)).)..)).))))))---------).))))..).))))). ( -50.70) >DroYak_CAF1 23825 106 - 1 AGAACUGAAAAAGGCGCAGGCCGCCGCCCAGCAGAGGGGCGGAGUCCAGCAGCUGGUGGGCGGCUCAGGUGGUGGCGUCAAUGUAGCCUC---------CAACCAGGUGCAGUUG ..(((((.....(((....)))((((((((.(((..((((...)))).....))).))))))))...(((((.(((.........))).)---------).))).....))))). ( -48.60) >DroAna_CAF1 28547 109 - 1 CGAGCUGGAGAAGCAACGGGCCACCCUCCAGAAGAGGGGCGGCGUACCGCAGCUGGCA---GGCACUGGUGGUGCUCCGGGUACGGCCGGUGGACAGAACCAACAGAUGCAG--- ...(((.....)))...(((((((((((.....)))))((((....))))...)))).---..(((((((.((((.....)))).))))))).......))...........--- ( -44.30) >consensus AGAACUAGAGAAGGCGCUGGCCGCCGCCCAGCAGAGGGGCGGUGUCCAACAGUUAGUGGGCGGCUCUGGUGGUGCCGCCAAUGUAGCCGC_________CAACCAGGUGCAGUUA .............(((((((.(.((((((.......)))))).).)))...((((((.((((((.(.....).)))))).)).))))...................))))..... (-28.43 = -28.43 + 0.01)
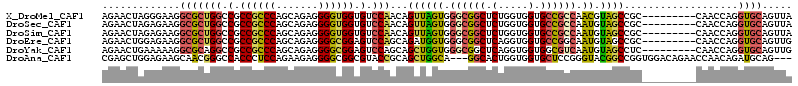

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:28:24 2006