
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 5,726,593 – 5,726,748 |
| Length | 155 |
| Max. P | 0.999798 |

| Location | 5,726,593 – 5,726,708 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.33 |
| Mean single sequence MFE | -40.78 |
| Consensus MFE | -35.36 |
| Energy contribution | -35.80 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.56 |
| SVM RNA-class probability | 0.963606 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 5726593 115 + 22224390 CAUUCAAUUAACAUUCCC-----AUGUCCACAGUUUGGAGGGCGCCAUCAAGAAGAUGGUGCACAAGGCCUUUUGGGAUCUGCUGCGUGUGCAGCUGGACCGCCAGCCGCCAUCGUAUGA ...(((........((((-----((.((((.....)))).)((((((((.....))))))))...........))))).....((((((.((.(((((....))))).)))).))))))) ( -40.20) >DroSec_CAF1 306 120 + 1 CAUUCAAUUAACAUUCCUUUUGAAUGUCCACAGUUUGGAGGGCGCCAUCAAGAAGAUGGUACACAAGGCCUUUUGGGAUCUGCUGCGUGUGCAGCUGGAACGCCAGCCGCCAUCGUAUGA (((((((............)))))))....(((.(..((((((((((((.....)))))).(....)))))))..)...))).((((((.((.(((((....))))).)))).))))... ( -38.80) >DroSim_CAF1 1082 120 + 1 CAUUCAAUUAACAUUCCUUUUGAAUGUCCACAGUUUGGAGGGCGCCAUCAAGAAGAUGGUGCACAAGGCCUUUUGGGAUCUGCUGCGUGUGCAGCUGGAACGCCAGCCGCCAUCGUAUGA (((((((............)))))))(((.(((...((..(((((((((.....)))))))).)....))..)))))).....((((((.((.(((((....))))).)))).))))... ( -40.90) >DroEre_CAF1 312 120 + 1 CAUUCAAUUAACAUUCCUUUUGUAUGUCCACAGUUUGGAGGGCGCCAUCAAGAAGAUGGUGCACAAGGCCUUCUGGGAUCUGCUGCGUCAGCAGCUAGACCGCCAGCCGCCAUCGUAUGA (((.(..............((((.(.((((.....)))).)((((((((.....))))))))))))(((...(((((.((((((((....))))).))).).))))..)))...).))). ( -42.00) >DroYak_CAF1 1155 120 + 1 CAUUCCAUUAAAAUACCCUUUUAAUGUACCCAGUUUGGAGGGCGCCAUCAAGAAGAUGGUGCACAAGGCGUUCUGGGAUCUGUUGCGUCAGCAGCUGGACCGCCAGCCGCCAUCGUAUGA .....((((((((.....))))))))..(((((..((...(((((((((.....)))))))).)....))..)))))......((((...((.(((((....))))).))...))))... ( -42.00) >consensus CAUUCAAUUAACAUUCCUUUUGAAUGUCCACAGUUUGGAGGGCGCCAUCAAGAAGAUGGUGCACAAGGCCUUUUGGGAUCUGCUGCGUGUGCAGCUGGACCGCCAGCCGCCAUCGUAUGA (((.(..........(((((....((....))....)))))((((((((.....))))))))....(((...(((((.((((((((....))))).))).).))))..)))...).))). (-35.36 = -35.80 + 0.44)


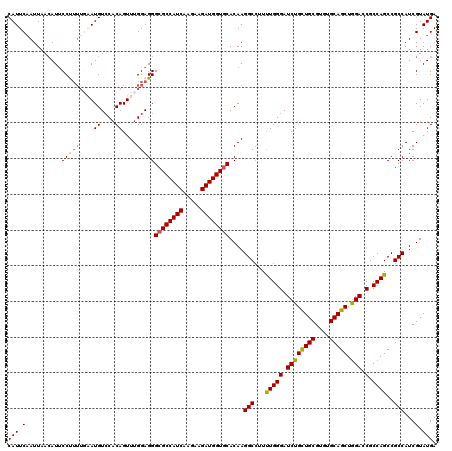
| Location | 5,726,628 – 5,726,748 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.33 |
| Mean single sequence MFE | -52.34 |
| Consensus MFE | -50.40 |
| Energy contribution | -50.28 |
| Covariance contribution | -0.12 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.96 |
| SVM decision value | 4.10 |
| SVM RNA-class probability | 0.999798 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
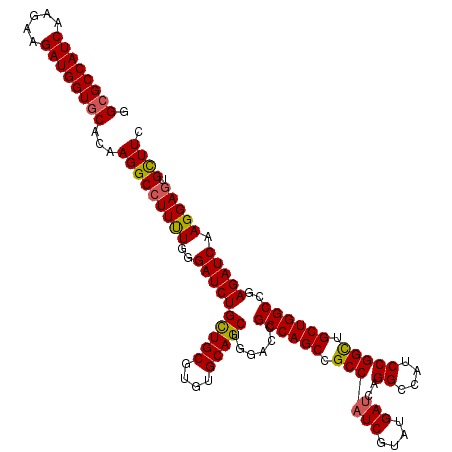
>X_DroMel_CAF1 5726628 120 + 22224390 GGCGCCAUCAAGAAGAUGGUGCACAAGGCCUUUUGGGAUCUGCUGCGUGUGCAGCUGGACCGCCAGCCGCCAUCGUAUGAUCAGGCCAUCCGGCUGCUGGCUGAGAUCAAGGAGUGCUUC .((((((((.....))))))))...(((((((((..((((((((((....)))))......((((((.((((((....)))..((....))))).))))))..))))).))))).)))). ( -52.80) >DroSec_CAF1 346 120 + 1 GGCGCCAUCAAGAAGAUGGUACACAAGGCCUUUUGGGAUCUGCUGCGUGUGCAGCUGGAACGCCAGCCGCCAUCGUAUGAUCAGGCCAUCCGGUUGCUGGCCGAGAUCAAGGAGUGCUUC .(..(((((.....)))))..)...(((((((((..((((((((((....)))))......((((((.((((((....)))..((....))))).))))))..))))).))))).)))). ( -45.40) >DroSim_CAF1 1122 120 + 1 GGCGCCAUCAAGAAGAUGGUGCACAAGGCCUUUUGGGAUCUGCUGCGUGUGCAGCUGGAACGCCAGCCGCCAUCGUAUGAUCAGGCCAUCCGGCUGCUGGCCGAGAUCAAGGAGUGCUUC .((((((((.....))))))))...(((((((((..((((((((((....)))))......((((((.((((((....)))..((....))))).))))))..))))).))))).)))). ( -52.40) >DroEre_CAF1 352 120 + 1 GGCGCCAUCAAGAAGAUGGUGCACAAGGCCUUCUGGGAUCUGCUGCGUCAGCAGCUAGACCGCCAGCCGCCAUCGUAUGAUCAGGCCAUCCGGCUGCUGGCCGAGAUCAAGGAGUGCUUC .((((((((.....))))))))...(((((((((..((((((((((....)))))....(.((((((.((((((....)))..((....))))).)))))).)))))).))))).)))). ( -56.10) >DroYak_CAF1 1195 120 + 1 GGCGCCAUCAAGAAGAUGGUGCACAAGGCGUUCUGGGAUCUGUUGCGUCAGCAGCUGGACCGCCAGCCGCCAUCGUAUGACCAGGCCAUCCGGCUGCUGGCCGAGAUCAAGGAGUGUUUC .((((((((.....))))))))...(((((((((..(((((..((.(((((((((((((..(((...................)))..))))))))))))))))))))..))))))))). ( -55.01) >consensus GGCGCCAUCAAGAAGAUGGUGCACAAGGCCUUUUGGGAUCUGCUGCGUGUGCAGCUGGACCGCCAGCCGCCAUCGUAUGAUCAGGCCAUCCGGCUGCUGGCCGAGAUCAAGGAGUGCUUC .((((((((.....))))))))...(((((((((..((((((((((....)))))......((((((.((((((....)))..((....))))).))))))..))))).))))).)))). (-50.40 = -50.28 + -0.12)

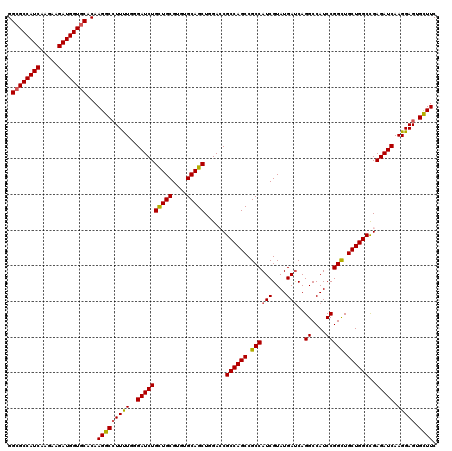
| Location | 5,726,628 – 5,726,748 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.33 |
| Mean single sequence MFE | -47.40 |
| Consensus MFE | -43.16 |
| Energy contribution | -43.44 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.78 |
| Structure conservation index | 0.91 |
| SVM decision value | 2.17 |
| SVM RNA-class probability | 0.989524 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 5726628 120 - 22224390 GAAGCACUCCUUGAUCUCAGCCAGCAGCCGGAUGGCCUGAUCAUACGAUGGCGGCUGGCGGUCCAGCUGCACACGCAGCAGAUCCCAAAAGGCCUUGUGCACCAUCUUCUUGAUGGCGCC ...((((.(((((((((..((((((.((((.((((.....))))....)))).))))))......(((((....))))))))))....))))....)))).(((((.....))))).... ( -48.10) >DroSec_CAF1 346 120 - 1 GAAGCACUCCUUGAUCUCGGCCAGCAACCGGAUGGCCUGAUCAUACGAUGGCGGCUGGCGUUCCAGCUGCACACGCAGCAGAUCCCAAAAGGCCUUGUGUACCAUCUUCUUGAUGGCGCC ...((((.(((((((((..((((((..(((.((((.....))))....)))..))))))......(((((....))))))))))....))))....)))).(((((.....))))).... ( -43.30) >DroSim_CAF1 1122 120 - 1 GAAGCACUCCUUGAUCUCGGCCAGCAGCCGGAUGGCCUGAUCAUACGAUGGCGGCUGGCGUUCCAGCUGCACACGCAGCAGAUCCCAAAAGGCCUUGUGCACCAUCUUCUUGAUGGCGCC ...((((.(((((((((..((((((.((((.((((.....))))....)))).))))))......(((((....))))))))))....))))....)))).(((((.....))))).... ( -47.90) >DroEre_CAF1 352 120 - 1 GAAGCACUCCUUGAUCUCGGCCAGCAGCCGGAUGGCCUGAUCAUACGAUGGCGGCUGGCGGUCUAGCUGCUGACGCAGCAGAUCCCAGAAGGCCUUGUGCACCAUCUUCUUGAUGGCGCC ...((((....(((((..(((((.........))))).)))))......(((..((((.(((((.(((((....))))))))))))))...)))..)))).(((((.....))))).... ( -52.00) >DroYak_CAF1 1195 120 - 1 GAAACACUCCUUGAUCUCGGCCAGCAGCCGGAUGGCCUGGUCAUACGAUGGCGGCUGGCGGUCCAGCUGCUGACGCAACAGAUCCCAGAACGCCUUGUGCACCAUCUUCUUGAUGGCGCC ............(((((..(((((((((.((((.((((.(((((...))))).)..))).)))).)))))))..))...)))))..............((.(((((.....))))).)). ( -45.70) >consensus GAAGCACUCCUUGAUCUCGGCCAGCAGCCGGAUGGCCUGAUCAUACGAUGGCGGCUGGCGGUCCAGCUGCACACGCAGCAGAUCCCAAAAGGCCUUGUGCACCAUCUUCUUGAUGGCGCC ...(((..(((((((((..((((((.((((.((((.....))))....)))).))))))......(((((....))))))))))....))))...)))((.(((((.....))))).)). (-43.16 = -43.44 + 0.28)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:26:30 2006